Trong bài đăng này, chúng tôi trình bày cách tinh chỉnh hiệu quả mô hình ngôn ngữ protein hiện đại (pLM) để dự đoán nội địa hóa protein dưới tế bào bằng cách sử dụng Amazon SageMaker.
Protein là cỗ máy phân tử của cơ thể, chịu trách nhiệm cho mọi thứ từ việc di chuyển cơ bắp đến phản ứng với nhiễm trùng. Mặc dù có sự đa dạng như vậy nhưng tất cả các protein đều được tạo thành từ các chuỗi phân tử lặp đi lặp lại gọi là axit amin. Bộ gen của con người mã hóa 20 axit amin tiêu chuẩn, mỗi loại có cấu trúc hóa học hơi khác nhau. Chúng có thể được biểu thị bằng các chữ cái trong bảng chữ cái, sau đó cho phép chúng ta phân tích và khám phá protein dưới dạng chuỗi văn bản. Số lượng lớn các trình tự và cấu trúc protein có thể mang lại cho protein khả năng sử dụng đa dạng.
Protein cũng đóng một vai trò quan trọng trong việc phát triển thuốc, vừa là mục tiêu tiềm năng vừa là phương pháp điều trị. Như được trình bày trong bảng sau, nhiều loại thuốc bán chạy nhất vào năm 2022 là protein (đặc biệt là kháng thể) hoặc các phân tử khác như mRNA được chuyển hóa thành protein trong cơ thể. Vì điều này, nhiều nhà nghiên cứu khoa học đời sống cần trả lời các câu hỏi về protein nhanh hơn, rẻ hơn và chính xác hơn.
| Họ tên | nhà chế tạo | Doanh số toàn cầu năm 2022 ($ tỷ USD) | Chỉ định |
| công ty | Pfizer / BioNTech | $40.8 | Covid-19 |
| Spikevax | Hiện đại | $21.8 | Covid-19 |
| Humira | AbbVie | $21.6 | Viêm khớp, bệnh Crohn và những bệnh khác |
| keytruda | Merck | $21.0 | Ung thư khác nhau |
Nguồn dữ liệu: Urquhart, L. Các công ty và thuốc hàng đầu theo doanh số bán hàng năm 2022. Nature Reviews Khám phá thuốc 22, 260–260 (2023).
Bởi vì chúng ta có thể biểu diễn protein dưới dạng chuỗi ký tự nên chúng ta có thể phân tích chúng bằng các kỹ thuật ban đầu được phát triển cho ngôn ngữ viết. Điều này bao gồm các mô hình ngôn ngữ lớn (LLM) được đào tạo trước trên các bộ dữ liệu khổng lồ, sau đó có thể được điều chỉnh cho phù hợp với các tác vụ cụ thể, như tóm tắt văn bản hoặc chatbot. Tương tự, pLM được đào tạo trước trên cơ sở dữ liệu chuỗi protein lớn bằng cách sử dụng phương pháp học tự giám sát, không gắn nhãn. Chúng ta có thể điều chỉnh chúng để dự đoán những thứ như cấu trúc 3D của protein hoặc cách nó có thể tương tác với các phân tử khác. Các nhà nghiên cứu thậm chí còn sử dụng pLM để thiết kế các protein mới ngay từ đầu. Những công cụ này không thay thế chuyên môn khoa học của con người, nhưng chúng có tiềm năng tăng tốc độ phát triển tiền lâm sàng và thiết kế thử nghiệm.
Một thách thức với những mô hình này là kích thước của chúng. Cả LLM và pLM đều tăng trưởng theo cấp độ lớn trong vài năm qua, như được minh họa trong hình dưới đây. Điều này có nghĩa là có thể mất nhiều thời gian để huấn luyện chúng đạt đủ độ chính xác. Điều đó cũng có nghĩa là bạn cần sử dụng phần cứng, đặc biệt là GPU, có dung lượng bộ nhớ lớn để lưu trữ các thông số của mô hình.
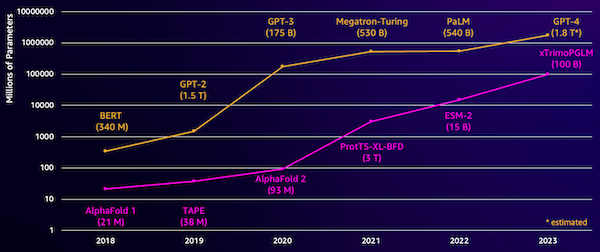
Thời gian đào tạo dài, cộng với số lượng trường hợp lớn, đồng nghĩa với chi phí cao, có thể khiến công việc này nằm ngoài tầm với của nhiều nhà nghiên cứu. Ví dụ, vào năm 2023, một Nhóm nghiên cứu đã mô tả việc đào tạo pLM 100 tỷ thông số trên 768 GPU A100 trong 164 ngày! May mắn thay, trong nhiều trường hợp, chúng ta có thể tiết kiệm thời gian và nguồn lực bằng cách điều chỉnh pLM hiện có cho phù hợp với nhiệm vụ cụ thể của mình. Kỹ thuật này được gọi là tinh chỉnhvà cũng cho phép chúng tôi mượn các công cụ nâng cao từ các loại mô hình ngôn ngữ khác.
Tổng quan về giải pháp
Vấn đề cụ thể mà chúng tôi giải quyết trong bài viết này là nội địa hóa dưới tế bào: Với một chuỗi protein, liệu chúng ta có thể xây dựng một mô hình có thể dự đoán liệu nó sống ở bên ngoài (màng tế bào) hay bên trong tế bào không? Đây là một thông tin quan trọng có thể giúp chúng ta hiểu được chức năng và liệu nó có trở thành mục tiêu thuốc tốt hay không.
Chúng tôi bắt đầu bằng cách tải xuống tập dữ liệu công khai bằng cách sử dụng Xưởng sản xuất Amazon SageMaker. Sau đó, chúng tôi sử dụng SageMaker để tinh chỉnh mô hình ngôn ngữ protein ESM-2 bằng phương pháp đào tạo hiệu quả. Cuối cùng, chúng tôi triển khai mô hình này làm điểm cuối suy luận theo thời gian thực và sử dụng mô hình này để kiểm tra một số protein đã biết. Sơ đồ sau đây minh họa quy trình làm việc này.

Trong các phần sau, chúng ta sẽ thực hiện các bước để chuẩn bị dữ liệu đào tạo, tạo tập lệnh đào tạo và chạy công việc đào tạo SageMaker. Tất cả các mã đặc trưng trong bài đăng này đều có sẵn trên GitHub.
Chuẩn bị dữ liệu đào tạo
Chúng tôi sử dụng một phần của Bộ dữ liệu DeepLoc-2, chứa hàng nghìn protein SwissProt với các vị trí được xác định bằng thực nghiệm. Chúng tôi lọc các chuỗi chất lượng cao trong khoảng 100–512 axit amin:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Tiếp theo, chúng tôi mã hóa các chuỗi và chia chúng thành các tập huấn luyện và đánh giá:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Cuối cùng, chúng tôi tải dữ liệu đánh giá và đào tạo đã xử lý lên Dịch vụ lưu trữ đơn giản của Amazon (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Tạo kịch bản đào tạo
Chế độ tập lệnh SageMaker cho phép bạn chạy mã đào tạo tùy chỉnh của mình trong các bộ chứa khung máy học (ML) được tối ưu hóa do AWS quản lý. Đối với ví dụ này, chúng tôi điều chỉnh một tập lệnh hiện có để phân loại văn bản từ Ôm Mặt. Điều này cho phép chúng tôi thử một số phương pháp để nâng cao hiệu quả công việc đào tạo của mình.
Cách 1: Lớp tập tạ
Giống như nhiều bộ dữ liệu sinh học, dữ liệu DeepLoc được phân bổ không đồng đều, nghĩa là không có số lượng protein màng và protein không màng bằng nhau. Chúng tôi có thể lấy mẫu lại dữ liệu của mình và loại bỏ các bản ghi khỏi lớp đa số. Tuy nhiên, điều này sẽ làm giảm tổng dữ liệu huấn luyện và có khả năng ảnh hưởng đến độ chính xác của chúng tôi. Thay vào đó, chúng tôi tính toán trọng số của lớp trong quá trình đào tạo và sử dụng chúng để điều chỉnh tổn thất.
Trong tập lệnh đào tạo của chúng tôi, chúng tôi phân lớp Trainer lớp học từ transformers với một WeightedTrainer lớp có tính đến trọng số của lớp khi tính toán tổn thất entropy chéo. Điều này giúp ngăn chặn sự thiên vị trong mô hình của chúng tôi:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossCách 2: Tích lũy gradient
Tích lũy gradient là một kỹ thuật huấn luyện cho phép các mô hình mô phỏng quá trình huấn luyện trên quy mô lô lớn hơn. Thông thường, kích thước lô (số lượng mẫu được sử dụng để tính toán độ dốc trong một bước huấn luyện) bị giới hạn bởi dung lượng bộ nhớ GPU. Với việc tích lũy độ dốc, trước tiên mô hình sẽ tính toán độ dốc trên các lô nhỏ hơn. Sau đó, thay vì cập nhật trọng số mô hình ngay lập tức, độ dốc sẽ được tích lũy qua nhiều đợt nhỏ. Khi độ dốc tích lũy bằng kích thước lô lớn hơn mục tiêu, bước tối ưu hóa được thực hiện để cập nhật mô hình. Điều này cho phép các mô hình đào tạo với số lượng lớn hơn một cách hiệu quả mà không vượt quá giới hạn bộ nhớ GPU.
Tuy nhiên, cần phải tính toán thêm cho các lượt chuyển tiếp và lùi theo lô nhỏ hơn. Kích thước lô tăng lên thông qua tích lũy gradient có thể làm chậm quá trình đào tạo, đặc biệt nếu sử dụng quá nhiều bước tích lũy. Mục đích là để tối đa hóa việc sử dụng GPU nhưng tránh tình trạng chậm quá mức do có quá nhiều bước tính toán độ dốc bổ sung.
Cách 3: Điểm kiểm tra độ dốc
Điểm kiểm tra độ dốc là một kỹ thuật giúp giảm bộ nhớ cần thiết trong quá trình đào tạo trong khi vẫn giữ thời gian tính toán hợp lý. Các mạng thần kinh lớn chiếm nhiều bộ nhớ vì chúng phải lưu trữ tất cả các giá trị trung gian từ quá trình chuyển tiếp để tính toán độ dốc trong quá trình truyền ngược. Điều này có thể gây ra vấn đề về bộ nhớ. Một giải pháp là không lưu trữ các giá trị trung gian này nhưng sau đó chúng phải được tính toán lại trong quá trình truyền ngược, việc này tốn rất nhiều thời gian.
Điểm kiểm tra độ dốc cung cấp một cách tiếp cận cân bằng. Nó chỉ lưu lại một số giá trị trung gian, được gọi là trạm kiểm soát, và tính toán lại những cái khác nếu cần. Do đó, nó sử dụng ít bộ nhớ hơn so với việc lưu trữ mọi thứ, nhưng cũng ít tính toán hơn so với việc tính toán lại mọi thứ. Bằng cách chọn chiến lược kích hoạt nào cho điểm kiểm tra, điểm kiểm tra độ dốc cho phép đào tạo các mạng thần kinh lớn với mức sử dụng bộ nhớ và thời gian tính toán có thể quản lý được. Kỹ thuật quan trọng này giúp việc huấn luyện các mô hình rất lớn có thể gặp phải hạn chế về bộ nhớ là khả thi.
Trong tập lệnh đào tạo của chúng tôi, chúng tôi bật kích hoạt độ dốc và điểm kiểm tra bằng cách thêm các tham số cần thiết vào TrainingArguments vật:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Phương pháp 4: Điều chỉnh LLM ở cấp độ thấp
Các mô hình ngôn ngữ lớn như ESM-2 có thể chứa hàng tỷ tham số rất tốn kém để đào tạo và chạy. Các nhà nghiên cứu đã phát triển một phương pháp đào tạo có tên là Thích ứng cấp thấp (LoRA) để tinh chỉnh những mô hình khổng lồ này hiệu quả hơn.
Ý tưởng chính đằng sau LoRA là khi tinh chỉnh mô hình cho một nhiệm vụ cụ thể, bạn không cần cập nhật tất cả các tham số ban đầu. Thay vào đó, LoRA bổ sung các ma trận mới nhỏ hơn vào mô hình để chuyển đổi đầu vào và đầu ra. Chỉ những ma trận nhỏ hơn này mới được cập nhật trong quá trình tinh chỉnh, nhanh hơn nhiều và sử dụng ít bộ nhớ hơn. Các thông số mô hình ban đầu vẫn giữ nguyên.
Sau khi tinh chỉnh bằng LoRA, bạn có thể hợp nhất các ma trận nhỏ đã điều chỉnh lại thành mô hình ban đầu. Hoặc bạn có thể tách chúng ra nếu muốn nhanh chóng tinh chỉnh mô hình cho các tác vụ khác mà không quên các tác vụ trước đó. Nhìn chung, LoRA cho phép LLM thích ứng một cách hiệu quả với các nhiệm vụ mới với chi phí thấp hơn thông thường.
Trong tập lệnh đào tạo của chúng tôi, chúng tôi định cấu hình LoRA bằng cách sử dụng PEFT thư viện từ Ôm Mặt:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Gửi công việc đào tạo SageMaker
Sau khi xác định tập lệnh đào tạo của mình, bạn có thể định cấu hình và gửi công việc đào tạo SageMaker. Đầu tiên, chỉ định các siêu tham số:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Tiếp theo, xác định số liệu nào cần thu thập từ nhật ký đào tạo:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Cuối cùng, xác định công cụ ước tính Ôm mặt và gửi nó đi đào tạo về loại phiên bản ml.g5.2xlarge. Đây là loại phiên bản tiết kiệm chi phí được cung cấp rộng rãi ở nhiều Khu vực AWS:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)Bảng sau đây so sánh các phương pháp đào tạo khác nhau mà chúng tôi đã thảo luận và ảnh hưởng của chúng đến các yêu cầu về thời gian chạy, độ chính xác và bộ nhớ GPU trong công việc của chúng tôi.
| Cấu hình | Thời gian có thể tính phí (phút) | Đánh giá độ chính xác | Mức sử dụng bộ nhớ GPU tối đa (GB) |
| Mô hình cơ sở | 28 | 0.91 | 22.6 |
| Cơ sở + GA | 21 | 0.90 | 17.8 |
| Cơ sở + GC | 29 | 0.91 | 10.2 |
| Căn cứ + LoRA | 23 | 0.90 | 18.6 |
Tất cả các phương pháp đều tạo ra mô hình có độ chính xác đánh giá cao. Việc sử dụng LoRA và kích hoạt gradient đã giảm thời gian chạy (và chi phí) lần lượt là 18% và 25%. Việc sử dụng điểm kiểm tra độ dốc đã giảm mức sử dụng bộ nhớ GPU tối đa xuống 55%. Tùy thuộc vào những ràng buộc của bạn (chi phí, thời gian, phần cứng), một trong những cách tiếp cận này có thể hợp lý hơn những cách tiếp cận khác.
Mỗi phương pháp này tự hoạt động tốt, nhưng điều gì sẽ xảy ra khi chúng ta sử dụng chúng kết hợp? Bảng sau đây tóm tắt các kết quả.
| Cấu hình | Thời gian có thể tính phí (phút) | Đánh giá độ chính xác | Mức sử dụng bộ nhớ GPU tối đa (GB) |
| Tất cả các phương pháp | 12 | 0.80 | 3.3 |
Trong trường hợp này, chúng tôi thấy độ chính xác giảm 12%. Tuy nhiên, chúng tôi đã giảm thời gian chạy xuống 57% và mức sử dụng bộ nhớ GPU xuống 85%! Đây là mức giảm lớn cho phép chúng tôi đào tạo trên nhiều loại phiên bản có hiệu quả về mặt chi phí.
Làm sạch
Nếu bạn đang theo dõi bằng tài khoản AWS của chính mình, hãy xóa mọi điểm cuối và dữ liệu suy luận theo thời gian thực mà bạn đã tạo để tránh phải trả thêm phí.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Kết luận
Trong bài đăng này, chúng tôi đã trình bày cách tinh chỉnh các mô hình ngôn ngữ protein như ESM-2 một cách hiệu quả cho một nhiệm vụ phù hợp về mặt khoa học. Để biết thêm thông tin về cách sử dụng thư viện Transformers và PEFT để huấn luyện pLMS, hãy xem các bài đăng Học sâu với protein và ESMBind (ESMB): Sự thích ứng ở cấp độ thấp của ESM-2 để dự đoán vị trí liên kết với protein trên blog Ôm Mặt. Bạn cũng có thể tìm thêm ví dụ về việc sử dụng máy học để dự đoán các đặc tính của protein trong Phân tích Protein tuyệt vời trên AWS Kho lưu trữ GitHub.
Lưu ý
 Brian trung thành là Kiến trúc sư giải pháp AI/ML cấp cao trong nhóm Khoa học đời sống và chăm sóc sức khỏe toàn cầu tại Amazon Web Services. Ông có hơn 17 năm kinh nghiệm trong lĩnh vực công nghệ sinh học và học máy, đồng thời đam mê giúp khách hàng giải quyết các thách thức về gen và protein. Trong thời gian rảnh rỗi, anh ấy thích nấu ăn và ăn uống với bạn bè và gia đình.
Brian trung thành là Kiến trúc sư giải pháp AI/ML cấp cao trong nhóm Khoa học đời sống và chăm sóc sức khỏe toàn cầu tại Amazon Web Services. Ông có hơn 17 năm kinh nghiệm trong lĩnh vực công nghệ sinh học và học máy, đồng thời đam mê giúp khách hàng giải quyết các thách thức về gen và protein. Trong thời gian rảnh rỗi, anh ấy thích nấu ăn và ăn uống với bạn bè và gia đình.
- Phân phối nội dung và PR được hỗ trợ bởi SEO. Được khuếch đại ngay hôm nay.
- PlatoData.Network Vertical Generative Ai. Trao quyền cho chính mình. Truy cập Tại đây.
- PlatoAiStream. Thông minh Web3. Kiến thức khuếch đại. Truy cập Tại đây.
- Trung tâmESG. Than đá, công nghệ sạch, Năng lượng, Môi trường Hệ mặt trời, Quản lý chất thải. Truy cập Tại đây.
- PlatoSức khỏe. Tình báo thử nghiệm lâm sàng và công nghệ sinh học. Truy cập Tại đây.
- nguồn: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



