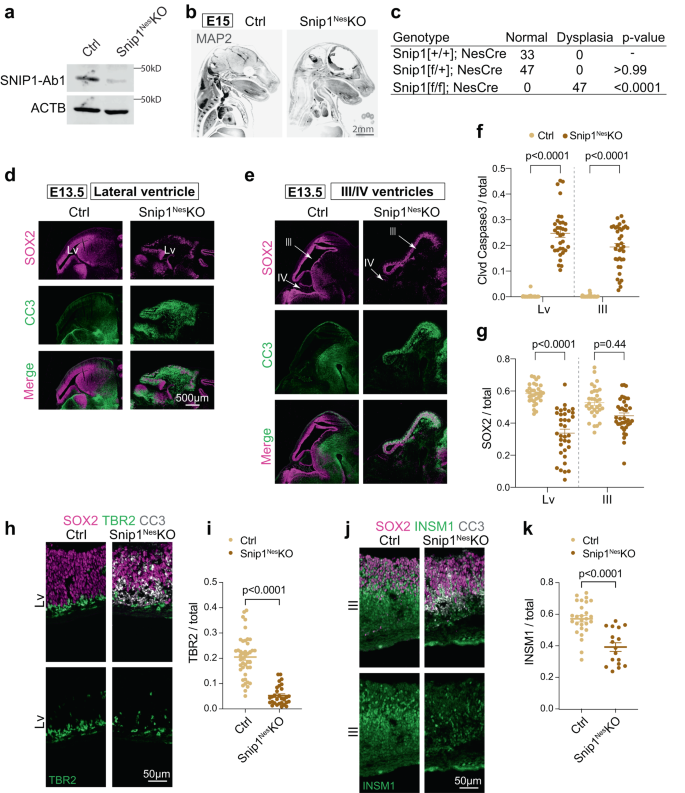
SNIP1 là cần thiết cho sự tồn tại của các NPC trong não phôi thai chuột
Đầu tiên chúng tôi kiểm tra biểu hiện của snip1 trong não phôi thai chuột bằng RNAscope40. Vào ngày phôi thai E11.5 và E13.5, snip1 các bản phiên mã được biểu thị trong hầu hết các tế bào và được biểu hiện mạnh mẽ trong biểu mô thần kinh lót trong tâm thất, nơi cư trú của các NPC (Hình bổ sung. 1b, c). Để nghiên cứu SNIP1 trong não phôi thai, chúng tôi đã sử dụng làm tổ (Không) ::Cre để làm cạn kiệt SNIP1 một cách có điều kiện trong các NPC, sau đây được gọi là snip1Không–KO. Nes::Cre được thể hiện trong các NPC để kết hợp lại các trang web flox và loại bỏ exon 2 của snip1 (Sung. 1a, Hình bổ sung. 1 ngày – thứ sáu, Hình bổ sung. 2a–d). Đến E15, snip1Không-KO phôi cho thấy sự mỏng đi nghiêm trọng của các mô não và chứng loạn sản với sự xâm nhập 100% (Hình. 1b, c, p < 0.0001 theo phép thử chính xác của Fisher). Để hiểu nền tảng tế bào của chứng loạn sản não ở snip1Không–KO, chúng tôi đã kiểm tra sự tăng sinh tế bào và quá trình tự hủy của NPC. Các NPC được xác định bằng biểu hiện của dấu hiệu tế bào gốc thần kinh SRY-box 2 (SOX2). Để xác định các tế bào đang tăng sinh in vivo, chúng tôi đã tiêm BrdU vào những con mẹ đang mang thai và/hoặc phát hiện dấu hiệu tăng sinh Ki67. Định lượng các dấu hiệu này trong neuroepithelia không cho thấy sự khác biệt đáng kể trong các NPC tăng sinh trong kiểm soát anh chị em và snip1Không–Phôi KO (Hình bổ sung. 2e–h).
a WB kiểm soát và snip1Không-KO NPC tại E13.5. Ít nhất 5 lần nhắc lại đối chứng và snip1Không-KO NPC cho thấy kết quả tương tự. SNIP1-Ab1; kháng thể chống SNIP1 từ ProteinTech. b NẾU MAP2 được kiểm soát và snip1Không-KO phôi ở E15. Phôi được xóa bằng phương pháp iDISCO và được chụp ảnh bằng kính hiển vi tấm sáng. Hai lần lặp lại IF cho kết quả tương tự. Thanh, 2 mm. c Sự xâm nhập của chứng loạn sản não ở phôi E13.5. Loạn sản não được xác định bởi sự mỏng đi của mô não. Ý nghĩa thống kê được tính toán bằng thử nghiệm chính xác của Fisher. d–e NẾU của SOX2 và tách caspase 3 (CC3) trong các phương pháp lạnh đối xứng dọc của não E13.5. Các vùng mầm xung quanh tâm thất bên (Lv, não trước), tâm thất thứ ba, (não giữa) và tâm thất thứ tư (não sau) đã được kiểm tra. Thanh, 500 m. f, g Định lượng các tế bào dương tính với CC3 và SOX2 trong lớp biểu mô thần kinh của tâm thất kiểm soát và snip1Không-KO phôi ở E13.5. Nhuộm DAPI được sử dụng để đếm tổng số ô. Mỗi điểm dữ liệu đại diện cho một hình ảnh. Tám phôi đối chứng và 7 snip1Không-KO phôi đã được phân tích. TRONG (f), đối với tâm thất bên, n = 38 hình ảnh (kiểm soát) và n = 34 (snip1Không-KO); cho tâm thất thứ ba, n = 38 (kiểm soát) và n = 36 (snip1Không-KO). TRONG (g), đối với tâm thất bên, n = 33 (kiểm soát) và n = 35 (snip1Không-KO); cho tâm thất thứ ba, n = 32 (kiểm soát) và n = 37 (snip1Không-KO). Dữ liệu được trình bày dưới dạng trung bình ± SEM và ANOVA hai chiều được sử dụng để phân tích thống kê. h–j NẾU của SOX2 và CC3 được phủ bằng các dấu hiệu dòng dõi thần kinh TBR2 và INSM1 của bộ não E13.5. Thanh, 50 m. i, k Định lượng các tế bào dương tính với TBR2 hoặc INSM1 trong lớp biểu mô thần kinh của tâm thất bên hoặc tâm thất thứ ba. Mỗi điểm dữ liệu đại diện cho một hình ảnh. Năm đến 8 phôi đối chứng và 3-7 snip1Không-KO phôi đã được phân tích. TRONG (i), n = 43 hình ảnh (kiểm soát) và n = 30 (snip1Không-KO); TRONG (k), n = 27 (kiểm soát) và n = 17 (snip1Không-KO). Dữ liệu được trình bày dưới dạng trung bình ± SEM và ANOVA hai chiều được sử dụng để phân tích thống kê. Dữ liệu nguồn được cung cấp trong tệp Dữ liệu nguồn (a, f, g, i, k).
Tiếp theo, chúng tôi đã thăm dò quá trình tự hủy bằng IF của (cl) -caspase 3. Tại E13.5, tất cả các tâm thất của snip1Không–KO hiển thị cảm ứng mạnh mẽ của cl-caspase 3 trong các vùng dưới màng cứng của neuroepithelia (Hình. 1 ngày – thứ sáu). Các tín hiệu cl-caspase 3 chồng lấp các NPC dương tính với SOX2 và các tổ tiên trung gian dương tính với TBR2 (Hình bổ sung. 2i–l), đã giảm rõ rệt trong snip1Không–KO biểu mô thần kinh (Hình. 1d–tôi). Các tổ tiên trung gian dương tính với INSM1 đã giảm tương tự trong snip1Không–KO não giữa (Hình. 1j, k). Những dữ liệu này cho thấy SNIP1 ngăn chặn quá trình chết theo chương trình ở NPC và các tổ tiên trung gian trong não đang phát triển.
Để nắm bắt các sự kiện trước đó của snip1Không–KO não, chúng tôi đã kiểm tra cl-caspase 3 trong não phôi thai E11.5. Đến E11.5, tín hiệu cl-caspase 3 đã được phát hiện trên khắp snip1Không–Não KO (Hình bổ sung. 3a–d). Như Không::Cre được bật bởi E10.541, sự cạn kiệt SNIP1 có khả năng gây ra quá trình chết theo chương trình trong vòng 24 giờ. Việc định lượng các tế bào dương tính với SOX2 ở tâm thất bên, thứ ba và thứ tư cho thấy rằng chỉ các NPC trong tâm thất thứ tư mới bị cạn kiệt bởi E11.5 (Hình bổ sung. 3e). Sau đây, chúng tôi tập trung phân tích tâm thất bên và tâm thất thứ ba để nghiên cứu kiểm soát quá trình tự hủy của SNIP1.
Để xác định rằng sự suy giảm SNIP1 liên tục gây ra hiện tượng chết theo chương trình đã quan sát được, chúng tôi đã sử dụng Emx1::Cre để làm cạn kiệt SNIP1 một cách có điều kiện trong các NPC của telencephalon ở lưng42. Các snip1Emx1–Phôi KO cũng cho thấy cảm ứng mạnh mẽ của quá trình tự hủy, mất các tổ tiên trung gian dương tính với TBR2 và chứng loạn sản của tiền đình (Hình bổ sung. 3f–k). Những phát hiện này hỗ trợ rằng trong não chuột đang phát triển, cảm ứng apoptosis và chứng loạn sản là đặc trưng cho sự suy giảm SNIP1. Những dữ liệu này hỗ trợ thêm cho kết luận của chúng tôi rằng SNIP1 là cần thiết cho cơ chế chống apoptotic và hỗ trợ sinh tồn trong NPC và các tổ tiên trung gian.
SNIP1 ngăn chặn chương trình apoptosis nội tại
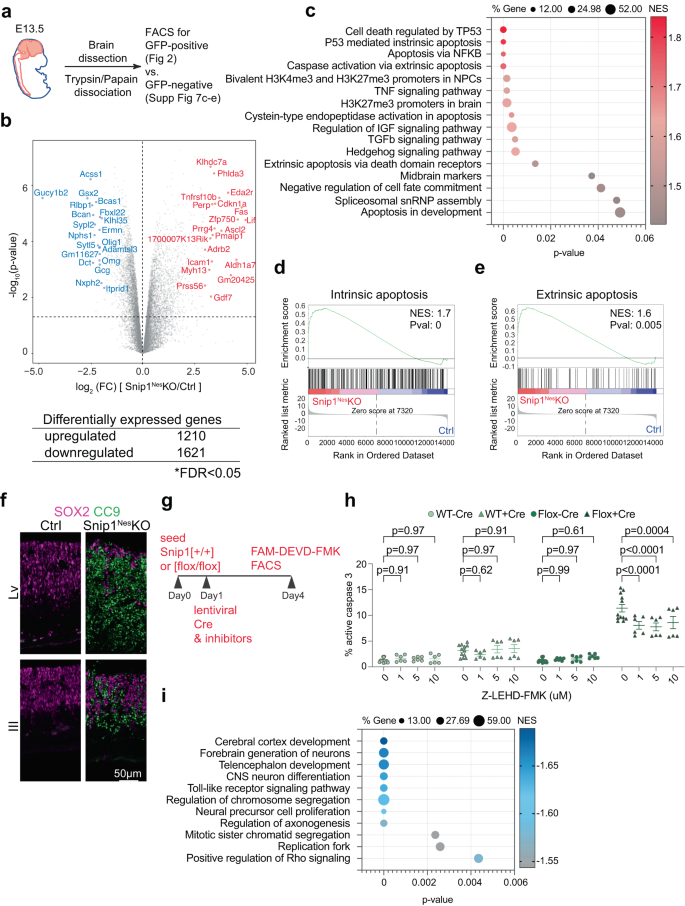
Để làm sáng tỏ nền tảng phân tử của khiếm khuyết snip1Không–KO NPC, chúng tôi đã thực hiện giải trình tự RNA (RNA-seq) của các NPC dương tính với SOX2 được sắp xếp từ E13.5 snip1Không–KO và điều khiển anh chị em (Hình. 2a, Hình bổ sung. 4a). Chúng tôi đã phân tích các gen có giá trị đếm trên một triệu (CPM) > 1 trong cả kiểm soát hoặc snip1Không–KO NPC. Sử dụng tiêu chí về tỷ lệ phát hiện sai (FDR) < 0.05 để so sánh từng bộ dữ liệu 4 lần lặp lại từ kiểm soát và snip1Không-KO NPC, chúng tôi đã xác định được 1210 gen điều hòa ngược và 1621 gen điều hòa giảm trong snip1Không-KO (Hình. 2b, Hình bổ sung. 4b–d).
a Sơ đồ của bộ sưu tập NPC não. b Biểu đồ núi lửa và số lượng gen được biểu hiện khác nhau giữa đối chứng và snip1Không-KO NPC. Bảng này cho thấy số lượng gen vượt qua ngưỡng FDR < 0.05. FDR được tính theo phương pháp Stewamini & Hochberg. P các giá trị được tính bằng Voom-limma hai mặt t thử nghiệm. c, i Biểu đồ bong bóng của các bộ gen được làm giàu trong các gen được điều hòa tăng và các gen được điều hòa giảm trong snip1Không-KO so với NPC điều khiển. Các gen biểu hiện khác nhau được xếp hạng đầu tiên theo sự thay đổi nếp gấp của chúng, p-giá trị và mức biểu thức trước khi Phân tích làm giàu bộ gen (GSEA) được thực hiện. P-giá trị được tính toán bằng phép thử hoán vị bên phải với điều chỉnh FDR. d, e GSEA đại diện của các gen được điều hòa trong snip1Không-KO so với NPC điều khiển. Các gen được điều hòa đã được làm giàu trong các bộ gen liên quan đến cả quá trình tự hủy bên trong và bên ngoài. Các gen biểu hiện khác nhau được xếp hạng đầu tiên theo sự thay đổi nếp gấp của chúng và p-value trước khi GSEA được thực hiện. f NẾU của caspase 9 (CC9) đã phân tách được phủ SOX2 trong các phương pháp làm lạnh sagittal của não E13.5. Thanh, 50 m. g Sơ đồ tải nạp mCherry-Cre lentillin và điều trị bằng chất ức chế ở snip1[+/+] và snip1[flox/flox] NPC. h Tỷ lệ tế bào có caspase 3 đang hoạt động được định lượng bằng FACS. Chất ức chế Caspase 9 (Z-LEHD-FMK) đã được thêm vào ở các nồng độ khác nhau cùng với lentillin mCherry-Cre. Tỷ lệ dân số dương tính với FAM-FLICA (caspase 3 đang hoạt động) (trên tổng dân số) được hiển thị. N = 12 cho điều khiển DMSO và n = 6 cho phần còn lại của mẫu. Dữ liệu được trình bày dưới dạng trung bình ± SEM và ANOVA hai chiều được sử dụng để phân tích thống kê. Chiến lược gating và sơ đồ FACS đại diện được hiển thị trong Thông tin bổ sung (Hình bổ sung. 4e, Hình bổ sung 14a). Dữ liệu nguồn được cung cấp trong tệp Dữ liệu nguồn (c, h, i).
Phân tích làm giàu bộ gen (GSEA) tiết lộ rằng các gen được điều hòa trong snip1Không–Các NPC KO đã được làm phong phú thêm các chức năng liên quan đến quá trình chết theo chương trình qua trung gian p53, H3K27me3 hoặc các chất kích thích hóa trị hai trong NPC và não, các dấu hiệu não giữa, các hạt ribonucleoprotein hạt nhân nhỏ spliceosomal và các con đường truyền tín hiệu liên quan đến TNF, IGF, TGFβ và Hedgehog (Hình. 2c). Vì các dấu hiệu apoptotic bên trong và bên ngoài đã được làm giàu trong các gen được điều hòa trong snip1Không–Các NPC KO (Hình. 2d, e), chúng tôi đã kiểm tra việc kích hoạt hai con đường này. NẾU cho thấy rất ít tín hiệu của cl-caspase 8, một tác nhân của con đường tự hủy bên ngoài43,44 (Hình bổ sung. 5a) nhưng các tín hiệu mạnh mẽ của cl-caspase 9, một tác nhân của con đường tự chết theo chương trình nội tại45,46,47, trong suốt biểu mô thần kinh của tâm thất (Hình. 2f). Ngoài ra, định lượng cl-caspase 3 bằng FACS của nhuộm FAM-DEVD-FMK48,49 cho thấy rằng trong khi ức chế caspase 8 làm thay đổi nhẹ quá trình chết theo chương trình, thì việc ức chế caspase 9 làm giảm mạnh quá trình chết theo chương trình ở snip1Không–Các NPC KO (Hình. 2g, h, Hình bổ sung. 4e, Hình bổ sung. 5b). Chất ức chế caspase 9, Z-LEHD-FMK, đã ức chế hiệu quả caspase 9 ở mức 1 μM và gây độc tế bào ở mức 20 và 40 μM (Hình. 2h, Hình bổ sung. 4f). Để xác định nguyên nhân làm tăng quá trình chết theo chương trình, chúng tôi không phát hiện thấy sự gia tăng thiệt hại DNA (Hình bổ sung. 5c–f) nhưng đã phát hiện thấy tín hiệu p53 tăng mạnh trong các NPC dương tính với SOX2 (Hình bổ sung. 5g–tôi). Những dữ liệu này cho thấy rằng snip1Không–Phôi KO hiển thị sự kiểm soát không được kiểm soát đối với quá trình tự hủy nội tại qua trung gian p53 trong các NPC. Chúng tôi đề xuất rằng SNIP1 chủ yếu ngăn chặn quá trình tự hủy theo chương trình nội tại như một phần của chương trình phát triển thần kinh.
Gen điều hòa ngược ở snip1Không–Các NPC KO đã được làm phong phú thêm các chức năng liên quan đến sự phát triển của não trước và vỏ não, sự biệt hóa tế bào thần kinh CNS, sự phân ly nhiễm sắc thể, sự tăng sinh của NPC, sự hình thành sợi trục, ngã ba sao chép và các con đường truyền tín hiệu liên quan đến TLR và Rho (Hình XNUMX). 2i). Mặc dù snip1Không–Các mô ở não trước của KO biểu hiện sự mỏng đi nghiêm trọng do hậu quả của quá trình chết theo chương trình, dấu hiệu não trước FOXG1 và dấu hiệu giữa / não sau OTX2 được phát hiện tương tự giữa đối chứng và snip1Không–Bộ não KO (Hình bổ sung. 5j). Những dữ liệu này cho thấy rằng sự cạn kiệt SNIP1 không làm thay đổi đặc điểm kỹ thuật của tiền đình.
Các gen điều hòa giảm khác trong snip1Không–Các NPC KO đã tham gia vào việc kiểm soát khả năng tự đổi mới (Hình. 2i). đặc trưng của snip1Không–Các NPC KO trong ống nghiệm cho thấy, so với các NPC đối chứng, các NPC được nuôi cấy snip1Không–Các NPC KO đã giảm biểu thức SOX2 (Hình bổ sung. 6a, b). Bằng cách cho phép các NPC hình thành các tế bào thần kinh ở trạng thái lơ lửng và thông qua việc truyền nối tiếp, chúng tôi đã quan sát thấy rằng số lượng tế bào thần kinh và diện tích mặt cắt ngang thấp hơn đáng kể trong snip1Không–KO so với đối chứng (Hình bổ sung. 6c–e). Thể hiện quá mức SNIP1 của con người (85% nhận dạng với SNIP1 của chuột) là đủ để giải cứu khả năng tự đổi mới trong môi trường nuôi cấy snip1Không–Các NPC KO (Hình bổ sung. 6f,g), đề xuất bảo tồn chức năng SNIP1 giữa người và chuột. Nhìn chung, chúng tôi kết luận rằng SNIP1 thúc đẩy sự sống của tế bào để duy trì khả năng tự đổi mới của NPC.
SNIP1 ngăn chặn sự khác biệt trong NPC
Bởi vì ở E13.5, não phôi trải qua quá trình hình thành thần kinh, chúng tôi đã kiểm tra dấu hiệu tế bào thần kinh chưa trưởng thành TUJ1. Độ dày tương đối của vùng dương tính TUJ1 không khác biệt đáng kể giữa snip1Không–KO và kiểm soát tại E13.5 (Hình bổ sung. 7a, b). Xét rằng snip1Không–Các NPC KO đang dần cạn kiệt, sự thiếu khác biệt này là một điều bất ngờ. Do đó, chúng tôi đã kiểm tra hiệu ứng phân tử của SNIP1 trong việc phân biệt các tế bào thần kinh đã mất biểu hiện của dấu hiệu NPC SOX2. Chúng tôi đã thực hiện RNA-seq của SOX2: tế bào âm tính GFP được sắp xếp từ E13.5 snip1Không–Bộ não điều khiển KO và anh chị em (Hình. 2a, Hình bổ sung. 4a). Sử dụng các tiêu chí thay đổi lần> 2 và p < 0.05 để so sánh mỗi bộ dữ liệu 2 lần lặp lại từ điều khiển và snip1Không-KO, chúng tôi đã xác định được 658 gen điều hòa ngược và 150 gen điều hòa giảm ở snip1Không-KO (Hình bổ sung. 7c). GSEA tiết lộ rằng các gen được điều chỉnh tăng trong snip1Không–Các tế bào KO đã được làm phong phú thêm các chức năng liên quan đến giải phóng mặt bằng apoptotic, đặc điểm kỹ thuật và sự khác biệt của tế bào thần kinh, các dấu hiệu trung gian và các chất kích thích mật độ CpG cao đã biết chiếm bởi các dấu hiệu hóa trị hai (H3K27me3 và H3K4me3) trong NPC50 (Hình bổ sung. 7d). Gen điều hòa ngược ở snip1Không–Các tế bào KO đã được làm phong phú thêm các chức năng liên quan đến spliceosome, dịch mã và ribosome, tổ chức nucleosome và quá trình tự hủy thông qua p21 chứ không phải p53 (Hình bổ sung. 7e). Những kết quả này cho thấy rằng SNIP1 ngăn chặn quá trình chết theo chương trình, đặc điểm kỹ thuật và sự khác biệt của tế bào thần kinh, các chương trình di truyền não giữa và các gen chiếm H3K27me3. Tại E13.5, mặc dù quá trình chết theo chương trình được điều chỉnh tăng làm giảm snip1Không–KO NPC và các tổ tiên trung gian, phần còn lại của chúng tạo ra các tế bào có đặc điểm kỹ thuật và sự biệt hóa tế bào thần kinh được điều chỉnh tăng. Những sự kết hợp này có khả năng dẫn đến không có sự khác biệt rõ ràng về độ dày vỏ não dương tính với TUJ1 giữa snip1Không–KO và kiểm soát.
Tiếp theo, chúng tôi đã thử nghiệm SNIP1 thông qua phân biệt NPC trong ống nghiệm. Chúng tôi đã cạn kiệt SNIP1 vào Ngày 1 hoặc Ngày 5 bằng cách tải nạp snip1[flox/flox] Các NPC có kiểm soát lentivirus hoặc Cre và biểu hiện gen được định hình vào Ngày 14 (Hình bổ sung. 7f). QPCR định lượng cho thấy rằng so với các tế bào điều khiển, các tế bào đã cạn kiệt SNIP1 đã điều hòa các dấu hiệu tế bào thần kinh và thần kinh đệm chứ không phải các dấu hiệu NPC (Hình bổ sung. 7g, h). Thời điểm cạn kiệt SNIP1 (Ngày 1 so với Ngày 5) không ảnh hưởng đến kiểu biểu hiện gen này (Hình bổ sung. 7g, h). Những kết quả này hỗ trợ thêm rằng SNIP1 ngăn chặn sự hình thành thần kinh trong các NPC. Khi loại bỏ toàn cầu (KO) Snip1 trong phôi cá ngựa vằn làm giảm các tế bào thần kinh GABAergic và glutamatergic35, chúng tôi đã hỏi liệu sự cạn kiệt SNIP1 có làm thay đổi đặc điểm kỹ thuật của dòng tế bào thần kinh phụ ở chuột hay không. Do mất mô não nghiêm trọng trong snip1Không–KO, chúng tôi không thể phân tích mạnh mẽ sự phát triển của não bộ ngoài E13.5. Tại E13.5, mức độ phiên mã của các dấu hiệu tế bào thần kinh GABAergic Gad1 và Slc6a1 thấp hơn trong snip1Không–KO và các dấu hiệu tế bào thần kinh glutamatergic không khác nhau giữa snip1Không–KO và kiểm soát (Hình bổ sung. 7i,j). Định lượng miễn dịch huỳnh quang cho thấy các tế bào dương tính với GABA- (GABAergic neuron marker) thấp hơn đáng kể trong snip1Không-KO (Hình bổ sung. 7k, tôi), gợi ý rằng sự tham gia của SNIP1 trong việc chỉ định các dòng tế bào thần kinh phụ có thể được bảo tồn ở các loài bậc cao hơn.
SNIP1 trực tiếp điều chỉnh các gen có sửa đổi H3K27
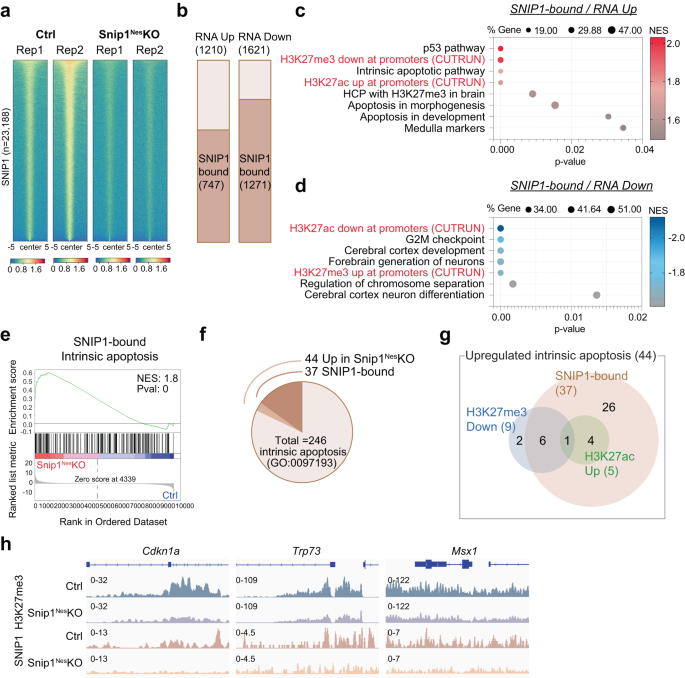
Để xác định xem các protein SNIP1 có liên kết trực tiếp với các locus gen để điều chỉnh biểu hiện của chúng hay không, chúng tôi đã lập hồ sơ phân phối SNIP1 trên toàn bộ gen bằng CUT&RUN51 in snip1Không-KO và điều khiển NPC. Sử dụng SICER52 và MACS253 với FDR < 0.05 để so sánh 2 bộ dữ liệu, mỗi bộ dữ liệu từ đối chứng và snip1Không–KO, chúng tôi đã xác định được 23,188 vùng giới hạn SNIP1 trong các NPC kiểm soát và chỉ 4187 vùng trong snip1Không–Các NPC KO (Hình bổ sung. 8a c). Các vùng 4187 chiếm 18% trong số các vùng được kiểm soát và là hệ quả của các protein SNIP1 còn sót lại trong snip1Không–Các NPC KO (Hình. 1a). Bản đồ nhiệt cho thấy tín hiệu SNIP1 CUT&RUN giảm mạnh trong snip1Không-KO, cho thấy tính đặc hiệu cao của SNIP1 CUT&RUN trong NPC (Hình. 3a). Khoảng 50% các đỉnh giới hạn SNIP1 nằm trong các vùng khởi động (trong vòng 2 kb của các vị trí bắt đầu phiên mã), 7.4% nằm ở exon, 23.3% ở các intron, 0.7% ở các vị trí kết thúc phiên mã, 9.7% ở 5′ xa (2–50 kb từ một gen), 3.4% ở các vùng 3′ xa (2–50 kb từ một gen) và 5.5% ở các vùng xen kẽ (vượt quá 50 kb từ một gen) (Hình bổ sung. 8d). Chỉ 18.6, 1% các đỉnh giới hạn SNIPXNUMX nằm ở xa một gen (Hình bổ sung. 8d).
a Bản đồ nhiệt thể hiện cường độ liên kết của 2 bản sao sinh học của SNIP1 CUT&RUN trong điều khiển và snip1Không–KO NPC. Cường độ liên kết cho 5 kb ở hai bên của tất cả 23,188 đỉnh SNIP1 CUT&RUN được hiển thị. Màu xanh biểu thị cường độ thấp và màu đỏ biểu thị cường độ cao. b Biểu đồ thanh hiển thị số lượng gen điều hòa và điều hòa giảm (snip1Không–KO so với kiểm soát, FDR < 0.05) bị ràng buộc bởi SNIP1 tại cơ thể gen của chúng. c, d Biểu đồ bong bóng của các bộ gen được làm giàu trong các gen liên kết với SNIP1 đã trở thành gen điều hòa ngược và gen điều hòa giảm trong snip1Không-KO so với NPC điều khiển. Khi thêm dữ liệu H3K27me3/ac CUT&RUN của chúng tôi vào bộ gen GSEA, các mức H3K27me3 và H3K27ac lần lượt cho thấy sự tương quan và tương quan với biểu hiện gen. Dữ liệu nguồn được cung cấp trong tệp Dữ liệu nguồn. e GSEA đại diện của các gen được điều hòa trong snip1Không-KO NPC so với NPC điều khiển bị ràng buộc bởi SNIP1 trong NPC điều khiển. Các gen gây chết tế bào theo chương trình nội tại chủ yếu liên kết với SNIP1 và được làm phong phú thêm trong các gen được điều hòa trong snip1Không-KO NPC. Các gen biểu hiện khác nhau được xếp hạng đầu tiên theo sự thay đổi nếp gấp của chúng và p-value trước khi GSEA được thực hiện. f Biểu đồ hình tròn cho thấy tỷ lệ các gen gây chết tế bào nội tại được điều chỉnh tăng trong snip1Không-KO NPC và/hoặc bị ràng buộc bởi SNIP1. g Biểu đồ Venn hiển thị số lượng gen gây chết tế bào theo chương trình nội tại được điều hòa tăng theo 3 loại. Sử dụng dữ liệu SNIP1, H3K27me3 và H3K27ac CUT&RUN của chúng tôi, 44 gen được phân loại thành 1) NPC kiểm soát gắn SNIP1, 2) giảm mức H3K27me3 trong snip1Không-KO so với NPC điều khiển (p < 0.05) và/hoặc 3) tăng mức H3K27ac trong snip1Không-KO so với NPC điều khiển (p <0.05). h Các rãnh H3K27me3 và SNIP1 CUT&RUN được Trình xem bộ gen tích hợp (IGV) trực quan hóa tại các gen gây chết tế bào chết theo chương trình nội tại được điều chỉnh tăng. Cdkn1a, Chr17: 29,090,888−29,095,850. trp73, Chr4: 154,132,565−154,143,373. Msx1, Chr5: 37,818,429−37,828,924.
Tiếp theo, chúng tôi đã kiểm tra xem tỷ lệ chiếm dụng SNIP1 có tương quan với biểu hiện gen hay không. Trong số 1210 gen điều hoà ngược và 1621 gen điều hoà giảm ở snip1Không-KO, 747 (62%) và 1,271 (78%) lần lượt là các mục tiêu SNIP1 trong các NPC điều khiển (Hình XNUMX). 3b). Những chồng chéo này là đáng kể, với p = 5.55e-25 và 2.24e-155 tương ứng bằng thử nghiệm siêu bội (với tổng số 21,636 gen biểu hiện), cho thấy rằng SNIP1 chiếm các gen này để điều chỉnh biểu hiện của chúng. GSEA đã chỉ ra rằng các mục tiêu SNIP1 đã được điều chỉnh lại trong snip1Không–KO đã được làm giàu trong con đường p53, tủy / midbrain và quá trình tự hủy (Hình XNUMX). 3c), trong khi các mục tiêu SNIP1 đã được điều chỉnh giảm trong snip1Không–KO đã được làm phong phú trong điểm kiểm tra G2/M, sự phát triển của vỏ não và sự phân ly nhiễm sắc thể (Hình XNUMX). 3d). Trong số 1,621 gen điều hòa giảm ở snip1Không–KO, 1,093 bị ràng buộc SNIP1 (không bị ràng buộc PRC2) và được làm phong phú trong các chương trình di truyền ở điểm kiểm tra G2-M, trục chính phân bào, các con đường truyền tín hiệu chính cho sự phát triển thần kinh và quá trình tự hủy (Hình bổ sung. 8e), gợi ý rằng SNIP1 thúc đẩy biểu hiện của chúng. Các gen gây chết tế bào nội tại đã được làm giàu trong các mục tiêu SNIP1 đã được điều chỉnh lại trong snip1Không-KO (Hình. 3e, f, Hình bổ sung. 8f,g). Trong số 44 gen trong bộ gen gây chết tế bào tự chết nội tại, 37 gen khởi đầu bị ràng buộc bởi SNIP1 trong các NPC kiểm soát (p = 4.62e-5; Quả sung. 3g), gợi ý rằng SNIP1 trực tiếp ngăn chặn các gen này. Những dữ liệu này cho thấy SNIP1 trực tiếp điều chỉnh các chương trình di truyền quan trọng đối với quá trình tự hủy và chu kỳ tế bào.
Gen điều hòa ngược ở snip1Không-KO được làm phong phú thêm các gen có chất kích thích mật độ CpG cao là 1) H3K27me3 chiếm giữ trong não chuột phôi thai50 hoặc 2) hóa trị hai trong NPC chuột54 (Hình bổ sung. 9a, b). Điều này đã thôi thúc chúng tôi xem xét kỹ lưỡng liệu SNIP1 có kiểm soát các chương trình di truyền thông qua các sửa đổi H3K27 hay không. Chúng tôi đã định hình H3K27me3 và H3K27ac bằng CUT&RUN trong snip1Không-KO và kiểm soát các NPC dương tính với SOX2 (Hình bổ sung. 9c–g). Chúng tôi đã quan sát thấy mối tương quan chặt chẽ giữa các gen được điều hòa tăng, tỷ lệ chiếm H3K27me3 thấp hơn và tỷ lệ chiếm H3K27ac cao hơn, trong khi các gen bị điều hòa giảm có tỷ lệ chiếm H3K27me3 cao hơn và tỷ lệ chiếm H3K27ac thấp hơn trong snip1Không-KO (Hình. 3c, d, Hình bổ sung. 9h, tôi). Trong số 44 gen gây chết tế bào chết theo chương trình nội tại được điều hòa tăng cường, 9 gen cho thấy tỷ lệ chiếm hữu H3K27me3 giảm và 5 gen cho thấy tỷ lệ chiếm hữu H3K27ac tăng (p < 0.05, Hình. 3g, h, Hình bổ sung. 9j, k). Những dữ liệu này cho thấy SNIP1 tại chromatin kiểm soát các sửa đổi H3K27 và biểu hiện gen.
Các đường dẫn tín hiệu TGFβ và NFκB kiểm soát nội địa hóa SNIP1 thành chất nhiễm sắc
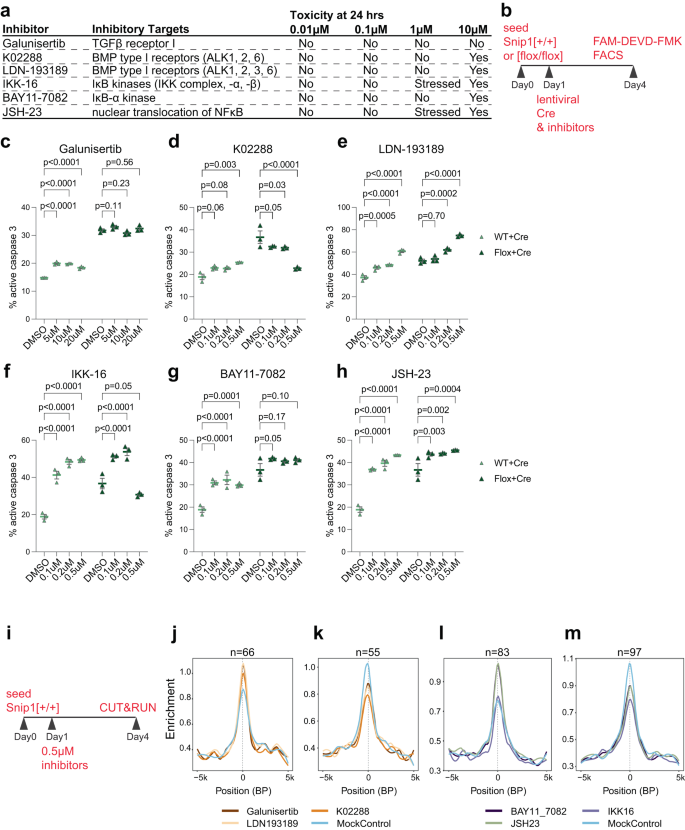
Khi SNIP1 tham gia vào TGFβ22,23,24,25 và NFκB26,27,28,29,30 đường dẫn tín hiệu, chúng tôi nhằm mục đích kiểm tra xem sự ức chế của chúng có ảnh hưởng đến quá trình tự hủy trong các NPC đã cạn kiệt SNIP1 hay không. Chúng tôi đã thử nghiệm 3 chất ức chế tín hiệu TGFβ hoặc NFκB về độc tính đối với NPC (Hình. 4a, b). Ở nồng độ 0.1–0.5 μM, K02288 nhắm mục tiêu TGFβ (chứ không phải Galunisertib hoặc LDN-193189) liên tục giảm quá trình tự hủy theo chương trình trong các NPC đã cạn kiệt SNIP1 (Hình. 4c–e). Ngược lại, 2 chất ức chế tín hiệu TGFβ và 3 chất ức chế tín hiệu NFκB làm tăng quá trình chết theo chương trình ở các NPC (Hình. 4 giờ sáng). Do đó, chúng tôi đã quyết định kiểm tra xem có bất kỳ chất ức chế nào trong số 6 chất ức chế làm thay đổi liên kết SNIP1 với chất nhiễm sắc hay không. Chúng tôi đã xử lý các NPC bằng DMSO hoặc chất ức chế ở mức 0.5 μM trong 3 ngày và thực hiện CUT&RUN để kiểm tra quá trình nội địa hóa SNIP1 trên chất nhiễm sắc (Hình. 4i). Phân tích 2 dữ liệu SNIP1 CUT&RUN sao chép cho mỗi điều kiện xử lý, chúng tôi đã sử dụng p < 0.05 để xác định sự khác biệt đáng kể và nhất quán trong tín hiệu SNIP1 CUT&RUN giữa phương pháp điều trị bằng chất ức chế và đối chứng. Chúng tôi đã xác định các thay đổi SNIP1 CUT & RUN do xử lý K02288 gây ra (Dữ liệu bổ sung 1). Chúng tôi cũng đã xác định được những thay đổi đáng kể trong SNIP1 CUT&RUN ở các gen liên kết với SNIP1 trùng lặp trong bất kỳ 2 phương pháp điều trị ức chế nào (Dữ liệu bổ sung 1). Cấu hình trung bình của tín hiệu SNIP1 CUT & RUN tại các trình khởi động gắn SNIP1 trong các NPC được xử lý bằng các chất ức chế khác nhau đã xác nhận rằng sự ức chế đối với TGFβ (Hình XNUMX). 4j, k) và NFκB (Hình. 4l, m) báo hiệu liên kết SNIP1 đã thay đổi đáng kể với các nhà quảng bá. Những dữ liệu này cho thấy rằng các đường dẫn tín hiệu TGFβ và NFκB kiểm soát SNIP1 liên kết với các locus gen cụ thể trong NPC.
a Các chất ức chế nhắm mục tiêu các thành phần trong đường dẫn tín hiệu TGFβ và NFκB và khả năng gây độc tế bào của chúng ở các nồng độ khác nhau. b Sơ đồ xét nghiệm chất ức chế. Vào Ngày 1, WT hoặc snip1[flox/flox] Các NPC được điều trị bằng chất ức chế hoặc DMSO và được tải nạp bằng lentivirus Cre để làm cạn kiệt SNIP1. FAM-DEVD-FMK đã được FACS sử dụng để thử nghiệm cl-caspase 3 vào Ngày thứ 4. c–h Tỷ lệ tế bào có caspase 3 đang hoạt động được định lượng bằng FACS. Các chất ức chế đã được thêm vào ở các nồng độ khác nhau cùng với lentillin mCherry-Cre. Tỷ lệ phần trăm FAM-FLICA (caspase 3 hoạt động)-/ dân số dương tính gấp đôi mCherry (trong số dân số dương tính với mCherry) được hiển thị. N = 3 cho mỗi lần điều trị. Dữ liệu được trình bày dưới dạng trung bình ± SEM và ANOVA hai chiều được sử dụng để phân tích thống kê. Các sơ đồ FACS đại diện được hiển thị trong Hình bổ sung. 14c, d. Dữ liệu nguồn được cung cấp trong tệp Dữ liệu nguồn. i Sơ đồ SNIP1 CUT&RUN có xử lý bằng chất ức chế. Vào ngày đầu tiên, các NPC được điều trị bằng kiểm soát DMSO hoặc các chất ức chế khác. Tái tạo SNIP1 CUT&RUN đã được thực hiện cho mỗi trong số 1 phương pháp điều trị vào ngày thứ 7. j–m Sơ đồ hồ sơ so sánh cường độ liên kết trung bình của SNIP1 trong NPC tại các mục tiêu giới hạn SNIP1 có liên kết SNIP1 cao hơn hoặc thấp hơn đáng kể trong chất ức chế so với điều trị kiểm soát DMSO. n cho biết số khu vực. Các khu vực được coi là mục tiêu SNIP1 thực sự khi mức SNIP1 giảm trong snip1Không-KO so với các NPC điều khiển với p <0.05.
PRC2 yêu cầu SNIP1 để bản địa hóa thành chất nhiễm sắc và lắng đọng H3K27me3
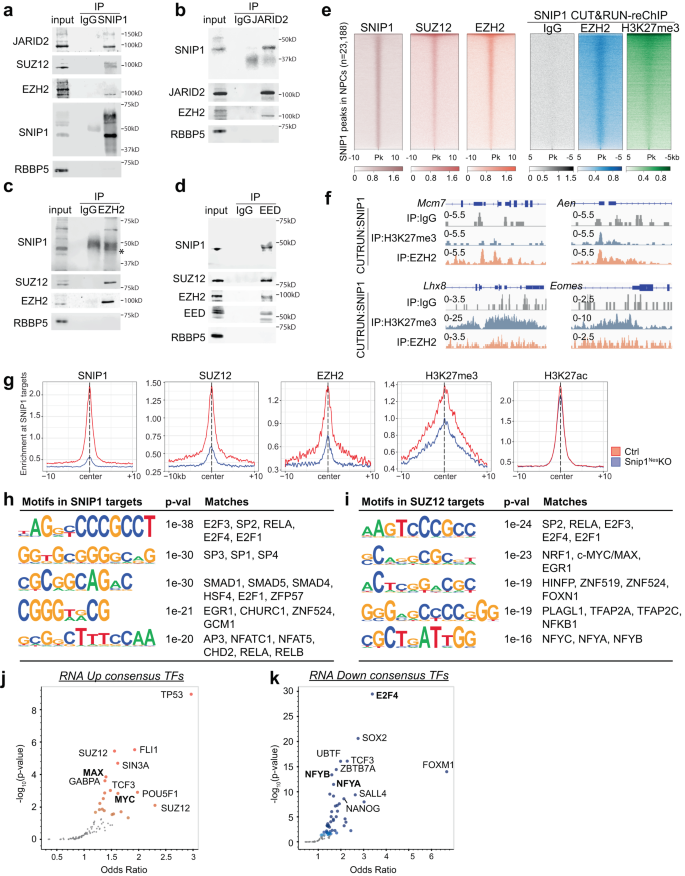
Chúng tôi đã nghiên cứu sự tương tác giữa SNIP1, H3K27 methyltransferase PRC2 và histone acetyltransferase p300 và CBP. Kháng thể chống SNIP1 được đồng ức chế miễn dịch với các tiểu đơn vị PRC2 đã biết Jumonji và miền tương tác giàu AT có chứa 2 (JARID2), Chất ức chế zeste 12 (SUZ12) và EZH2, nhưng không phải là RBBP5 kiểm soát âm tính trong chiết xuất hạt nhân NPC (Hình. 5a). Kháng thể chống SNIP1 không đồng kích thích miễn dịch với các tiểu đơn vị PRC2 trong các NPC đã cạn kiệt SNIP1 (Hình bổ sung. 10a), hỗ trợ tính đặc hiệu của kháng thể. SNIP2 và các tiểu đơn vị PRC2 khác được đồng ức chế miễn dịch với kháng thể Anti-JARID1, EZH2 hoặc EED nhưng không phải là RBBP5 đối chứng âm tính trong chiết xuất hạt nhân NPC (Hình XNUMX). 5b–d). Kháng thể chống p300 hoặc CBP không thể đồng kích thích miễn dịch SNIP1, cho thấy rằng trong các NPC, tương tác vật lý của chúng là không thể phát hiện được (Hình bổ sung. 10b, c). Liên kết PRC2–SNIP1 được hỗ trợ bởi các nghiên cứu proteomics khác cho thấy PRC2 đồng kích thích miễn dịch SNIP119,20.
một d Đồng kích thích miễn dịch theo sau là WB để kiểm tra sự tương tác giữa SNIP1 và PRC2. (a) SNIP1, (b) JARID2, (c) EZH2, hoặc (d) EED đã được kích thích miễn dịch trong chiết xuất hạt nhân NPC. RBBP5 là một kiểm soát tiêu cực. Dữ liệu nguồn được cung cấp dưới dạng tệp Dữ liệu nguồn. e Bản đồ nhiệt sắp xếp các đỉnh chất nhiễm sắc được làm giàu bằng SNIP1, SUZ12 và EZH2 trong các NPC. Các đỉnh từ SNIP1 CUT&RUN–reChIP với IgG, EZH2 và H3K27me3 được căn chỉnh theo các đỉnh giới hạn SNIP1. Cường độ cho 5 hoặc 10 kb ở hai bên của 23,188 đỉnh giới hạn SNIP1 được hiển thị. Màu tối biểu thị cường độ cao và màu sáng biểu thị cường độ thấp. f Các bản nhạc của SNIP1 CUT&RUN–reChIP với IgG, EZH2 và H3K27me3 được hiển thị bằng Trình xem bộ gen tích hợp (IGV). mcm7, Chr5: 138,169,717−138,173,621. Aen, Chr7: 78,894,526−78,898,271. Lhx8, Chr3: 154,325,066−154,334,835. Eomes, Chr9: 118,474,178−118,480,775. g Biểu đồ hồ sơ so sánh cường độ liên kết trung bình của SNIP1, PRC2 và H3K27me3/ac trong snip1Không-KO so với các NPC điều khiển tại các mục tiêu SNIP1. Các khu vực được coi là mục tiêu SNIP1 thực sự khi mức SNIP1 bị giảm trong snip1Không-KO so với các NPC điều khiển với p <0.05. h, i Mô típ của các khu vực giới hạn SNIP1 và SUZ12 nơi mức độ của chúng giảm đáng kể trong snip1Không–KO NPC với số lần thay đổi >2 và p < 0.05. Phân tích HOMER de novo đã được thực hiện và năm họa tiết có giá trị thấp nhất p-giá trị và có mô típ phù hợp với động vật có xương sống được liệt kê ở đây. j, k Sơ đồ núi lửa của các yếu tố phiên mã có liên kết với các gen biểu hiện khác nhau của chúng tôi (FDR <0.05) đã được báo cáo. Các gen đã được tìm kiếm dựa trên các TF đồng thuận của ENCODE và ChEA từ cơ sở dữ liệu ChIP-X bằng Enrichr56. Màu tối hơn hiển thị nhỏ hơn p-giá trị và điểm lớn thông qua p-giá trị <0.05. Các yếu tố phiên mã được in đậm đã được tìm thấy trong các phân tích mô-đun CUT&RUN của chúng tôi trong Hình 5h, i.
Chúng tôi đã kiểm tra xem SNIP1 có làm thay đổi biểu thức của tiểu đơn vị PRC2 hay không. Mặc dù sự cạn kiệt SNIP1 đã giảm xuống Ezh2 mức độ phiên mã, nó không làm thay đổi mức độ protein của các tiểu đơn vị PRC2, H3K27me3 hoặc H3K27ac (Hình bổ sung. 10d–g). Tiếp theo, chúng tôi đã thực hiện CUT&RUN để định hình SUZ12 và EZH2 trong snip1Không-KO và điều khiển NPC (Hình bổ sung. 10h–j). Phù hợp với các kết quả đồng kích thích miễn dịch, SUZ12 và EZH2 cùng chiếm giữ các trang web mục tiêu liên kết với SNIP1 trên toàn bộ bộ gen (Hình XNUMX). 5e). Để phân tích các tương tác PRC2–SNIP1 trên chất nhiễm sắc, chúng tôi đã thực hiện CUT&RUN-reChIP. Trong xét nghiệm này, chất nhiễm sắc do SNIP1 CUT&RUN giải phóng đã bị ức chế miễn dịch bởi IgG, EZH2 hoặc H3K27me3. Các locus đại diện mcm7, Aen, Lhx8và Eomes có đồng sở hữu SNIP1 với EZH2 và H3K27me3 nhưng không kiểm soát âm tính IgG (Hình. 5f). Bộ gen rộng, CUT&RUN-reChIP cho thấy EZH2 và H3K27me3 có độ trùng lặp cao với SNIP1 và PRC2 ở các đỉnh giới hạn SNIP1 (Hình XNUMX). 5e).
Khi kiểm tra vai trò của SNIP1 đối với sự chiếm chỗ của chất nhiễm sắc PRC2, chúng tôi thấy rằng mức SUZ12, EZH2 và H3K27me3 trong chất nhiễm sắc đã giảm đáng kể trong snip1Không-KO (Hình. 5g, Hình bổ sung. 10k, 11a). Ngược lại, mức H3K27ac ít bị thay đổi hơn trong snip1Không-KO NPC (Hình. 5g, Hình bổ sung. 10k, 11a). Tiếp theo, chúng tôi đã sử dụng một thử nghiệm trong ống nghiệm để phân tích tác động động học của sự cạn kiệt SNIP1 trên PRC2. So với lentivirus đối chứng, sự tải nạp Cre của lentivirus của snip1[flox/flox] Các NPC đã làm cạn kiệt bảng điểm SNIP1 lần lượt là 70% và 99.9% vào ngày thứ hai và thứ ba. Điều này không làm thay đổi mức độ phiên mã của các thành phần PRC2 (Hình bổ sung. 11b). Sử dụng EZH2 và SUZ12 CUT&RUN, chúng tôi đã quan sát thấy PRC2 giảm mạnh trên chất nhiễm sắc vào ngày thứ ba khi SNIP1 cạn kiệt (Hình bổ sung. 11c). Cùng với nhau, những dữ liệu này hỗ trợ rằng PRC2 yêu cầu SNIP1 để liên kết với chất nhiễm sắc.
Để mô tả các mục tiêu SNIP1-PRC2, chúng tôi đã thực hiện khám phá mô-đun de novo bằng phần mềm HOMER55. Sử dụng các tiêu chí thay đổi lần> 2 và p < 0.05 trong kiểm soát so với snip1Không-KO, các mục tiêu SNIP1 đã được làm phong phú thêm trong các mô típ của protein E2F, SP1 / 3/4 và EGR1 (Hình XNUMX). 5h). Các mô-típ của các trình tương tác SNIP1, protein SMAD và RELA đã được báo cáo trước đó cũng được tìm thấy trong số các mục tiêu SNIP121,26. Trong các đỉnh giới hạn SUZ12 đã giảm liên kết trong snip1Không-KO, các họa tiết đã được làm phong phú với các protein SP2, RELA, E2F, EGR1, HINFP, PLAGL1 và các tiểu đơn vị NF-Y (Hình XNUMX). 5i). Sự giống nhau của các họa tiết liên kết với SNIP1 và SUZ12 chỉ ra các tương tác tiềm năng của SNIP1 và PRC2 với một số yếu tố phiên mã này.
Tiếp theo, chúng tôi đã kiểm tra xem các họa tiết gắn với SNIP1- hoặc SUZ12 có được biểu hiện quá mức trong các gen được biểu hiện khác nhau hay không. Sử dụng Enrichr56, chúng tôi thấy rằng các gen được điều hòa trong snip1Không-KO là mục tiêu của TP53, FLI1, SUZ12, MAX và MYC, trong khi các gen bị điều hòa giảm là mục tiêu của E2F4, SOX2, NFYB và NFYA (Hình. 5j, k). Các protein E2F đã được phát hiện bằng các phân tích mô-đun và Enrichr và các mục tiêu E2F4 mcm7 và anp32e là các mục tiêu SNIP1 và PRC2 đã giảm ràng buộc trong snip1Không-KO (Hình. 5k, Hình bổ sung. 11d). Trong số các gen điều hòa ở snip1KhôngCác mục tiêu -KO, MYC và MAX được xác định bằng cả phân tích mô-đun và Enrichr (Hình. 5j, Hình bổ sung. 11e). Những dữ liệu này cho thấy rằng các protein E2F, MYC và MAX có thể ảnh hưởng đến các hoạt động SNIP1-PRC2 để điều hòa gen.
PRC2 thúc đẩy quá trình chết theo chương trình khi không có SNIP1
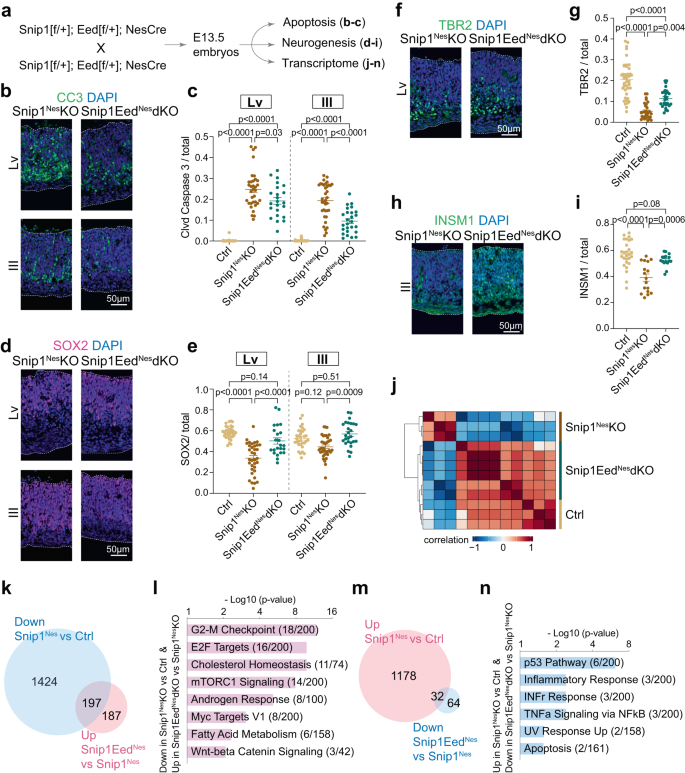
Chúng tôi đã thử nghiệm vai trò của PRC2 trong sự sống sót của NPC in vivo. Chúng tôi đã sử dụng Không::Cre để loại bỏ exon 3 đến 6 của Eed (một tiểu đơn vị lõi PRC2) để tạo EedKhông-KO và snip1Không-EedKhông-dKO (Hình bổ sung. 12a, b). Sự suy giảm EED trong NPC không gây ra quá trình chết theo chương trình trong EedKhông-KO E13.5 não (Hình bổ sung. 12c–g). Những người khác đã nghiên cứu EED trong kiểm soát và EedEmx1-KO vây lưng telencephalon và đưa ra kết luận57 khác với những quan sát của chúng tôi trong EedKhông-KO não E13.5. Sự khác biệt này có thể được giải thích bằng biểu hiện của Emx1::Cre và Không::Cre ở các giai đoạn phát triển và vùng não khác nhau. Apoptosis đã được quan sát thấy ở các vùng não kiểm soát và EedKhông-KO não (Hình bổ sung. 12c, d). Vì EED đã được chứng minh là có ảnh hưởng đến sự hình thành thần kinh ở E14.5 chứ không phải E16.557, tác dụng chính xác của EED/PRC2 đối với các chức năng của NPC phụ thuộc vào giai đoạn phát triển và bối cảnh tế bào. Phân tích của chúng tôi cho thấy rằng so với snip1Không–KO, snip1Không-EedKhông-dKO có số lượng tế bào 3 dương tính với cl-caspase ít hơn đáng kể (Hình. 6a c), nhiều NPC dương tính với SOX2 hơn (Hình. 6d, e) và nhiều tổ tiên trung gian dương tính với TBR2- hoặc INSM1 hơn (Hình. 6f–i). Các tế bào thần kinh chưa trưởng thành dương tính với TUJ1 không bị ảnh hưởng rõ rệt bởi tương tác chức năng SNIP1THER PRC2 (Hình bổ sung. 12h, tôi). EED cạn kiệt trong snip1Không–Não phôi KO làm giảm quá trình tự hủy và giải cứu các NPC và tổ tiên trung gian. Do đó, SNIP1 ngăn chặn quá trình chết theo chương trình ở não đang phát triển bằng cách chống lại PRC2.
a Sơ đồ của phép lai di truyền làm cạn kiệt cả SNIP1 và EED đối với các thử nghiệm xuôi dòng. b, c NẾU phân tích CC3 được phủ bằng DAPI của bộ não E13.5. Thanh, 50 m. Mỗi điểm dữ liệu đại diện cho một hình ảnh. Tám phôi đối chứng, 7 snip1Không-KO phôi, còn 5 snip1Không-EedKhông-dKO phôi đã được phân tích. Đối với tâm thất bên, n = 38 hình ảnh (điều khiển), n = 34 (snip1Không-KO), và n = 23 (snip1Không-EedKhông-dKO). Đối với tâm thất thứ ba, n = 38 (kiểm soát), n = 36 (snip1Không-KO), và n = 26 (snip1Không-EedKhông-dKO). d–tôi NẾU của NPC đánh dấu SOX2 và các dấu hiệu tổ tiên trung gian TBR2 và INSM1 được phủ bằng DAPI của bộ não E13.5. Thanh, 50 mm. Dân số của (e) SOX2 dương tính, (g) TBR2 dương tính, hoặc (i) Các tế bào dương tính với INSM1 trong lớp biểu mô thần kinh của tâm thất bên và/hoặc tâm thất thứ ba đã được định lượng. Mỗi điểm dữ liệu đại diện cho một hình ảnh. TRONG (g), n = 43 hình ảnh (điều khiển), n = 30 (snip1Không-KO), và n = 29 (snip1Không-EedKhông-dKO). TRONG (i), n = 27 (kiểm soát), n = 17 (snip1Không-KO), và n = 16 (snip1Không-EedKhông-dKO). cho bảng c, e, gvà i, dữ liệu được trình bày dưới dạng trung bình ± SEM và ANOVA hai chiều được sử dụng để phân tích thống kê. j Phân cụm dữ liệu RNA-seq không được giám sát từ điều khiển (n = 3), snip1Không-KO (n = 3), và snip1Không-EedKhông-dKO (n = 6) não ở E13.5. Các RNA từ các vùng não trước và não giữa đã được giải trình tự và hợp nhất để phân tích xuôi dòng. Màu xanh biểu thị mối tương quan âm và màu đỏ biểu thị mối tương quan dương. k, m Biểu đồ Venn hiển thị số lượng gen biểu hiện khác nhau với FDR <0.05. Danh sách các gen điều hòa giảm trong snip1Không-KO so với các gen kiểm soát và điều hòa trong snip1Không-EedKhông-dKO vs. snip1Không-KO được so sánh trong (k). Danh sách các gen được điều chỉnh tăng trong snip1Không-KO so với các gen kiểm soát và điều hòa giảm trong snip1Không-EedKhông-dKO vs. snip1Không-KO được so sánh trong (m). l, n Bản thể gen của các gen được giải cứu tương ứng với hình. 6k, m. Các gen được tìm kiếm dựa trên Cơ sở dữ liệu chữ ký phân tử (MSigDB) Hallmark 2020 bằng Enrichr56. Dữ liệu nguồn được cung cấp trong tệp Dữ liệu nguồn (c, e, g, i, l, n).
Chúng tôi lập hồ sơ các bản phiên mã kiểm soát, snip1Không–KHÔNG, và snip1Không-EedKhông-dKO mô não. Phân cụm không được giám sát dựa trên 3,000 gen được biểu hiện khác biệt nhất (dựa trên các giá trị biến thể trung bình) cho thấy mức độ tương đồng cao hơn trong các bản phiên mã giữa đối chứng và snip1Không-EedKhông-dKO so với snip1Không–KO (Hình. 6j). Sử dụng thay đổi lần >2 và p < 0.05 để so sánh các bộ dữ liệu từ snip1Không-KO và snip1Không-EedKhông-dKO, chúng tôi đã xác định được 184 gen điều hòa ngược và 994 gen điều hòa giảm trong snip1Không-EedKhông-dKO (Hình bổ sung. 13a).
Để cung cấp một lời giải thích phân tử tiềm năng về việc giải cứu các NPC và tổ tiên trung gian trong snip1Không-EedKhông-dKO, chúng tôi đã phân tích các gen biểu hiện khác nhau giữa các đối chứng, snip1Không-EedKhông-dKO, và snip1Không-KO. Chúng tôi thấy rằng 197 gen điều hòa giảm trong snip1Không–KO lấy lại một phần biểu hiện trong snip1Không-EedKhông-dKO (Hình. 6k). Những gen được giải cứu này có liên quan đến điểm kiểm tra G2/M, mục tiêu E2F, cân bằng nội môi cholesterol, tín hiệu mTORC1 và phản ứng androgen (Hình. 6l). Ngược lại, 32 gen được điều hòa trong snip1Không-KO trở thành downregulated trong snip1Không-EedKhông-KO (Sung. 6m) và có liên quan đến con đường p53, phản ứng gamma gây viêm và interferon, tín hiệu NFκB và quá trình chết theo chương trình (Hình. 6n). Trong số 32 gen này, Cdkn1a là gen gây chết tế bào theo chương trình nội tại duy nhất có mức H3K27me3 thấp hơn đáng kể trong snip1Không-KO so với kiểm soát (FDR <0.05, XNUMX; Hình bổ sung. 13b), gợi ý rằng SNIP1 trực tiếp quảng bá PRC2 và H3K27me3 tại Cdkn1a quỹ tích. Hồ sơ của chúng tôi về tỷ lệ chiếm bộ gen SNIP1-PRC2 cho thấy rằng SNIP1 ức chế sự lắng đọng H3K27me3 tại các locus bao gồm các mục tiêu E2F (Hình bổ sung. 11d) nhưng thúc đẩy sự lắng đọng H3K27me3 tại các locus khác bao gồm các gen apoptotic Cdkn1a, Nkx2-9, Etv4, Pxdc1và Tap1 (Hình bổ sung. 13b, c). Được kết hợp với nhau, SNIP1 thực hiện kiểm soát phụ thuộc vào cục bộ đối với sự lắng đọng H3K27me3 và biểu hiện gen để cân bằng quá trình phân chia NPC, quá trình chết theo chương trình và sự khác biệt trong não đang phát triển.
- Phân phối nội dung và PR được hỗ trợ bởi SEO. Được khuếch đại ngay hôm nay.
- PlatoData.Network Vertical Generative Ai. Trao quyền cho chính mình. Truy cập Tại đây.
- PlatoAiStream. Thông minh Web3. Kiến thức khuếch đại. Truy cập Tại đây.
- Trung tâmESG. Ô tô / Xe điện, Than đá, công nghệ sạch, Năng lượng, Môi trường Hệ mặt trời, Quản lý chất thải. Truy cập Tại đây.
- BlockOffsets. Hiện đại hóa quyền sở hữu bù đắp môi trường. Truy cập Tại đây.
- nguồn: https://www.nature.com/articles/s41467-023-40487-4