이 게시물에서는 최첨단 단백질 언어 모델(pLM)을 효율적으로 미세 조정하여 단백질 세포내 위치를 예측하는 방법을 보여줍니다. 아마존 세이지 메이커.
단백질은 근육을 움직이는 것부터 감염에 반응하는 것까지 모든 것을 담당하는 신체의 분자 기계입니다. 이러한 다양성에도 불구하고 모든 단백질은 아미노산이라고 불리는 반복되는 분자 사슬로 구성됩니다. 인간 게놈은 20개의 표준 아미노산을 암호화하며, 각각은 약간 다른 화학 구조를 가지고 있습니다. 이는 알파벳 문자로 표시될 수 있으며 이를 통해 단백질을 텍스트 문자열로 분석하고 탐색할 수 있습니다. 단백질 서열과 구조의 엄청난 수는 단백질의 다양한 용도를 제공합니다.
단백질은 또한 잠재적 표적으로서 뿐만 아니라 치료제로서 약물 개발에서 중요한 역할을 합니다. 다음 표에서 볼 수 있듯이 2022년에 가장 많이 팔린 약물 중 다수는 단백질(특히 항체)이거나 체내에서 단백질로 번역되는 mRNA와 같은 다른 분자였습니다. 이 때문에 많은 생명과학 연구자들은 단백질에 관한 질문에 더 빠르고, 저렴하고, 정확하게 답해야 합니다.
| 성함 | 제조업 자 | 2022년 글로벌 매출($ 수십억 달러) | 표시 |
| 커미르나티 | 화이자 / 바이오텍 | $40.8 | Covid-19 |
| 스파이크백스 | 현대 | $21.8 | Covid-19 |
| 휴미라 | AbbVie | $21.6 | 관절염, 크론병 등 |
| Keytruda | 머크 | $21.0 | 다양한 암 |
데이터 출처: Urquhart, L. 2022년 매출 기준 상위 기업 및 의약품. Nature Reviews Drug Discovery 22, 260–260 (2023).
우리는 단백질을 문자의 순서로 나타낼 수 있기 때문에 원래 문자 언어용으로 개발된 기술을 사용하여 단백질을 분석할 수 있습니다. 여기에는 대규모 데이터 세트에 대해 사전 훈련된 LLM(대형 언어 모델)이 포함되며, 이를 텍스트 요약이나 챗봇과 같은 특정 작업에 맞게 조정할 수 있습니다. 마찬가지로, pLM은 레이블이 지정되지 않은 자기 지도 학습을 사용하여 대규모 단백질 서열 데이터베이스에서 사전 훈련됩니다. 우리는 이를 적용하여 단백질의 3D 구조나 다른 분자와 상호 작용하는 방식 등을 예측할 수 있습니다. 연구자들은 pLM을 사용하여 처음부터 새로운 단백질을 디자인하기도 했습니다. 이러한 도구는 인간의 과학적 전문 지식을 대체하지는 않지만 전임상 개발 및 시험 설계 속도를 높일 수 있는 잠재력을 가지고 있습니다.
이 모델의 한 가지 과제는 크기입니다. 다음 그림에서 볼 수 있듯이 LLM과 pLM 모두 지난 몇 년 동안 엄청난 규모로 성장했습니다. 이는 충분한 정확도로 훈련하는 데 오랜 시간이 걸릴 수 있음을 의미합니다. 이는 또한 모델 매개변수를 저장하기 위해 대용량 메모리를 갖춘 하드웨어, 특히 GPU를 사용해야 함을 의미합니다.
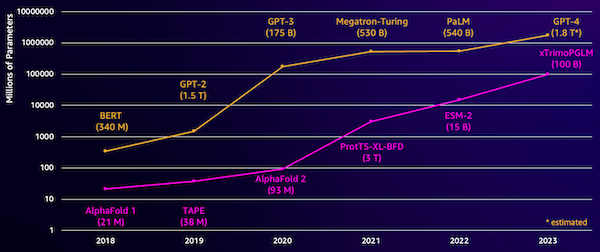
긴 훈련 시간과 대규모 인스턴스는 높은 비용을 의미하므로 많은 연구자가 이 작업을 수행하기 어려울 수 있습니다. 예를 들어, 2023년에는 연구팀 100일 동안 768개의 A100 GPU에서 164억 개의 매개변수 pLM을 훈련하는 방법을 설명했습니다! 다행히도 많은 경우 기존 pLM을 특정 작업에 맞게 조정하여 시간과 리소스를 절약할 수 있습니다. 이 기술을 미세 조정, 또한 다른 유형의 언어 모델링에서 고급 도구를 빌릴 수 있습니다.
솔루션 개요
이 게시물에서 다루는 구체적인 문제는 다음과 같습니다. 세포하 위치화: 단백질 서열이 주어지면 그것이 외부(세포막)에 있는지, 세포 내부에 있는지 예측할 수 있는 모델을 구축할 수 있나요? 이는 기능을 이해하고 좋은 약물 표적이 될 수 있는지 여부를 이해하는 데 도움이 될 수 있는 중요한 정보입니다.
다음을 사용하여 공개 데이터 세트를 다운로드하는 것부터 시작합니다. 아마존 세이지 메이커 스튜디오. 그런 다음 SageMaker를 사용하여 효율적인 훈련 방법을 사용하여 ESM-2 단백질 언어 모델을 미세 조정합니다. 마지막으로 모델을 실시간 추론 엔드포인트로 배포하고 이를 사용하여 일부 알려진 단백질을 테스트합니다. 다음 다이어그램은 이 워크플로를 보여줍니다.

다음 섹션에서는 교육 데이터를 준비하고, 교육 스크립트를 생성하고, SageMaker 교육 작업을 실행하는 단계를 안내합니다. 이 게시물에 소개된 모든 코드는 다음에서 사용할 수 있습니다. GitHub의.
훈련 데이터 준비
우리는 DeepLoc-2 데이터세트이는 실험적으로 결정된 위치를 가진 수천 개의 SwissProt 단백질을 포함합니다. 100~512개 아미노산 사이의 고품질 시퀀스를 필터링합니다.
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
다음으로 시퀀스를 토큰화하고 이를 훈련 및 평가 세트로 나눕니다.
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
마지막으로 처리된 훈련 및 평가 데이터를 업로드합니다. 아마존 단순 스토리지 서비스 (Amazon S3) :
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)학습 스크립트 만들기
SageMaker 스크립트 모드 AWS에서 관리하는 최적화된 기계 학습(ML) 프레임워크 컨테이너에서 사용자 지정 훈련 코드를 실행할 수 있습니다. 이 예에서는 다음을 적용합니다. 텍스트 분류를 위한 기존 스크립트 허깅 페이스에서. 이를 통해 훈련 작업의 효율성을 향상시키기 위한 여러 가지 방법을 시도할 수 있습니다.
방법 1: 가중치가 부여된 교육 수업
많은 생물학적 데이터 세트와 마찬가지로 DeepLoc 데이터는 고르지 않게 분포되어 있습니다. 즉, 막 단백질과 비막 단백질의 수가 동일하지 않습니다. 데이터를 다시 샘플링하고 다수 클래스의 레코드를 삭제할 수 있습니다. 그러나 이렇게 하면 전체 훈련 데이터가 줄어들고 잠재적으로 정확도가 저하될 수 있습니다. 대신 훈련 작업 중에 클래스 가중치를 계산하고 이를 사용하여 손실을 조정합니다.
훈련 스크립트에서 우리는 Trainer 수업에서 transformers 와 WeightedTrainer 교차 엔트로피 손실을 계산할 때 클래스 가중치를 고려하는 클래스입니다. 이는 모델의 편향을 방지하는 데 도움이 됩니다.
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else loss방법 2: 그라데이션 누적
경사 누적은 모델이 더 큰 배치 크기에 대한 훈련을 시뮬레이션할 수 있게 해주는 훈련 기술입니다. 일반적으로 배치 크기(한 훈련 단계에서 기울기를 계산하는 데 사용되는 샘플 수)는 GPU 메모리 용량에 의해 제한됩니다. 기울기 누적을 사용하면 모델은 더 작은 배치의 기울기를 먼저 계산합니다. 그런 다음 모델 가중치를 즉시 업데이트하는 대신 여러 개의 작은 배치에 걸쳐 그래디언트가 누적됩니다. 누적된 기울기가 더 큰 목표 배치 크기와 같을 때 모델을 업데이트하기 위해 최적화 단계가 수행됩니다. 이를 통해 모델은 GPU 메모리 제한을 초과하지 않고 효과적으로 더 큰 배치로 학습할 수 있습니다.
그러나 더 작은 배치 정방향 및 역방향 전달에는 추가 계산이 필요합니다. 경사 누적을 통해 배치 크기가 증가하면 특히 너무 많은 누적 단계가 사용되는 경우 훈련 속도가 느려질 수 있습니다. 목표는 GPU 사용량을 최대화하되 너무 많은 추가 경사 계산 단계로 인한 과도한 속도 저하를 방지하는 것입니다.
방법 3: 그라데이션 체크포인트
그라데이션 체크포인트는 계산 시간을 합리적으로 유지하면서 훈련 중에 필요한 메모리를 줄이는 기술입니다. 대규모 신경망은 역방향 패스 동안 기울기를 계산하기 위해 순방향 패스의 모든 중간 값을 저장해야 하기 때문에 많은 메모리를 차지합니다. 이로 인해 메모리 문제가 발생할 수 있습니다. 한 가지 해결책은 이러한 중간 값을 저장하지 않는 것입니다. 그러나 역방향 전달 중에 다시 계산해야 하므로 시간이 많이 걸립니다.
그라데이션 체크포인트는 균형 잡힌 접근 방식을 제공합니다. 중간 값 중 일부만 저장합니다. 체크 포인트, 필요에 따라 다른 항목을 다시 계산합니다. 따라서 모든 것을 저장하는 것보다 메모리를 덜 사용하지만 모든 것을 다시 계산하는 것보다 계산도 더 적습니다. 체크포인트할 활성화를 전략적으로 선택함으로써 그래디언트 체크포인트를 통해 대규모 신경망을 관리 가능한 메모리 사용량과 계산 시간으로 훈련할 수 있습니다. 이 중요한 기술을 사용하면 메모리 제한에 직면할 수 있는 매우 큰 모델을 훈련하는 것이 가능해집니다.
훈련 스크립트에서는 필요한 매개변수를 TrainingArguments 목적:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)방법 4: LLM의 낮은 순위 적응
ESM-2와 같은 대규모 언어 모델에는 훈련 및 실행 비용이 많이 드는 수십억 개의 매개변수가 포함될 수 있습니다. 연구원 이러한 거대한 모델을 보다 효율적으로 미세 조정하기 위해 LoRA(Low-Rank Adaptation)라는 훈련 방법을 개발했습니다.
LoRA의 핵심 아이디어는 특정 작업을 위해 모델을 미세 조정할 때 원래 매개변수를 모두 업데이트할 필요가 없다는 것입니다. 대신 LoRA는 입력과 출력을 변환하는 모델에 더 작은 새로운 행렬을 추가합니다. 미세 조정 중에는 이러한 작은 행렬만 업데이트되므로 속도가 훨씬 빠르고 메모리도 덜 사용됩니다. 원래 모델 매개변수는 고정된 상태로 유지됩니다.
LoRA로 미세 조정한 후 작은 적응 행렬을 다시 원래 모델에 병합할 수 있습니다. 또는 이전 작업을 잊지 않고 다른 작업을 위해 모델을 신속하게 미세 조정하려는 경우 별도로 보관할 수 있습니다. 전반적으로 LoRA를 사용하면 LLM이 일반적인 비용의 일부만으로 새로운 작업에 효율적으로 적응할 수 있습니다.
훈련 스크립트에서는 다음을 사용하여 LoRA를 구성합니다. PEFT Hugging Face의 라이브러리:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)SageMaker 훈련 작업 제출
훈련 스크립트를 정의한 후 SageMaker 훈련 작업을 구성하고 제출할 수 있습니다. 먼저 하이퍼파라미터를 지정합니다.
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}다음으로 훈련 로그에서 캡처할 측정항목을 정의합니다.
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]마지막으로 Hugging Face 추정기를 정의하고 ml.g5.2xlarge 인스턴스 유형에 대한 교육을 위해 제출합니다. 이는 많은 AWS 리전에서 널리 사용할 수 있는 비용 효율적인 인스턴스 유형입니다.
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)다음 표에서는 논의한 다양한 훈련 방법과 해당 방법이 작업의 런타임, 정확도 및 GPU 메모리 요구 사항에 미치는 영향을 비교합니다.
| 구성 | 청구 가능 시간(분) | 평가 정확도 | 최대 GPU 메모리 사용량(GB) |
| 기본 모델 | 28 | 0.91 | 22.6 |
| 기본 + GA | 21 | 0.90 | 17.8 |
| 베이스 + GC | 29 | 0.91 | 10.2 |
| 베이스 + LoRA | 23 | 0.90 | 18.6 |
모든 방법은 평가 정확도가 높은 모델을 생성했습니다. LoRA와 그래디언트 활성화를 사용하면 실행 시간(및 비용)이 각각 18%와 25% 감소했습니다. 그래디언트 체크포인트를 사용하면 최대 GPU 메모리 사용량이 55% 감소했습니다. 제약 조건(비용, 시간, 하드웨어)에 따라 이러한 접근 방식 중 하나가 다른 접근 방식보다 더 합리적일 수 있습니다.
이러한 각 방법은 그 자체로는 잘 수행되지만, 이를 조합하여 사용하면 어떻게 될까요? 다음 표에는 결과가 요약되어 있습니다.
| 구성 | 청구 가능 시간(분) | 평가 정확도 | 최대 GPU 메모리 사용량(GB) |
| 모든 방법 | 12 | 0.80 | 3.3 |
이 경우 정확도가 12% 감소합니다. 그러나 런타임은 57%, GPU 메모리 사용량은 85% 줄였습니다! 이는 비용 효율적인 다양한 인스턴스 유형에 대해 교육할 수 있게 해주는 엄청난 감소입니다.
정리
자신의 AWS 계정을 사용하는 경우 추가 비용이 발생하지 않도록 생성한 실시간 추론 엔드포인트와 데이터를 모두 삭제하세요.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()결론
이 게시물에서는 과학적으로 관련된 작업을 위해 ESM-2와 같은 단백질 언어 모델을 효율적으로 미세 조정하는 방법을 시연했습니다. Transformers 및 PEFT 라이브러리를 사용하여 pLMS를 교육하는 방법에 대한 자세한 내용은 게시물을 확인하세요. 단백질을 이용한 딥러닝 과 ESMBind(ESMB): 단백질 결합 부위 예측을 위한 ESM-2의 낮은 순위 적응 Hugging Face 블로그에서. 또한 머신러닝을 사용하여 단백질 특성을 예측하는 더 많은 예를 다음에서 찾아볼 수 있습니다. AWS의 놀라운 단백질 분석 GitHub 저장소.
저자에 관하여
 브라이언 로열 Amazon Web Services 글로벌 의료 및 생명 과학 팀의 수석 AI/ML 솔루션 설계자입니다. 그는 생명 공학 및 기계 학습 분야에서 17년 이상의 경력을 보유하고 있으며 고객이 게놈 및 단백질체 문제를 해결하도록 돕는 데 열정적입니다. 여가 시간에는 친구 및 가족과 함께 요리하고 식사하는 것을 즐깁니다.
브라이언 로열 Amazon Web Services 글로벌 의료 및 생명 과학 팀의 수석 AI/ML 솔루션 설계자입니다. 그는 생명 공학 및 기계 학습 분야에서 17년 이상의 경력을 보유하고 있으며 고객이 게놈 및 단백질체 문제를 해결하도록 돕는 데 열정적입니다. 여가 시간에는 친구 및 가족과 함께 요리하고 식사하는 것을 즐깁니다.
- SEO 기반 콘텐츠 및 PR 배포. 오늘 증폭하십시오.
- PlatoData.Network 수직 생성 Ai. 자신에게 권한을 부여하십시오. 여기에서 액세스하십시오.
- PlatoAiStream. 웹3 인텔리전스. 지식 증폭. 여기에서 액세스하십시오.
- 플라톤ESG. 탄소, 클린테크, 에너지, 환경, 태양광, 폐기물 관리. 여기에서 액세스하십시오.
- PlatoHealth. 생명 공학 및 임상 시험 인텔리전스. 여기에서 액세스하십시오.
- 출처: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



