Dies ist ein Gastbeitrag, der gemeinsam von Ajay K Gupta, Jean Felipe Teotonio und Paul A Churchyard von HSR.health verfasst wurde.
HSR.Gesundheit ist ein Geodatenunternehmen für Gesundheitsrisikoanalysen, dessen Vision darin besteht, dass globale Gesundheitsherausforderungen durch menschlichen Einfallsreichtum und die gezielte und genaue Anwendung von Datenanalysen lösbar sind. In diesem Beitrag stellen wir einen Ansatz zur Zoonoseprävention vor, der Folgendes nutzt: Geodatenfunktionen von Amazon SageMaker um ein Tool zu entwickeln, das Gesundheitswissenschaftlern genauere Informationen zur Krankheitsausbreitung liefert, damit sie schneller mehr Leben retten können.
Zoonosekrankheiten betreffen sowohl Tiere als auch Menschen. Der Übergang einer Krankheit vom Tier auf den Menschen, bekannt als Spillover, ist ein Phänomen, das auf unserem Planeten immer wieder auftritt. Laut Gesundheitsorganisationen wie den Centers for Disease Control and Prevention (CDC ) und der Weltgesundheitsorganisation (WHO), ein Spillover-Ereignis auf einem Nassmarkt in Wuhan, China, verursachte höchstwahrscheinlich die Coronavirus-Krankheit 2019 (COVID-19). Studien deuten darauf hin, dass ein in Flughunden vorkommendes Virus erhebliche Mutationen durchgemacht hat, die es ihm ermöglichen, Menschen zu infizieren. Der erste Patient oder „Patient Null“ für COVID-19 löste wahrscheinlich einen nachfolgenden lokalen Ausbruch aus, der sich schließlich international ausbreitete. HSR.der Knochen und des BewegungsapparatesDer Zoonotic Spillover Risk Index soll bei der Identifizierung dieser frühen Ausbrüche helfen, bevor sie internationale Grenzen überschreiten und weitreichende globale Auswirkungen haben.
Die wichtigste Waffe der öffentlichen Gesundheit gegen die Ausbreitung regionaler Ausbrüche ist die Krankheitsüberwachung: ein komplettes ineinandergreifendes System der Krankheitsmeldung, Untersuchung und Datenkommunikation zwischen verschiedenen Ebenen eines öffentlichen Gesundheitssystems. Dieses System ist nicht nur von menschlichen Faktoren abhängig, sondern auch von Technologie und Ressourcen, um Krankheitsdaten zu sammeln, Muster zu analysieren und einen konsistenten und kontinuierlichen Datenfluss von lokalen über regionale bis hin zu zentralen Gesundheitsbehörden zu schaffen.
Die Geschwindigkeit, mit der sich COVID-19 von einem lokalen Ausbruch zu einer globalen Krankheit entwickelte, die auf jedem einzelnen Kontinent auftritt, sollte ein ernüchterndes Beispiel für die dringende Notwendigkeit sein, innovative Technologien zu nutzen, um effizientere und genauere Krankheitsüberwachungssysteme zu schaffen.
Das Risiko einer Ausbreitung zoonotischer Krankheiten steht in engem Zusammenhang mit mehreren sozialen, ökologischen und geografischen Faktoren, die Einfluss darauf haben, wie oft Menschen mit Wildtieren interagieren. HSR.Gesundheit Der Zoonose-Spillover-Risikoindex verwendet über 20 verschiedene geografische, soziale und umweltbedingte Faktoren, von denen in der Vergangenheit bekannt ist, dass sie das Risiko der Mensch-Tier-Interaktion und damit das Risiko einer Zoonose-Spillover-Risiko beeinflussen. Viele dieser Faktoren können durch eine Kombination aus Satellitenbildern und Fernerkundung kartiert werden.
In diesem Beitrag untersuchen wir, wie HSR.der Knochen und des Bewegungsapparates nutzt die Geodatenfunktionen von SageMaker, um relevante Merkmale aus Satellitenbildern und Fernerkundungen abzurufen und so den Risikoindex zu entwickeln. Die Geodatenfunktionen von SageMaker erleichtern Datenwissenschaftlern und Ingenieuren für maschinelles Lernen (ML) das Erstellen, Trainieren und Bereitstellen von Modellen mithilfe von Geodaten. Mit den Geodatenfunktionen von SageMaker können Sie große Geodatensätze effizient transformieren oder anreichern, die Modellerstellung mit vorab trainierten ML-Modellen beschleunigen und Modellvorhersagen und Geodaten auf einer interaktiven Karte mithilfe beschleunigter 3D-Grafiken und integrierter Visualisierungstools erkunden.
Verwendung von ML und Geodaten zur Risikominderung
ML ist für die Anomalieerkennung bei räumlichen oder zeitlichen Daten äußerst effektiv, da es aus Daten lernen kann, ohne explizit für die Identifizierung bestimmter Arten von Anomalien programmiert zu werden. Raumdaten, die sich auf die physische Position und Form von Objekten beziehen, enthalten häufig komplexe Muster und Beziehungen, die für herkömmliche Algorithmen möglicherweise schwierig zu analysieren sind.
Die Integration von ML mit Geodaten verbessert die Fähigkeit, Anomalien und ungewöhnliche Muster systematisch zu erkennen, was für Frühwarnsysteme von entscheidender Bedeutung ist. Diese Systeme sind in Bereichen wie Umweltüberwachung, Katastrophenmanagement und Sicherheit von entscheidender Bedeutung. Die prädiktive Modellierung unter Verwendung historischer Geodaten ermöglicht es Unternehmen, potenzielle zukünftige Ereignisse zu erkennen und sich darauf vorzubereiten. Diese Ereignisse reichen von Naturkatastrophen und Verkehrsbehinderungen bis hin zu Krankheitsausbrüchen, wie in diesem Beitrag erläutert wird.
Erkennung von Zoonose-Spillover-Risiken
Zur Vorhersage von zoonotischen Spillover-Risiken, HSR.Gesundheit hat einen multimodalen Ansatz gewählt. Durch die Verwendung einer Mischung verschiedener Datentypen – einschließlich Umwelt-, biogeografischer und epidemiologischer Informationen – ermöglicht diese Methode eine umfassende Bewertung der Krankheitsdynamik. Eine solch vielfältige Perspektive ist entscheidend für die Entwicklung proaktiver Maßnahmen und die Ermöglichung einer schnellen Reaktion auf Ausbrüche.
Der Ansatz umfasst folgende Komponenten:
- Daten zu Krankheiten und Ausbrüchen – HSR.Gesundheit nutzt die umfangreichen Krankheits- und Ausbruchsdaten von Gideon und die Weltgesundheitsorganisation (WHO), zwei vertrauenswürdige Quellen globaler epidemiologischer Informationen. Diese Daten dienen als grundlegende Säule im Analyse-Framework. Für Gideon ist der Zugriff auf die Daten über eine API möglich, für die WHO über HSR.Gesundheit hat ein großes Sprachmodell (LLM) entwickelt, um Ausbruchsdaten aus früheren Krankheitsausbruchsberichten zu extrahieren.
- Erdbeobachtungsdaten – Umweltfaktoren, Landnutzungsanalysen und die Erkennung von Lebensraumveränderungen sind integrale Bestandteile der Bewertung des Zoonoserisikos. Diese Erkenntnisse können aus satellitengestützten Erdbeobachtungsdaten abgeleitet werden. HSR.Gesundheit ist in der Lage, die Nutzung von Erdbeobachtungsdaten zu optimieren, indem es die Geodatenfunktionen von SageMaker nutzt, um auf große Geodatensätze zuzugreifen und diese zu bearbeiten. SageMaker Geospatial bietet einen umfangreichen Datenkatalog, einschließlich Datensätzen von USGS Landsat-8, Sentinel-1, Sentinel-2 und anderen. Es ist auch möglich, andere Datensätze einzubinden, beispielsweise hochauflösende Bilder von Planet Labs.
- Soziale Determinanten des Risikos – Über biologische und Umweltfaktoren hinaus, das Team von HSR.Gesundheit Dabei wurden auch soziale Determinanten berücksichtigt, die verschiedene sozioökonomische und demografische Indikatoren umfassen und eine entscheidende Rolle bei der Gestaltung der Zoonose-Spillover-Dynamik spielen.
Aus diesen Komponenten HSR.Gesundheit evaluierte eine Reihe verschiedener Faktoren und die folgenden Merkmale wurden als einflussreich für die Identifizierung von Zoonose-Spillover-Risiken identifiziert:
- Tierlebensräume und bewohnbare Zonen – Das Verständnis der Lebensräume potenzieller zoonotischer Wirte und ihrer bewohnbaren Zonen ist für die Beurteilung des Übertragungsrisikos von grundlegender Bedeutung.
- Bevölkerungszentren – Die Nähe zu dicht besiedelten Gebieten ist ein wichtiger Gesichtspunkt, da sie die Wahrscheinlichkeit von Mensch-Tier-Interaktionen beeinflusst.
- Verlust des Lebensraums – Die Verschlechterung natürlicher Lebensräume, insbesondere durch Abholzung, kann zoonotische Spillover-Ereignisse beschleunigen.
- Mensch-Wildland-Schnittstelle – Gebiete, in denen sich menschliche Siedlungen mit Lebensräumen von Wildtieren überschneiden, sind potenzielle Hotspots für die Übertragung von Zoonose.
- Soziale Merkmale – Sozioökonomische und kulturelle Faktoren können das Zoonosenrisiko und die HSR erheblich beeinflussen.der Knochen und des Bewegungsapparates untersucht diese ebenfalls.
- Merkmale der menschlichen Gesundheit – Der Gesundheitszustand der lokalen menschlichen Bevölkerung ist eine wesentliche Variable, da er die Anfälligkeit und Übertragungsdynamik beeinflusst.
Lösungsüberblick
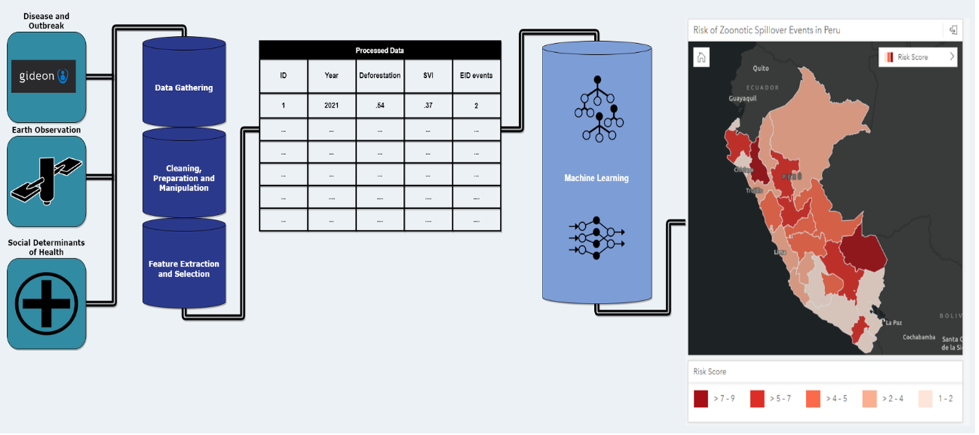
HSR.GesundheitDer Arbeitsablauf von umfasst die Datenvorverarbeitung, die Merkmalsextraktion und die Erstellung informativer Visualisierungen mithilfe von ML-Techniken. Dies ermöglicht ein klares Verständnis der Entwicklung der Daten von ihrer Rohform bis hin zu umsetzbaren Erkenntnissen.
Das Folgende ist eine visuelle Darstellung des Arbeitsablaufs, beginnend mit Eingabedaten von Gideon, Erdbeobachtungsdaten und Daten zur sozialen Determinante des Risikos.
Rufen Sie Satellitenbilder mit den Geodatenfunktionen von SageMaker ab und verarbeiten Sie sie
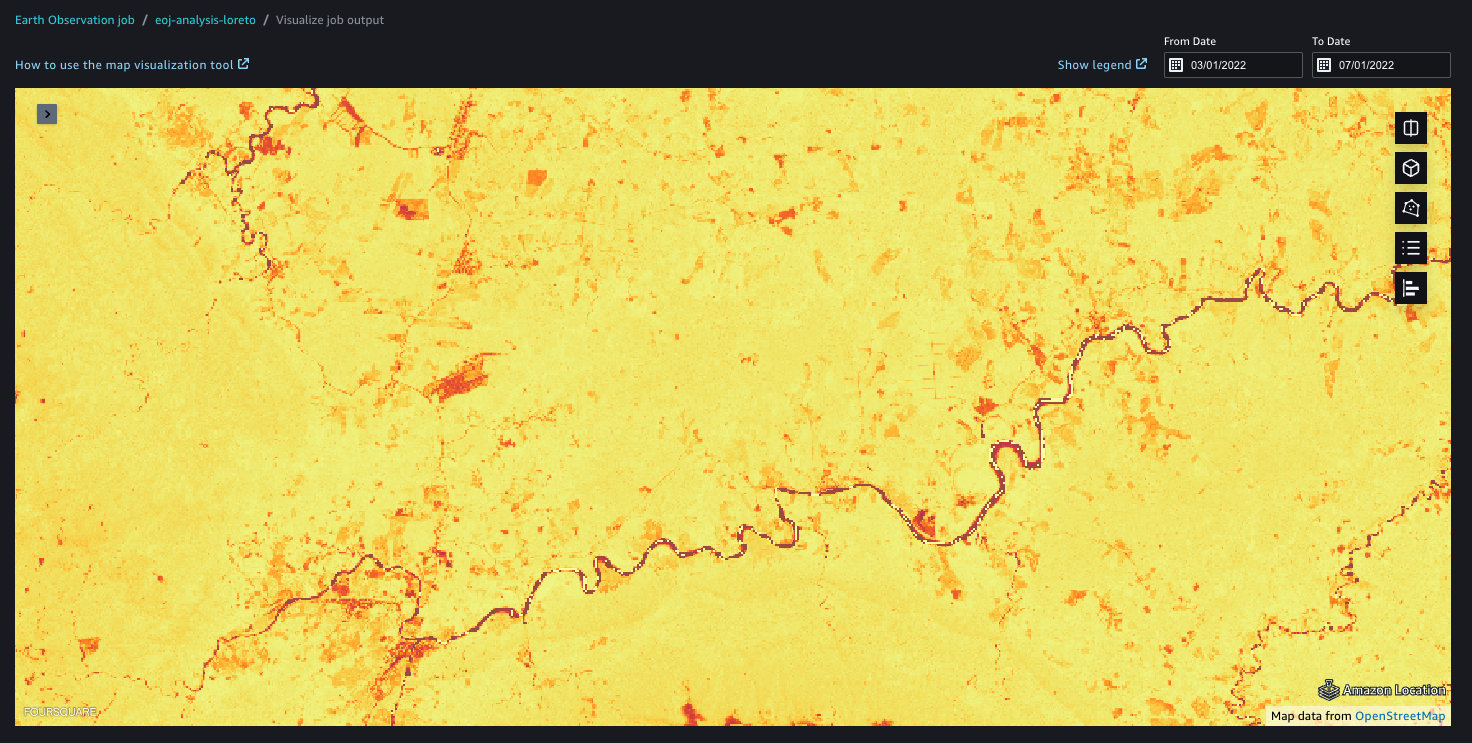
Satellitendaten bilden einen Eckpfeiler der zur Erstellung des Risikoindex durchgeführten Analyse und liefern wichtige Informationen über Umweltveränderungen. Um Erkenntnisse aus Satellitenbildern zu gewinnen, HSR.Gesundheit verwendet Erdbeobachtungsjobs (EOJs). EOJs ermöglichen die Erfassung und Transformation von Rasterdaten, die von der Erdoberfläche gesammelt werden. Ein EOJ erhält Satellitenbilder von einer bestimmten Datenquelle – beispielsweise einer Satellitenkonstellation – über ein bestimmtes Gebiet und einen bestimmten Zeitraum. Anschließend werden ein oder mehrere Modelle auf die abgerufenen Bilder angewendet.
Zusätzlich Amazon SageMaker-Studio bietet ein vorinstalliertes Geodaten-Notizbuch mit häufig verwendeten Geodaten-Bibliotheken. Dieses Notebook ermöglicht die direkte Visualisierung und Verarbeitung von Geodaten in einer Python-Notebook-Umgebung. EOJs können in der Geodaten-Notebook-Umgebung erstellt werden.
Zur Konfiguration eines EOJ werden folgende Parameter verwendet:
- InputConfig – Die Eingabekonfiguration legt die Datenquellen und Filterkriterien fest, die bei der Datenerfassung verwendet werden sollen:
- RasterDataCollectionArn – Gibt den Satelliten an, von dem Daten erfasst werden sollen.
- Interessenbereich – Der geografische Interessenbereich (AOI) definiert die Polygongrenzen für die Bildsammlung.
- Zeitbereichsfilter – Der interessierende Zeitraum:
{StartTime: <string>, EndTime: <string>}. - Eigenschaftsfilter – Zusätzliche Eigenschaftsfilter, wie z. B. akzeptabler Prozentsatz der Wolkenbedeckung oder gewünschte Azimutwinkel der Sonne.
- JobKonfig – Diese Konfiguration definiert die Art des Auftrags, der auf die abgerufenen Satellitenbilddaten angewendet werden soll. Es unterstützt Vorgänge wie Bandmathematik, Resampling, Geomosaik oder Wolkenentfernung.
Der folgende Beispielcode demonstriert die Ausführung eines EOJ zur Cloud-Entfernung, repräsentativ für die von HSR durchgeführten Schritte.Gesundheit:
HSR.Gesundheit verwendete mehrere Vorgänge, um die Daten vorzuverarbeiten und relevante Merkmale zu extrahieren. Dazu gehören Vorgänge wie die Klassifizierung der Landbedeckung, die Kartierung von Temperaturschwankungen und Vegetationsindizes.
Ein für die Angabe der Vegetationsgesundheit relevanter Vegetationsindex ist der Normalized Difference Vegetation Index (NDVI). Der NDVI quantifiziert die Vegetationsgesundheit mithilfe von Nahinfrarotlicht, das von der Vegetation reflektiert wird, und rotem Licht, das von der Vegetation absorbiert wird. Die Überwachung des NDVI im Laufe der Zeit kann Veränderungen in der Vegetation aufdecken, beispielsweise die Auswirkungen menschlicher Aktivitäten wie Abholzung.
Der folgende Codeausschnitt zeigt, wie ein Vegetationsindex wie der NDVI basierend auf den Daten berechnet wird, die durch die Wolkenentfernung übergeben wurden:
Mithilfe der Geodatenfunktionen von SageMaker können wir die Auftragsausgabe visualisieren. Die Geodatenfunktionen von SageMaker können Ihnen dabei helfen, Modellvorhersagen auf einer Basiskarte zu überlagern und eine geschichtete Visualisierung bereitzustellen, um die Zusammenarbeit zu erleichtern. Mit dem GPU-gestützten interaktiven Visualizer und Python-Notebooks ist es möglich, Millionen von Datenpunkten in einer Ansicht zu untersuchen und so die gemeinsame Erforschung von Erkenntnissen und Ergebnissen zu erleichtern.
Die in diesem Beitrag beschriebenen Schritte veranschaulichen nur eine der vielen rasterbasierten Funktionen von HSR.Gesundheit extrahiert hat, um den Risikoindex zu erstellen.
Kombination rasterbasierter Funktionen mit Gesundheits- und Sozialdaten
Nach dem Extrahieren der relevanten Features im Rasterformat wird HSR.Gesundheit nutzten Zonenstatistiken, um die Rasterdaten innerhalb der Verwaltungsgrenzenpolygone zu aggregieren, denen die Sozial- und Gesundheitsdaten zugeordnet sind. Die Analyse umfasst eine Kombination aus Raster- und Vektor-Geodaten. Diese Art der Aggregation ermöglicht die Verwaltung von Rasterdaten in einem Geodatenrahmen, was deren Integration mit den Gesundheits- und Sozialdaten erleichtert, um den endgültigen Risikoindex zu erstellen.
Der folgende Codeausschnitt zeigt, wie Rasterdaten an administrativen Vektorgrenzen aggregiert werden:
Um die extrahierten Merkmale effektiv auszuwerten, werden ML-Modelle verwendet, um Faktoren vorherzusagen, die jedes Merkmal darstellen. Eines der verwendeten Modelle ist eine Support Vector Machine (SVM). Das SVM-Modell hilft dabei, Muster und Zusammenhänge innerhalb von Daten aufzudecken, die als Grundlage für Risikobewertungen dienen.
Der Index stellt eine quantitative Bewertung des Risikoniveaus dar, berechnet als gewichteter Durchschnitt dieser Faktoren, um das Verständnis potenzieller Spillover-Ereignisse in verschiedenen Regionen zu erleichtern.
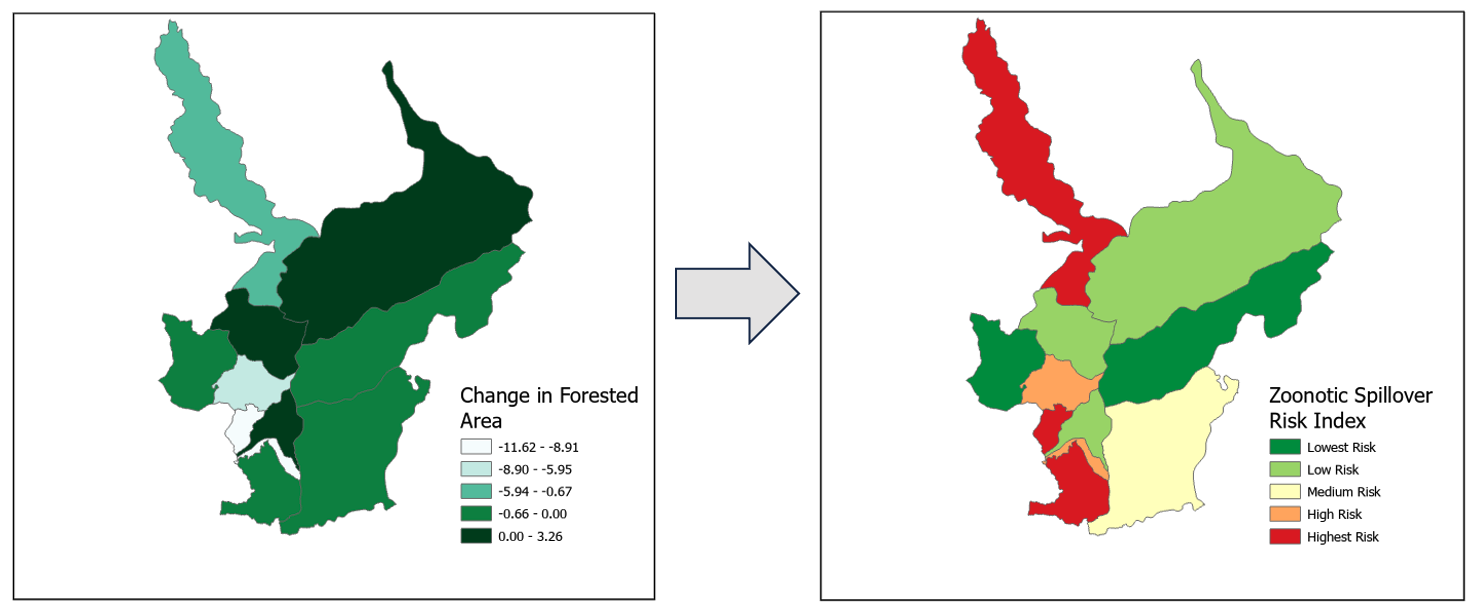
Die folgende Abbildung links zeigt die Aggregation der Bildklassifizierung aus der Testgebietsszene im Norden Perus aggregiert auf die Bezirksverwaltungsebene mit der berechneten Veränderung der Waldfläche zwischen 2018–2023. Die Entwaldung ist einer der Schlüsselfaktoren, die das Risiko einer Ausbreitung von Zoonosen bestimmen. Die Abbildung rechts zeigt den Schweregrad des Zoonose-Spillover-Risikos in den abgedeckten Regionen, vom höchsten (rot) bis zum niedrigsten (dunkelgrün) Risiko. Das Gebiet wurde aufgrund der Vielfalt der in der Szene erfassten Landbedeckung, darunter unter anderem Stadt, Wald, Sand, Wasser, Grasland und Landwirtschaft, als eines der Trainingsgebiete für die Bildklassifizierung ausgewählt. Darüber hinaus ist dies einer von vielen Bereichen, die aufgrund der Entwaldung und der Interaktion zwischen Mensch und Tier für potenzielle zoonotische Spillover-Ereignisse von Interesse sind.
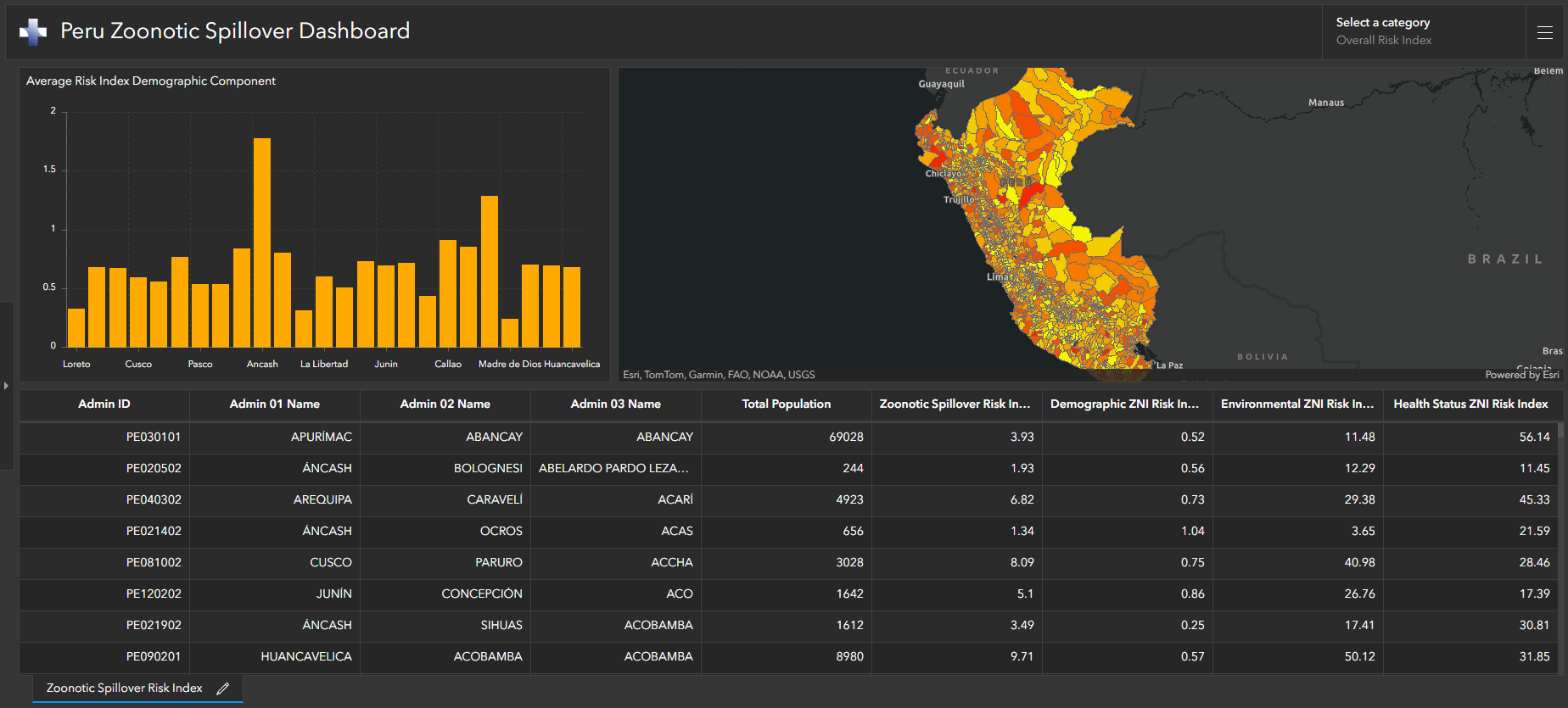
Durch die Übernahme dieses multimodalen Ansatzes, der historische Daten zu Krankheitsausbrüchen, Erdbeobachtungsdaten, soziale Determinanten und ML-Techniken umfasst, können wir das Risiko eines Zoonose-Spillovers besser verstehen und vorhersagen und letztendlich Krankheitsüberwachungs- und Präventionsstrategien auf Bereiche mit dem größten Ausbruchsrisiko ausrichten. Der folgende Screenshot zeigt ein Dashboard der Ergebnisse einer Zoonose-Spillover-Risikoanalyse. Diese Risikoanalyse zeigt, wo Ressourcen und Überwachung für neue potenzielle Zoonosenausbrüche eingesetzt werden können, damit die nächste Krankheit eingedämmt werden kann, bevor sie zu einer Endemie oder einer neuen Pandemie wird.
Ein neuartiger Ansatz zur Pandemieprävention
Im Jahr 1998 infizierten sich entlang des Nipah-Flusses in Malaysia zwischen Herbst 1998 und Frühjahr 1999 265 Menschen mit einem damals unbekannten Virus, das akute Enzephalitis und schwere Atemnot verursachte. 105 von ihnen starben, was einer Todesrate von 39.6 % entspricht. Im Gegensatz dazu liegt die unbehandelte Sterblichkeitsrate bei COVID-19 bei 6.3 %. Seitdem hat das Nipah-Virus, wie es jetzt genannt wird, seinen Waldlebensraum verlassen und über 20 tödliche Ausbrüche verursacht, hauptsächlich in Indien und Bangladesch.
Viren wie Nipah tauchen jedes Jahr auf und stellen unser tägliches Leben vor Herausforderungen, insbesondere in Ländern, in denen der Aufbau starker, dauerhafter und robuster Systeme zur Krankheitsüberwachung und -erkennung schwieriger ist. Diese Erkennungssysteme sind entscheidend für die Reduzierung der mit solchen Viren verbundenen Risiken.
Lösungen, die ML- und Geodaten nutzen, wie zum Beispiel den Zoonotic Spillover Risk Index, können lokale Gesundheitsbehörden dabei unterstützen, die Ressourcenzuweisung für Bereiche mit dem höchsten Risiko zu priorisieren. Auf diese Weise können sie gezielte und lokale Überwachungsmaßnahmen einführen, um regionale Ausbrüche zu erkennen und zu stoppen, bevor sie sich über die Grenzen hinaus ausbreiten. Dieser Ansatz kann die Auswirkungen eines Krankheitsausbruchs erheblich begrenzen und Leben retten.
Zusammenfassung
Dieser Beitrag zeigte, wie HSR.Gesundheit hat den Zoonotic Spillover Risk Index erfolgreich entwickelt, indem Geodaten, Gesundheit, soziale Determinanten und ML integriert wurden. Durch den Einsatz von SageMaker hat das Team einen skalierbaren Workflow erstellt, der die größten Bedrohungen einer potenziellen zukünftigen Pandemie erkennen kann. Ein wirksames Management dieser Risiken kann zu einer Verringerung der globalen Krankheitslast führen. Die erheblichen wirtschaftlichen und sozialen Vorteile der Verringerung des Pandemierisikos können nicht genug betont werden, da sich die Vorteile regional und global erstrecken.
HSR.Gesundheit nutzte die Geodatenfunktionen von SageMaker für eine erste Implementierung des Zoonotic Spillover Risk Index und sucht nun nach Partnerschaften sowie nach Unterstützung von Gastländern und Finanzierungsquellen, um den Index weiterzuentwickeln und seine Anwendung auf weitere Regionen auf der ganzen Welt auszudehnen. Weitere Informationen zu HSR.Gesundheit und den Zoonotic Spillover Risk Index, besuchen Sie www.hsr.health.
Entdecken Sie das Potenzial der Integration von Erdbeobachtungsdaten in Ihre Gesundheitsinitiativen, indem Sie die Geodatenfunktionen von SageMaker erkunden. Weitere Informationen finden Sie unter Geodatenfunktionen von Amazon SageMaker, oder engagieren Sie sich mit weitere Beispiele um praktische Erfahrungen zu sammeln.
Über die Autoren
 Ajay K Gupta ist Mitbegründer und CEO von HSR.health, einem Unternehmen, das die Gesundheitsrisikoanalyse durch Geodatentechnologie und KI-Techniken revolutioniert und innoviert, um die Ausbreitung und Schwere von Krankheiten vorherzusagen. Und stellt diese Erkenntnisse der Industrie, den Regierungen und dem Gesundheitssektor zur Verfügung, damit sie zukünftige Risiken vorhersehen, mindern und nutzen können. Außerhalb der Arbeit findet man Ajay hinter dem Mikrofon und lässt sein Trommelfell platzen, während er seine Lieblingspopmusik von U2, Sting, George Michael oder Imagine Dragons singt.
Ajay K Gupta ist Mitbegründer und CEO von HSR.health, einem Unternehmen, das die Gesundheitsrisikoanalyse durch Geodatentechnologie und KI-Techniken revolutioniert und innoviert, um die Ausbreitung und Schwere von Krankheiten vorherzusagen. Und stellt diese Erkenntnisse der Industrie, den Regierungen und dem Gesundheitssektor zur Verfügung, damit sie zukünftige Risiken vorhersehen, mindern und nutzen können. Außerhalb der Arbeit findet man Ajay hinter dem Mikrofon und lässt sein Trommelfell platzen, während er seine Lieblingspopmusik von U2, Sting, George Michael oder Imagine Dragons singt.
 Jean Felipe Teotonio Jean Felipe ist ein engagierter Arzt und leidenschaftlicher Experte für Gesundheitsqualität und Epidemiologie von Infektionskrankheiten und leitet das öffentliche Gesundheitsteam von HSR.health. Er arbeitet auf das gemeinsame Ziel hin, die öffentliche Gesundheit zu verbessern, indem er die globale Krankheitslast verringert, indem er GeoAI-Ansätze nutzt, um Lösungen für die größten gesundheitlichen Herausforderungen unserer Zeit zu entwickeln. Außerhalb der Arbeit gehören zu seinen Hobbys das Lesen von Science-Fiction-Büchern, Wandern, die englische Premier League und das Spielen von Bassgitarre.
Jean Felipe Teotonio Jean Felipe ist ein engagierter Arzt und leidenschaftlicher Experte für Gesundheitsqualität und Epidemiologie von Infektionskrankheiten und leitet das öffentliche Gesundheitsteam von HSR.health. Er arbeitet auf das gemeinsame Ziel hin, die öffentliche Gesundheit zu verbessern, indem er die globale Krankheitslast verringert, indem er GeoAI-Ansätze nutzt, um Lösungen für die größten gesundheitlichen Herausforderungen unserer Zeit zu entwickeln. Außerhalb der Arbeit gehören zu seinen Hobbys das Lesen von Science-Fiction-Büchern, Wandern, die englische Premier League und das Spielen von Bassgitarre.
 Paul A Kirchhof, CTO und Chief Geospatial Engineer für HSR.health, nutzt seine umfassenden technischen Fähigkeiten und sein Fachwissen, um die Kerninfrastruktur des Unternehmens sowie seine patentierte und proprietäre GeoMD-Plattform aufzubauen. Darüber hinaus integrieren er und das Data-Science-Team Geodatenanalysen und KI/ML-Techniken in alle Gesundheitsrisikoindizes, die HSR.health erstellt. Außerhalb der Arbeit ist Paul autodidaktischer DJ und liebt Schnee.
Paul A Kirchhof, CTO und Chief Geospatial Engineer für HSR.health, nutzt seine umfassenden technischen Fähigkeiten und sein Fachwissen, um die Kerninfrastruktur des Unternehmens sowie seine patentierte und proprietäre GeoMD-Plattform aufzubauen. Darüber hinaus integrieren er und das Data-Science-Team Geodatenanalysen und KI/ML-Techniken in alle Gesundheitsrisikoindizes, die HSR.health erstellt. Außerhalb der Arbeit ist Paul autodidaktischer DJ und liebt Schnee.
 Janosch Woschitz ist Senior Solutions Architect bei AWS und auf Geodaten-KI/ML spezialisiert. Mit über 15 Jahren Erfahrung unterstützt er Kunden weltweit bei der Nutzung von KI und ML für innovative Lösungen, die Geodaten nutzen. Seine Fachkenntnisse umfassen maschinelles Lernen, Datentechnik und skalierbare verteilte Systeme, ergänzt durch einen fundierten Hintergrund in der Softwareentwicklung und Branchenexpertise in komplexen Bereichen wie dem autonomen Fahren.
Janosch Woschitz ist Senior Solutions Architect bei AWS und auf Geodaten-KI/ML spezialisiert. Mit über 15 Jahren Erfahrung unterstützt er Kunden weltweit bei der Nutzung von KI und ML für innovative Lösungen, die Geodaten nutzen. Seine Fachkenntnisse umfassen maschinelles Lernen, Datentechnik und skalierbare verteilte Systeme, ergänzt durch einen fundierten Hintergrund in der Softwareentwicklung und Branchenexpertise in komplexen Bereichen wie dem autonomen Fahren.
 Emmett Nelson ist Account Executive bei AWS und unterstützt gemeinnützige Forschungskunden in den Branchen Gesundheitswesen und Biowissenschaften, Erd-/Umweltwissenschaften und Bildung. Sein Hauptaugenmerk liegt auf der Ermöglichung von Anwendungsfällen in den Bereichen Analytik, KI/ML, Hochleistungsrechnen (HPC), Genomik und medizinische Bildgebung. Emmett kam 2020 zu AWS und hat seinen Sitz in Austin, TX.
Emmett Nelson ist Account Executive bei AWS und unterstützt gemeinnützige Forschungskunden in den Branchen Gesundheitswesen und Biowissenschaften, Erd-/Umweltwissenschaften und Bildung. Sein Hauptaugenmerk liegt auf der Ermöglichung von Anwendungsfällen in den Bereichen Analytik, KI/ML, Hochleistungsrechnen (HPC), Genomik und medizinische Bildgebung. Emmett kam 2020 zu AWS und hat seinen Sitz in Austin, TX.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- Quelle: https://aws.amazon.com/blogs/machine-learning/how-hsr-health-is-limiting-risks-of-disease-spillover-from-animals-to-humans-using-amazon-sagemaker-geospatial-capabilities/