Bài đăng này được đồng viết với Sachin Kadyan, một nhà phát triển hàng đầu của OpenFold.
Trong khám phá thuốc, việc hiểu cấu trúc 3D của protein là chìa khóa để đánh giá khả năng liên kết của một loại thuốc với nó, ảnh hưởng trực tiếp đến hiệu quả của nó. Tuy nhiên, dự đoán dạng protein 3D là rất phức tạp, đầy thách thức, tốn kém và tốn thời gian, và có thể mất nhiều năm khi sử dụng các phương pháp truyền thống như nhiễu xạ tia X. Việc áp dụng máy học (ML) để dự đoán những cấu trúc này có thể đẩy nhanh đáng kể thời gian dự đoán cấu trúc protein — từ hàng năm đến hàng giờ. Một số nhóm nghiên cứu nổi tiếng đã phát hành các thuật toán như AlphaFold2 (AF2), RoseTTAFold và các thuật toán khác. Các thuật toán này đã được công nhận bởi Khoa học tạp chí năm 2021 Đột phá của năm.
OpenFold, được phát triển bởi Đại học Columbia, là một mô hình dự đoán cấu trúc protein mã nguồn mở được thực hiện với PyTorch. OpenFold là sự tái tạo trung thực của mô hình dự đoán cấu trúc protein AlphaFold2, đồng thời cung cấp các cải tiến về hiệu suất so với AlphaFold2. Nó chứa một số tối ưu hóa dành riêng cho đào tạo và suy luận tận dụng các hiệu số bộ nhớ / thời gian khác nhau cho các độ dài protein khác nhau dựa trên quá trình đào tạo mô hình hoặc chạy suy luận. Đối với đào tạo, OpenFold hỗ trợ tối ưu hóa FlashAttention để tăng tốc thành phần chú ý liên kết đa trình tự (MSA). Tối ưu hóa FlashAttention cùng với biên dịch JIT đẩy nhanh tiến trình suy luận, mang lại hiệu suất gấp đôi cho các chuỗi protein ngắn hơn AlphaFold2.
Đối với các cấu trúc protein lớn hơn, OpenFold có sự chú ý tại chỗ và tối ưu hóa sự chú ý của bộ nhớ thấp, hỗ trợ các dự đoán về cấu trúc protein dài tới 4,600 dư lượng, dựa trên GPU A40 100 GB Đám mây điện toán đàn hồi Amazon (Amazon EC2) phiên bản p4d. Ngoài ra, với các kỹ thuật tối ưu hóa việc sử dụng bộ nhớ như giảm tải CPU, hoạt động tại chỗ và phân đoạn (tách bộ căng đầu vào), OpenFold có thể dự đoán cấu trúc cho các protein rất lớn mà AlphaFold không thể thực hiện được. Đường dẫn liên kết trong OpenFold hiệu quả hơn AlphaFold với chuỗi công cụ HHBlits / JackHMMER hoặc đường dẫn tạo MSA dựa trên MMSeqs2 nhanh hơn nhiều.
Đại học Columbia đã công bố công khai trọng số mô hình và dữ liệu đào tạo, bao gồm 400,000 tệp tin truy cập mẫu MSAs và PDB70, theo giấy phép được phép. Trọng số mô hình có sẵn thông qua các tập lệnh trong Kho GitHubvà MSA được lưu trữ bởi Đăng ký dữ liệu mở trên AWS (RODA). Việc sử dụng Python và PyTorch để triển khai cho phép OpenFold có quyền truy cập vào một loạt lớn các mô-đun ML và nhà phát triển, do đó đảm bảo cải tiến và tối ưu hóa liên tục của nó.
Trong bài đăng này, chúng tôi chỉ ra cách bạn có thể triển khai các mô hình OpenFold trên Dịch vụ Kubernetes đàn hồi của Amazon (Amazon EKS) và cách chia tỷ lệ các cụm EKS để giảm đáng kể thời gian tính toán MSA và suy luận cấu trúc protein. Amazon EKS là một dịch vụ vùng chứa được quản lý để chạy và mở rộng các ứng dụng Kubernetes trên AWS. Với Amazon EKS, bạn có thể chạy các công việc đào tạo phân tán một cách hiệu quả bằng cách sử dụng các phiên bản EC2 mới nhất mà không cần cài đặt, vận hành và duy trì mặt phẳng điều khiển hoặc các nút của riêng bạn. Nó phổ biến dàn nhạc cho quy trình làm việc ML và AI và dịch vụ điều phối vùng chứa ngày càng phổ biến trong kiến trúc suy luận điển hình cho các ứng dụng như công cụ đề xuất, phân tích cảm tính và xếp hạng quảng cáo cần phân phối một số lượng lớn các mô hình, với sự kết hợp giữa ML cổ điển và học sâu ( DL) mô hình.
Chúng tôi cho thấy hiệu suất của kiến trúc này để chạy tính toán và suy luận căn chỉnh trên tập dữ liệu Cameo nguồn mở phổ biến. Chạy khối lượng công việc này kết thúc trên tất cả 92 protein có sẵn trong Cameo tập dữ liệu sẽ mất tổng cộng 8 giờ, bao gồm tải xuống dữ liệu cần thiết, tính toán căn chỉnh và thời gian suy luận.
Tổng quan về giải pháp
Chúng tôi hướng dẫn cách thiết lập một cụm EKS bằng cách sử dụng Amazon FSx cho ánh dưới dạng hệ thống tệp phân tán của chúng tôi. Chúng tôi hướng dẫn bạn cách tải xuống các hình ảnh cần thiết, tệp mô hình, hình ảnh vùng chứa và tệp kê khai .yaml. Chúng tôi cũng chỉ ra cách bạn có thể phục vụ mô hình bằng cách sử dụng FastAPI để dự đoán cấu trúc protein 3D. Bước MSA trong quy trình gấp protein rất chuyên sâu về mặt tính toán và có thể chiếm phần lớn thời gian suy luận. Trong bài đăng này, chúng tôi chỉ ra cách sắp xếp nhiều công việc Kubernetes song song để sử dụng các cụm trên quy mô lớn để đẩy nhanh bước MSA. Cuối cùng, chúng tôi cung cấp so sánh hiệu suất cho các trường hợp máy tính khác nhau và cách bạn có thể theo dõi việc sử dụng CPU và GPU.
Bạn có thể sử dụng kiến trúc tham chiếu trong bài đăng này để kiểm tra các thuật toán gấp khác nhau, kiểm tra các mô hình được đào tạo trước hiện có trên dữ liệu mới hoặc cung cấp các API OpenFold hiệu quả để sử dụng rộng rãi hơn trong tổ chức của bạn.
Thiết lập cụm EKS với hệ thống tệp FSx cho Luster
Chúng tôi sử dụng ôi-ôi-ôi, một dự án mã nguồn mở cung cấp một bộ sưu tập lớn các tập lệnh và công cụ dễ sử dụng và có thể định cấu hình để cho phép bạn cung cấp các cụm EKS và chạy suy luận của bạn. Để tạo cụm bằng kho aws-do-eks, hãy làm theo các bước trong kho lưu trữ GitHub để thiết lập và khởi chạy cụm EKS. Nếu bạn gặp lỗi khi tạo cụm, hãy kiểm tra các lý do có thể xảy ra sau:
- Nếu không tạo được nhóm nút vì không đủ dung lượng, hãy kiểm tra tính khả dụng của phiên bản trong Vùng được yêu cầu và giới hạn dung lượng của bạn.
- Kiểm tra để đảm bảo rằng loại phiên bản được chỉ định có sẵn hoặc được hỗ trợ trong một AZ nhất định.
- Tạo cụm EKS Hình thành đám mây AWS ngăn xếp có thể đã không được xóa đúng cách. Bạn có thể phải kiểm tra các ngăn xếp CloudFormation đang hoạt động để xem liệu việc xóa ngăn xếp có thất bại hay không.
Sau khi cụm được tạo, bạn cần giao diện dòng lệnh kubectl (CLI) trên cá thể EC2 để thực hiện các hoạt động Kubernetes. Trên phiên bản Linux, hãy chạy lệnh sau để cài đặt kubectl CLI. Tham khảo đến lệnh có sẵn cho bất kỳ yêu cầu tùy chỉnh nào.
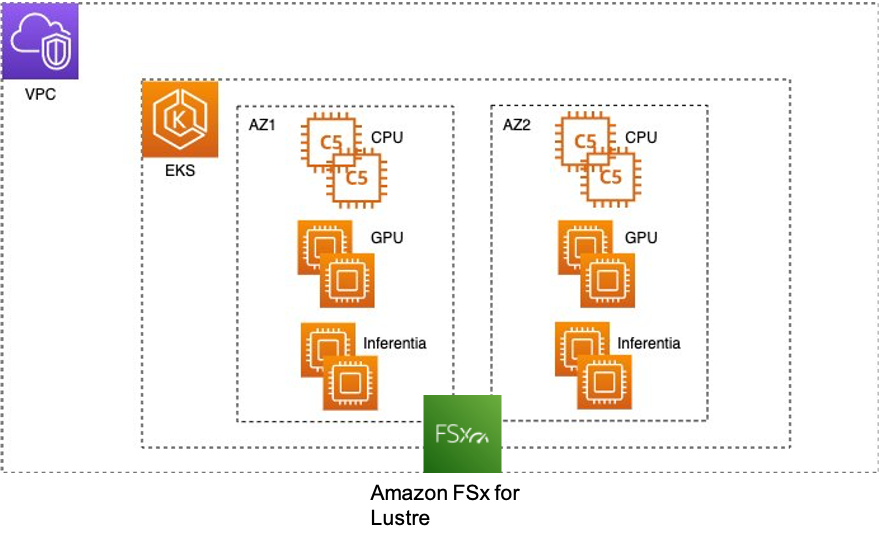
Một cụm EKS điển hình trong AWS trông giống như hình sau.
Chúng tôi cần một hệ thống tệp chia sẻ có thể mở rộng mà tất cả các nút tính toán trong cụm EKS có thể truy cập. FSx for Luster là một hệ thống tệp hiệu suất cao, được quản lý hoàn toàn, cung cấp độ trễ dưới mili giây, thông lượng lên đến hàng trăm GB / giây và hàng triệu IOPS. Để gắn kết hệ thống tệp FSx cho Luster vào cụm EKS, hãy tham khảo Tạo hệ thống tệp và sao chép dữ liệu.
Bạn có thể tạo hệ thống tệp FSx cho Luster trong Vùng khả dụng nơi đặt hầu hết máy tính của bạn để cung cấp độ trễ thấp nhất. Hệ thống tệp có thể được truy cập từ các nút hoặc nhóm trong bất kỳ Vùng sẵn sàng nào. Để đơn giản, trong ví dụ này, chúng tôi giữ các nút trong cùng một Vùng sẵn sàng.
Tải xuống dữ liệu OpenFold và tệp mô hình
Sao chép các ngân hàng dữ liệu tạo tác và protein cần thiết để suy luận từ Dịch vụ lưu trữ đơn giản của Amazon (Amazon S3) nhóm s3://aws-batch-architecture-for-alphafold-public-artifacts/ và s3://pdbsnapshots/ vào hệ thống tệp FSx cho Luster được thiết lập ở bước trước. Cơ sở dữ liệu bao gồm các tham số AlphaFold, dữ liệu phân tích Microbiome từ MGnify, Cơ sở dữ liệu Big Fantastic (BFD), cơ sở dữ liệu Ngân hàng Dữ liệu Protein, tệp mmCIF và cơ sở dữ liệu PDB SeqRes. Các tập lệnh để tải xuống và giải nén dữ liệu có sẵn trong download-openfold-data / scripts thư mục. Sử dụng tệp .yaml fsx-data-prep-pod.yaml để chạy một công việc Kubernetes để tải xuống dữ liệu. Bạn có thể khởi chạy nhiều công việc Kubernetes để đẩy nhanh quá trình này, vì quá trình tải xuống tệp có thể tốn thời gian và mất khoảng 4 giờ. Hoàn thành các bước sau để tải tất cả dữ liệu xuống FSx:
Trong ví dụ này, thư mục FSx cho Luster được chia sẻ của chúng tôi là /fsx-shared, được tạo sau khi khối lượng FSx cho Luster được gắn trên cụm EKS. Khi công việc hoàn tất, bạn sẽ thấy các thư mục sau trong fsx-shared thư mục.
Sao chép Tệp mô hình OpenFold và tải chúng xuống một thùng S3 và từ đó vào hệ thống tệp FSx cho Luster bằng cách sử dụng các bước trước đó. Ảnh chụp màn hình sau đây cho thấy bảy tệp phải có trong hệ thống tệp FSX của bạn sau khi bạn hoàn tất quá trình tải xuống.
Tạo tệp OpenFold Docker và tệp kê khai .yaml
Chúng tôi đã cung cấp một Tệp OpenFold Docker mà bạn có thể sử dụng để xây dựng một vùng chứa cơ sở chứa tất cả các phụ thuộc cần thiết để chạy OpenFold. Để chạy suy luận OpenFold với các mô hình OpenFold được đào tạo trước, bạn cần chạy mã sau:
Sản phẩm run_pretraining_openfold.py mã được cung cấp trong kho OpenFold GitHub là mã suy luận end-to-end lấy đầu vào của người dùng và tính toán các căn chỉnh nếu cần bằng cách sử dụng mã nhị phân jackhmmer và hhsuite, tải mô hình OpenFold và chạy suy luận. Nó cũng bao gồm các chức năng khác, bao gồm thư giãn protein, theo dõi mô hình và đa mô hình, để đặt tên cho một số. Chạy run_pretrained_openfold.py mã trong nhóm Kubernetes bằng tệp .yaml như sau:
Triển khai các mô hình OpenFold dưới dạng dịch vụ và thử nghiệm giải pháp
Để triển khai máy chủ mô hình OpenFold dưới dạng API, bạn cần hoàn thành các bước sau:
- Cập nhật suy luận_config.properties với các thông tin như tên mô hình OpenFold, đường dẫn đến thư mục căn chỉnh, số lượng mô hình sẽ được triển khai trên mỗi máy chủ, kiểu phiên bản máy chủ mô hình, số lượng máy chủ và cổng máy chủ.
- Xây dựng hình ảnh Docker với
build.sh. - Đẩy hình ảnh Docker bằng
push.sh. - Triển khai các mô hình với
deploy.sh.
Nếu bạn cần tùy chỉnh các API OpenFold, hãy sử dụng fastapi-server.py tệp, có tất cả các chức năng quan trọng cần thiết để tải các mô hình OpenFold, tính toán MSA và chạy suy luận.
Khởi tạo model_config, template_featurizer, data_processorvà feature_processor đường ống dẫn trong fastapi-server.py bằng cách gọi các lớp tương ứng của chúng. Các precompute_alignment API lấy thẻ và trình tự protein làm tham số tùy chọn và tạo sự liên kết nếu chưa có. Biến alignment_dir chỉ định vị trí lưu tất cả các căn chỉnh. API precompute_alignment tạo các thư mục liên kết cục bộ bằng cách sử dụng các thẻ của mỗi trình tự protein. Vì lý do này, hãy đảm bảo rằng các thẻ của mỗi protein là duy nhất. Khi API chạy xong, bfd_uniclust_hits.a3m, mgnify_hits.a3m, pdb70_hits.hhrvà uniref90_hits.a3m tệp được tạo trong thư mục căn chỉnh cục bộ.
Gọi openfold_predictions API suy luận, nhận thẻ protein, trình tự và ID mô hình. Sau khi thư mục căn chỉnh cục bộ được xác định, một từ điển tính năng đã xử lý được tạo, cung cấp một loạt đầu vào. Tiếp theo, một lệnh gọi suy luận chuyển tiếp được chạy với mô hình để đưa ra kết quả đầu ra, phải được xử lý sau với prep_output có chức năng tạo ra một loại protein không phân tử.
Khi fastapi-server.py mã được chạy, nó tải nhiều mô hình OpenFold trên mỗi GPU trong nhiều phiên bản. Để theo dõi mô hình nào đang được tải trên mỗi GPU, chúng tôi cần một từ điển mô hình toàn cầu lưu trữ các ID mô hình của từng mô hình. Bạn cần chỉ định tệp điểm kiểm tra nào bạn muốn sử dụng và số lượng mô hình được tải trên mỗi GPU và những mô hình đó được tải khi vùng chứa được chạy, như được hiển thị trong mã sau:
Tệp inference_config.properties có các đầu vào mà bạn cần điền, bao gồm tệp điểm kiểm tra nào sẽ sử dụng, loại phiên bản để suy luận, số lượng máy chủ mô hình và số lượng mô hình được tải trên mỗi máy chủ. Ngoài ra, nó bao gồm các đầu vào khác tương ứng với các đối số đầu vào trong run_pretraining_openfold.py mã, chẳng hạn như số lượng CPU, config_preset, và nhiều hơn nữa. Nếu bạn cần thêm chức năng bổ sung, chẳng hạn như bổ sung giãn protein, bạn có thể thêm các thông số liên quan trong inference_config.properties và thực hiện các thay đổi có liên quan trong fastapi-server.py mã số. Nếu bạn chỉ định các mô hình được chạy trên GPU và, ví dụ: hai máy chủ mô hình với hai mô hình được triển khai trên mỗi máy chủ, bốn ứng dụng Kubernetes sẽ được triển khai (xem bên dưới).
Điều quan trọng là phải chỉ định không gian tên mặc định, nếu không có thể có phức tạp khi truy cập khối lượng chia sẻ FSx cho Luster từ tài nguyên tính toán trong môi trường không gian tên tùy chỉnh.
Sản phẩm triển khai thư mục cung cấp tệp kê khai .yaml mẫu để triển khai mô hình OpenFold dưới dạng dịch vụ và tập lệnh shell create-yaml.sh tạo tệp .yaml cho từng dịch vụ trong một thư mục cụ thể. Ví dụ: nếu bạn chỉ định hai máy chủ mô hình và loại phiên bản p3.2xlarge, openfold-gpu-0.yaml và openfold-gpu-1.yaml các tệp được tạo trong app-openfold-gpu-p3.2xlarge thư mục. Sau đó, bạn có thể triển khai các dịch vụ mô hình như sau:
kubectl apply -f app-openfold-gpu-p3.2xlargeSau khi các dịch vụ được triển khai, bạn có thể xem các dịch vụ đã triển khai, như thể hiện trong ảnh chụp màn hình sau.
Chạy tính toán căn chỉnh
Việc hiển thị chức năng tính toán căn chỉnh như một API có thể có một số trường hợp sử dụng cụ thể, nhưng chúng tôi cần có khả năng sử dụng tối ưu cụm EKS để việc tính toán căn chỉnh có thể được thực hiện song song. Chúng tôi không cần các phiên bản dựa trên GPU đắt tiền để tính toán căn chỉnh, vì vậy chúng tôi cần thêm các phiên bản bộ nhớ hoặc máy tính chuyên sâu với một số lượng lớn CPU. Sau khi chúng tôi tạo một cụm EKS, chúng tôi có thể tạo một nhóm nút mới bằng cách chạy eks-nodegroup-create.sh script và chúng tôi có thể chia tỷ lệ các phiên bản từ nhóm tự động điều chỉnh tỷ lệ trên bảng điều khiển Amazon EC2 sau khi chúng tôi đảm bảo rằng các phiên bản nằm trong cùng Vùng khả dụng như FSx cho Luster. Vì tính toán căn chỉnh tốn nhiều bộ nhớ hơn, chúng tôi đã thêm các thể hiện r6 trong cụm EKS.
Sản phẩm cameo thư mục chứa tất cả các tập lệnh có liên quan (tệp Docker; mã Python; tập lệnh xây dựng, đẩy và shell; và tệp kê khai .yaml) giới thiệu cách chạy tính toán căn chỉnh trên tệp FASTA của chuỗi protein. Để chạy tính toán căn chỉnh trên tập dữ liệu FASTA tùy chỉnh của bạn, hãy hoàn thành các bước sau:
- Lưu tệp FASTA trong thư mục FSx.
- Tạo một tệp FASTA tạm thời cho mỗi trình tự protein và lưu nó trong thư mục FSx. Đối với tập dữ liệu Cameo, việc này được thực hiện bằng cách chạy
kubectl apply -f temp-fasta.yamltrongcameo-fastasthư mục. - Cập nhật
alignment_dircon đường trongprecompute_alignments.pymã, chỉ định thư mục đích để lưu các căn chỉnh. - Xây dựng và đẩy hình ảnh Docker lên Đăng ký container đàn hồi Amazon (ECR của Amazon).
- Cập nhật
run-cameo.yamltệp có loại phiên bản và đường dẫn đến hình ảnh Docker trong Amazon ECR và số lượng CPU nếu cần. - Cập nhật
run-grid.pyvới các đường dẫn từ bước 1 và 2. Đoạn mã này đưa vàorun-cameo.yamltệp dưới dạng mẫu, tạo một tệp .yaml cho mỗi công việc tính toán căn chỉnh và lưu chúng trongcameo-yamlsthư mục. - Cuối cùng, gửi tất cả các công việc bằng cách chạy
kubectl apply -f cameo-yamls.
Sản phẩm precompute_alignments.py mã tải một tệp FASTA gồm các trình tự protein. Các run-cameo.yaml tệp được hiển thị trong đoạn mã sau chỉ cần chỉ định loại phiên bản, thông số kỹ thuật gắn kết khối lượng chia sẻ và các đối số như số CPU để tính toán căn chỉnh:
Tùy thuộc vào tính khả dụng của các nút tính toán trong cụm, bạn có thể gửi nhiều công việc Kubernetes song song. Tùy thuộc vào nhu cầu của bạn, bạn có thể có một hoặc nhiều phiên bản dựa trên CPU chuyên dụng. Sau khi bạn tạo nhóm nút loại phiên bản CPU, bạn có thể dễ dàng tăng hoặc giảm tỷ lệ nó theo cách thủ công từ bảng điều khiển Amazon EC2. Nếu nhu cầu phát sinh đối với quy mô cụm tự động, điều đó cũng có thể khả thi với aws-do-eks nhưng sẽ được đưa vào phiên bản sau của giải pháp này.
Kiểm tra hiệu năng
Chúng tôi đã kiểm tra hiệu suất của kiến trúc của chúng tôi trên mã nguồn mở Cameo tập dữ liệu. Bộ dữ liệu này có tổng cộng 92 protein có độ dài khác nhau. Biểu đồ sau đây cho thấy một biểu đồ về độ dài trình tự, có độ dài trình tự trung bình là 236 và bốn trình tự lớn hơn 600.
Chúng tôi đã tạo âm mưu này với mã sau:
Việc tính toán căn chỉnh đòi hỏi nhiều bộ nhớ và không tính toán nhiều, có nghĩa là sử dụng các phiên bản được tối ưu hóa bộ nhớ sẽ tiết kiệm chi phí hơn so với các phiên bản được tối ưu hóa bằng tính toán. Đối với các thử nghiệm của chúng tôi, chúng tôi đã chọn các phiên bản r6i.xlarge, có 4 vCPU và bộ nhớ 32 GB và một nhóm được tách ra cho một công việc tính toán căn chỉnh cho mỗi chuỗi protein.
Bảng sau đây cho thấy kết quả cho các công việc tính toán căn chỉnh. Chúng tôi thấy rằng với 92 trường hợp r6i.xlarge, chúng tôi có thể hoàn thành việc tính toán liên kết cho 92 protein với giá dưới 60 đô la. Để tham khảo, chúng tôi đã so sánh 1 phiên bản c6i.12xlarge chỉ với một nhóm mất hơn 2 ngày để hoàn thành quá trình tính toán.
| Loại sơ thẩm | Tổng bộ nhớ khả dụng | Tổng số vCPU có sẵn | Bộ nhớ nhóm được yêu cầu | CPU Pod được yêu cầu | Số lượng vỏ | Mất thời gian | Chi phí hàng giờ theo yêu cầu | Tổng Chi Phí |
| r6i.xlarge | 32 GB | 4 | 16GB | 4 | 92 | 2.5 giờ | $ 0.252 / giờ | $58 |
| c6i.12xlarge | 96 GB | 48 | Mặc định | 4 | 1 | 49 giờ 43 phút | $ 2.04 / giờ | $101 |
Biểu đồ sau đây cho thấy thời gian tính toán liên kết so với độ dài trình tự protein.
Các biểu đồ sau đây cho thấy mức sử dụng CPU tối đa của 92 × 4 = 368 vCPU trong nhóm tự động mở rộng r6i.xlarge. Cốt truyện dưới cùng chỉ là sự tiếp nối của âm mưu trên cùng. Chúng tôi thấy rằng các CPU đã được sử dụng ở mức công suất tối đa và dần dần đi xuống 0 khi tất cả các công việc kết thúc.
Cuối cùng, sau khi các MSA được tính toán, chúng ta có thể chạy suy luận bằng cách gọi các API máy chủ mô hình. Bảng sau đây cho thấy tổng thời gian suy luận trên tập dữ liệu Cameo cho các trường hợp p3.2xlarge và g4dn.xlarge. Với máy p3.2xlarge, tính toán MSA trên 92 protein của bộ dữ liệu Cameo có thể được thực hiện nhanh hơn gấp ba lần so với máy g4dn.xlarge.
| Loại sơ thẩm | Số lượng GPU | Loại GPU | Tổng số vCPU có sẵn | Bộ nhớ CPU | Bộ nhớ GPU | Tổng thời gian suy luận trên tập dữ liệu Cameo | Chi phí hàng giờ theo yêu cầu | Tổng Chi Phí |
| p3.2xlund | 1 | Tesla V100 | 8 | 61 GiB | 16 | 1.36 giờ | $ 3.06 / giờ | $4 |
| g4dn.xlarge | 1 | Tesla T4 | 4 | 16 GiB | 16 GiB | 3.95 giờ | $ 0.526 / giờ | $2 |
Biểu đồ sau đây cho thấy tổng thời gian cần thiết để tải các tệp MSA và thực hiện suy luận trên một cá thể p3.2xlarge và một cá thể g4dn.xlarge dưới dạng hàm của độ dài trình tự protein. Đối với các chuỗi dài hơn 200, thời gian suy luận với phiên bản p3.2xlarge nhanh hơn ba lần so với phiên bản g4dn.xlarge, trong khi đối với các chuỗi ngắn hơn, nó nhanh hơn 1–2 lần.
Làm sạch
Điều quan trọng là phải rút bớt tài nguyên sau khi đào tạo mô hình để tránh chi phí liên quan đến việc chạy các phiên bản không hoạt động. Với mỗi tập lệnh tạo tài nguyên, Repo GitHub cung cấp một tập lệnh phù hợp để xóa chúng. Để dọn dẹp thiết lập của chúng tôi, chúng tôi phải xóa hệ thống tệp FSx trước khi xóa cụm, vì nó được liên kết với một mạng con trong VPC của cụm. Để xóa hệ thống tệp FSx, hãy chạy lệnh sau từ bên trong fsx thư mục:
Lưu ý rằng điều này sẽ không chỉ xóa ổ đĩa liên tục, nó cũng sẽ xóa hệ thống tệp FSx và tất cả dữ liệu trên hệ thống tệp sẽ bị mất.
Khi bước này hoàn tất, chúng tôi có thể xóa cụm bằng cách sử dụng tập lệnh sau trong Ví dụ thư mục:
Thao tác này sẽ xóa tất cả các nhóm hiện có, xóa cụm và xóa VPC đã tạo từ đầu.
Kết luận
Trong bài đăng này, chúng tôi đã chỉ ra cách sử dụng cụm EKS để chạy suy luận với các mô hình OpenFold. Chúng tôi đã xuất bản các hướng dẫn về Kiến trúc AWS EKS cho suy luận OpenFold GitHub repo, nơi bạn có thể tìm thấy hướng dẫn từng bước về cách tạo cụm EKS, gắn kết hệ thống tệp được chia sẻ với nó, tải xuống dữ liệu OpenFold, thực hiện tính toán MSA và triển khai các mô hình OpenFold dưới dạng API. Để biết thêm thông tin về OpenFold, hãy truy cập repo OpenFold GitHub.
Giới thiệu về tác giả
 Shubha Kumbadakone là Sr. GTM Chuyên gia về học máy tự quản lý, tập trung vào phần mềm và công cụ mã nguồn mở. Cô có hơn 17 năm kinh nghiệm trong lĩnh vực cơ sở hạ tầng đám mây và máy học và đang giúp khách hàng xây dựng chương trình đào tạo và suy luận phân tán trên quy mô lớn cho các mô hình ML của họ trên AWS. Cô cũng có bằng sáng chế về thuật toán bộ nhớ đệm để khôi phục nhanh chóng từ chế độ ngủ đông cho các hệ thống di động.
Shubha Kumbadakone là Sr. GTM Chuyên gia về học máy tự quản lý, tập trung vào phần mềm và công cụ mã nguồn mở. Cô có hơn 17 năm kinh nghiệm trong lĩnh vực cơ sở hạ tầng đám mây và máy học và đang giúp khách hàng xây dựng chương trình đào tạo và suy luận phân tán trên quy mô lớn cho các mô hình ML của họ trên AWS. Cô cũng có bằng sáng chế về thuật toán bộ nhớ đệm để khôi phục nhanh chóng từ chế độ ngủ đông cho các hệ thống di động.
 Ankur Srivastava là một Kiến trúc sư Giải pháp Sr. trong Nhóm ML Frameworks. Anh ấy tập trung vào việc giúp đỡ khách hàng bằng cách tự quản lý và đào tạo phân tán trên quy mô lớn trên AWS. Kinh nghiệm của anh ấy bao gồm bảo trì dự đoán công nghiệp, cặp song sinh kỹ thuật số, tối ưu hóa thiết kế theo xác suất và đã hoàn thành nghiên cứu tiến sĩ từ Kỹ sư cơ khí tại Đại học Rice và nghiên cứu sau tiến sĩ tại Viện Công nghệ Massachusetts.
Ankur Srivastava là một Kiến trúc sư Giải pháp Sr. trong Nhóm ML Frameworks. Anh ấy tập trung vào việc giúp đỡ khách hàng bằng cách tự quản lý và đào tạo phân tán trên quy mô lớn trên AWS. Kinh nghiệm của anh ấy bao gồm bảo trì dự đoán công nghiệp, cặp song sinh kỹ thuật số, tối ưu hóa thiết kế theo xác suất và đã hoàn thành nghiên cứu tiến sĩ từ Kỹ sư cơ khí tại Đại học Rice và nghiên cứu sau tiến sĩ tại Viện Công nghệ Massachusetts.
 Sachin Kadyan là nhà phát triển hàng đầu của OpenFold.
Sachin Kadyan là nhà phát triển hàng đầu của OpenFold.
- Coinsmart. Sàn giao dịch Bitcoin và tiền điện tử tốt nhất Châu Âu.Bấm vào đây
- Platoblockchain. Web3 Metaverse Intelligence. Khuếch đại kiến thức. Truy cập Tại đây.
- nguồn: https://aws.amazon.com/blogs/machine-learning/run-inference-at-scale-for-openfold-a-pytorch-based-protein-folding-ml-model-using-amazon-eks/