In dit bericht demonstreren we hoe we een state-of-the-art eiwittaalmodel (pLM) efficiënt kunnen verfijnen om subcellulaire lokalisatie van eiwitten te voorspellen met behulp van Amazon Sage Maker.
Eiwitten zijn de moleculaire machines van het lichaam, verantwoordelijk voor alles, van het bewegen van uw spieren tot het reageren op infecties. Ondanks deze verscheidenheid zijn alle eiwitten gemaakt van zich herhalende ketens van moleculen die aminozuren worden genoemd. Het menselijk genoom codeert voor twintig standaardaminozuren, elk met een iets andere chemische structuur. Deze kunnen worden weergegeven door letters van het alfabet, waardoor we eiwitten als een tekstreeks kunnen analyseren en verkennen. Het enorme mogelijke aantal eiwitsequenties en -structuren is wat eiwitten hun grote verscheidenheid aan toepassingen geeft.
Eiwitten spelen ook een sleutelrol bij de ontwikkeling van geneesmiddelen, als potentiële doelwitten maar ook als therapieën. Zoals blijkt uit de volgende tabel waren veel van de best verkopende medicijnen in 2022 eiwitten (vooral antilichamen) of andere moleculen zoals mRNA die in het lichaam in eiwitten werden omgezet. Hierdoor moeten veel life science-onderzoekers vragen over eiwitten sneller, goedkoper en nauwkeuriger beantwoorden.
| Naam | Fabrikant | Wereldwijde omzet 2022 ($ miljarden USD) | Indicaties |
| Gemeenschap | Pfizer / BioNTech | $40.8 | Covid-19 |
| spiekvax | modern | $21.8 | Covid-19 |
| Humira | Abbvie | $21.6 | Artritis, de ziekte van Crohn en anderen |
| Keytruda | Merck | $21.0 | Verschillende vormen van kanker |
Gegevensbron: Urquhart, L. Topbedrijven en medicijnen naar omzet in 2022. Natuurrecensies Drug Discovery 22, 260–260 (2023).
Omdat we eiwitten kunnen weergeven als reeksen karakters, kunnen we ze analyseren met behulp van technieken die oorspronkelijk voor geschreven taal zijn ontwikkeld. Dit omvat grote taalmodellen (LLM's) die vooraf zijn getraind op enorme datasets, die vervolgens kunnen worden aangepast voor specifieke taken, zoals het samenvatten van teksten of chatbots. Op dezelfde manier worden pLM's vooraf getraind in grote eiwitsequentiedatabases met behulp van ongelabeld, zelfgecontroleerd leren. We kunnen ze aanpassen om zaken als de 3D-structuur van een eiwit te voorspellen of hoe het kan interageren met andere moleculen. Onderzoekers hebben zelfs pLM's gebruikt om nieuwe eiwitten helemaal opnieuw te ontwerpen. Deze hulpmiddelen vervangen de menselijke wetenschappelijke expertise niet, maar ze hebben het potentieel om de preklinische ontwikkeling en het ontwerp van proeven te versnellen.
Een uitdaging bij deze modellen is hun formaat. Zowel LLM's als pLM's zijn de afgelopen jaren met ordes van grootte gegroeid, zoals geïllustreerd in de volgende figuur. Dit betekent dat het lang kan duren om ze met voldoende nauwkeurigheid te trainen. Het betekent ook dat u hardware, vooral GPU's, met grote hoeveelheden geheugen moet gebruiken om de modelparameters op te slaan.
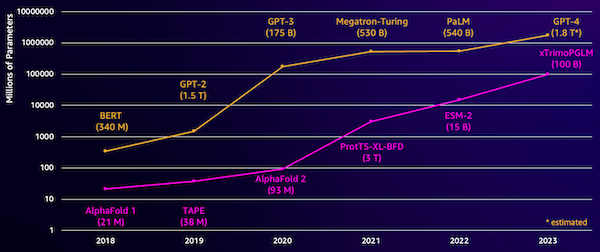
Lange trainingtijden en grote instances zorgen voor hoge kosten, waardoor dit werk voor veel onderzoekers buiten bereik kan komen. In 2023 zal bijvoorbeeld a onderzoeksteam beschreef het trainen van een pLM met 100 miljard parameters op 768 A100 GPU's gedurende 164 dagen! Gelukkig kunnen we in veel gevallen tijd en middelen besparen door een bestaande pLM aan te passen aan onze specifieke taak. Deze techniek heet scherpstellen, en stelt ons ook in staat geavanceerde tools te lenen van andere soorten taalmodellering.
Overzicht oplossingen
Het specifieke probleem dat we in dit bericht behandelen is subcellulaire lokalisatie: Kunnen we, gegeven een eiwitsequentie, een model bouwen dat kan voorspellen of het aan de buitenkant (celmembraan) of binnenkant van een cel leeft? Dit is een belangrijk stukje informatie dat ons kan helpen de functie te begrijpen en of het een goed medicijndoelwit zou zijn.
We beginnen met het downloaden van een openbare dataset met behulp van Amazon SageMaker Studio. Vervolgens gebruiken we SageMaker om het ESM-2-eiwittaalmodel te verfijnen met behulp van een efficiënte trainingsmethode. Ten slotte zetten we het model in als een real-time inferentie-eindpunt en gebruiken we het om enkele bekende eiwitten te testen. Het volgende diagram illustreert deze werkstroom.

In de volgende secties doorlopen we de stappen om uw trainingsgegevens voor te bereiden, een trainingsscript te maken en een SageMaker-trainingstaak uit te voeren. Alle code in dit bericht is beschikbaar op GitHub.
Bereid de trainingsgegevens voor
Wij gebruiken een deel van de DeepLoc-2-gegevensset, dat enkele duizenden SwissProt-eiwitten bevat met experimenteel bepaalde locaties. We filteren op hoogwaardige sequenties tussen 100 en 512 aminozuren:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Vervolgens tokeniseren we de reeksen en splitsen ze op in trainings- en evaluatiesets:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Ten slotte uploaden we de verwerkte trainings- en evaluatiegegevens naar Amazon eenvoudige opslagservice (Amazone S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Maak een trainingsscript
SageMaker-scriptmodus Hiermee kunt u uw aangepaste trainingscode uitvoeren in geoptimaliseerde machine learning (ML) framework-containers die worden beheerd door AWS. Voor dit voorbeeld passen we an bestaand script voor tekstclassificatie van Knuffelgezicht. Hierdoor kunnen we verschillende methoden uitproberen om de efficiëntie van ons trainingswerk te verbeteren.
Methode 1: Gewogen trainingsles
Zoals veel biologische datasets zijn de DeepLoc-gegevens ongelijk verdeeld, wat betekent dat er niet een gelijk aantal membraan- en niet-membraaneiwitten is. We zouden onze gegevens opnieuw kunnen bemonsteren en records uit de meerderheidsklasse kunnen weggooien. Dit zou echter de totale trainingsgegevens verminderen en mogelijk onze nauwkeurigheid schaden. In plaats daarvan berekenen we tijdens de trainingsopdracht de klassegewichten en gebruiken deze om het verlies aan te passen.
In ons trainingsscript verdelen we de Trainer klas van transformers met een WeightedTrainer klasse die rekening houdt met klassegewichten bij het berekenen van kruis-entropieverlies. Dit helpt vertekeningen in ons model te voorkomen:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMethode 2: Gradiëntaccumulatie
Gradiëntaccumulatie is een trainingstechniek waarmee modellen training op grotere batchgroottes kunnen simuleren. Normaal gesproken wordt de batchgrootte (het aantal monsters dat wordt gebruikt om de gradiënt in één trainingsstap te berekenen) beperkt door de GPU-geheugencapaciteit. Bij gradiëntaccumulatie berekent het model eerst de gradiënten op kleinere batches. Vervolgens worden, in plaats van de modelgewichten meteen bij te werken, de gradiënten over meerdere kleine batches verzameld. Wanneer de geaccumuleerde gradiënten gelijk zijn aan de beoogde grotere batchgrootte, wordt de optimalisatiestap uitgevoerd om het model bij te werken. Hierdoor kunnen modellen met effectief grotere batches trainen zonder de GPU-geheugenlimiet te overschrijden.
Er zijn echter extra berekeningen nodig voor de kleinere batches voorwaartse en achterwaartse passen. Grotere batchgroottes via gradiëntaccumulatie kunnen de training vertragen, vooral als er te veel accumulatiestappen worden gebruikt. Het doel is om het GPU-gebruik te maximaliseren, maar excessieve vertragingen als gevolg van te veel extra gradiëntberekeningsstappen te vermijden.
Methode 3: Verloopcontrolepunten
Gradient checkpointing is een techniek die het benodigde geheugen tijdens de training vermindert, terwijl de rekentijd redelijk blijft. Grote neurale netwerken nemen veel geheugen in beslag omdat ze alle tussenliggende waarden van de voorwaartse doorgang moeten opslaan om de gradiënten tijdens de achterwaartse doorgang te berekenen. Dit kan geheugenproblemen veroorzaken. Een oplossing is om deze tussenwaarden niet op te slaan, maar dan tijdens de achterwaartse pass opnieuw te berekenen, wat veel tijd kost.
Gradiëntcontrolepunten bieden een evenwichtige aanpak. Het slaat slechts enkele van de tussenliggende waarden op, genaamd checkpointsen herberekent de andere indien nodig. Daarom gebruikt het minder geheugen dan alles op te slaan, maar ook minder rekenkracht dan alles opnieuw te berekenen. Door strategisch te selecteren welke activaties moeten worden gecontroleerd, maakt gradiëntcontrolepunten het mogelijk grote neurale netwerken te trainen met beheersbaar geheugengebruik en rekentijd. Deze belangrijke techniek maakt het mogelijk om zeer grote modellen te trainen die anders tegen geheugenbeperkingen zouden aanlopen.
In ons trainingsscript schakelen we gradiëntactivering en controlepunten in door de benodigde parameters toe te voegen aan de TrainingArguments voorwerp:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Methode 4: Aanpassing van LLM's op lage rang
Grote taalmodellen zoals ESM-2 kunnen miljarden parameters bevatten die duur zijn om te trainen en uit te voeren. onderzoekers ontwikkelde een trainingsmethode genaamd Low-Rank Adaptation (LoRA) om het verfijnen van deze enorme modellen efficiënter te maken.
Het belangrijkste idee achter LoRA is dat u bij het verfijnen van een model voor een specifieke taak niet alle oorspronkelijke parameters hoeft bij te werken. In plaats daarvan voegt LoRA nieuwe kleinere matrices toe aan het model die de inputs en outputs transformeren. Alleen deze kleinere matrices worden bijgewerkt tijdens het afstemmen, wat veel sneller is en minder geheugen gebruikt. De oorspronkelijke modelparameters blijven bevroren.
Na verfijning met LoRA kunt u de kleine aangepaste matrices weer samenvoegen in het originele model. Of u kunt ze gescheiden houden als u het model snel wilt afstemmen op andere taken zonder eerdere taken te vergeten. Over het geheel genomen zorgt LoRA ervoor dat LLM's efficiënt kunnen worden aangepast aan nieuwe taken tegen een fractie van de gebruikelijke kosten.
In ons trainingsscript configureren we LoRA met behulp van de PEFT bibliotheek van Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Dien een SageMaker-trainingstaak in
Nadat u uw trainingsscript hebt gedefinieerd, kunt u een SageMaker-trainingstaak configureren en indienen. Geef eerst de hyperparameters op:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Bepaal vervolgens welke statistieken u wilt vastleggen uit de trainingslogboeken:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Definieer ten slotte een Hugging Face-schatter en verzend deze voor training op een ml.g5.2xlarge-instantietype. Dit is een kosteneffectief exemplaartype dat algemeen beschikbaar is in veel AWS-regio's:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)De volgende tabel vergelijkt de verschillende trainingsmethoden die we hebben besproken en hun effect op de runtime, nauwkeurigheid en GPU-geheugenvereisten van ons werk.
| Configuratie | Factureerbare tijd (min) | Evaluatienauwkeurigheid | Maximaal GPU-geheugengebruik (GB) |
| Basismodel | 28 | 0.91 | 22.6 |
| Basis + GA | 21 | 0.90 | 17.8 |
| Basis + GC | 29 | 0.91 | 10.2 |
| Basis + LoRA | 23 | 0.90 | 18.6 |
Alle methoden leverden modellen op met een hoge evaluatienauwkeurigheid. Het gebruik van LoRA en gradiëntactivering verminderde de looptijd (en kosten) met respectievelijk 18% en 25%. Het gebruik van gradiëntcontrolepunten verminderde het maximale GPU-geheugengebruik met 55%. Afhankelijk van uw beperkingen (kosten, tijd, hardware) kan een van deze benaderingen zinvoller zijn dan de andere.
Elk van deze methoden presteert op zichzelf goed, maar wat gebeurt er als we ze in combinatie gebruiken? De volgende tabel vat de resultaten samen.
| Configuratie | Factureerbare tijd (min) | Evaluatienauwkeurigheid | Maximaal GPU-geheugengebruik (GB) |
| Alle methoden | 12 | 0.80 | 3.3 |
In dit geval zien we een nauwkeurigheidsvermindering van 12%. We hebben echter de runtime met 57% en het GPU-geheugengebruik met 85% verlaagd! Dit is een enorme afname waardoor we kunnen trainen op een breed scala aan kosteneffectieve instancetypen.
Opruimen
Als u meedoet in uw eigen AWS-account, verwijder dan alle realtime eindpunten en gegevens die u heeft gemaakt om verdere kosten te voorkomen.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Conclusie
In dit bericht hebben we laten zien hoe je eiwittaalmodellen zoals ESM-2 efficiënt kunt verfijnen voor een wetenschappelijk relevante taak. Voor meer informatie over het gebruik van de Transformers- en PEFT-bibliotheken om pLMS te trainen, bekijk de berichten Diep leren met eiwitten en ESMBind (ESMB): Low Rank-aanpassing van ESM-2 voor voorspelling van eiwitbindingsplaatsen op de Hugging Face-blog. U kunt ook meer voorbeelden vinden van het gebruik van machine learning om eiwiteigenschappen te voorspellen in de Geweldige eiwitanalyse op AWS GitHub-opslagplaats.
Over de auteur
 Brian trouw is een Senior AI/ML Solutions Architect in het Global Healthcare and Life Sciences-team bij Amazon Web Services. Hij heeft meer dan 17 jaar ervaring in biotechnologie en machine learning en is gepassioneerd om klanten te helpen bij het oplossen van genomische en proteomische uitdagingen. In zijn vrije tijd kookt en eet hij graag met zijn vrienden en familie.
Brian trouw is een Senior AI/ML Solutions Architect in het Global Healthcare and Life Sciences-team bij Amazon Web Services. Hij heeft meer dan 17 jaar ervaring in biotechnologie en machine learning en is gepassioneerd om klanten te helpen bij het oplossen van genomische en proteomische uitdagingen. In zijn vrije tijd kookt en eet hij graag met zijn vrienden en familie.
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- PlatoData.Network Verticale generatieve AI. Versterk jezelf. Toegang hier.
- PlatoAiStream. Web3-intelligentie. Kennis versterkt. Toegang hier.
- PlatoESG. carbon, CleanTech, Energie, Milieu, Zonne, Afvalbeheer. Toegang hier.
- Plato Gezondheid. Intelligentie op het gebied van biotech en klinische proeven. Toegang hier.
- Bron: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



