Dit bericht is geschreven in samenwerking met Stephen Aylward, Matt McCormick, Brianna Major van Kitware en Justin Kirby van het Frederick National Laboratory for Cancer Research (FNLCR).
Amazon SageMaker Studiolab biedt gratis toegang naar een machine learning (ML) ontwikkelomgeving voor iedereen met een e-mailadres. Zoals de volledig uitgeruste Amazon SageMaker Studio, kunt u met Studio Lab uw eigen instellingen aanpassen Conda-omgeving en maak CPU- en GPU-schaalbaar JupyterLab versie 3 notebooks, met gemakkelijke toegang tot de nieuwste productiviteitstools voor datawetenschap en open-sourcebibliotheken. Bovendien omvatten gratis Studio Lab-accounts minimaal 15 GB permanente opslag, waardoor u uw projecten continu kunt onderhouden en uitbreiden over meerdere sessies en u in staat stelt direct verder te gaan waar u was gebleven en zelfs uw lopende werk en werkomgevingen met anderen kunt delen.
Een belangrijk probleem waarmee de medische beeldgemeenschap wordt geconfronteerd, is hoe onderzoekers in staat kunnen worden gesteld om met deze essentiële hulpmiddelen te experimenteren en te verkennen. Om deze uitdaging op te lossen, werkten AWS-teams samen met Kitgerei en Frederick Nationaal Laboratorium voor Kankeronderzoek (FNLCR) om drie belangrijke AI-bronnen voor medische beeldvorming samen te brengen voor Studio Lab en de hele open-source JupyterLab-gemeenschap:
Dankzij deze tools en gegevens kunnen AI-onderzoekers op het gebied van medische beeldvorming snel klinisch bruikbare deep learning-algoritmen ontwikkelen en grondig evalueren in een uitgebreide en gebruiksvriendelijke omgeving. Teamleden van FNLCR en Kitware werkten samen om een reeks Jupyter-notebooks te maken die gemeenschappelijke workflows demonstreren om programmatisch toegang te krijgen tot TCIA-gegevens en deze te visualiseren. Deze notebooks maken gebruik van Studio Lab zodat onderzoekers de notebooks kunnen gebruiken zonder dat ze hun eigen lokale Jupyter-ontwikkelomgeving hoeven op te zetten. U kunt snel nieuwe ideeën verkennen of uw werk integreren in presentaties, workshops en tutorials op conferenties.
Het volgende voorbeeld illustreert Studio Lab met een Jupyter-notebook die TCIA-prostaat-MRI-gegevens downloadt, deze segmenteert met MONAI en de resultaten weergeeft met itkWidgets.
Hoewel je gemakkelijk kleinere experimenten en demo's kunt uitvoeren met de voorbeeldnotitieboekjes die in dit bericht op Studio Lab worden gepresenteerd, is het aan te raden om Amazon SageMaker Studio wanneer u uw eigen medische beeldmodellen op schaal traint. Amazon SageMaker Studio is een geïntegreerde webgebaseerde ontwikkelomgeving (IDE) met beveiligings-, governance- en monitoringfuncties op bedrijfsniveau van waaruit u toegang hebt tot speciaal gebouwde tools om alle ML-ontwikkelingsstappen uit te voeren. Open-sourcebibliotheken zoals MONAI Core en itkWidgets draaien ook op Amazon SageMaker Studio.
Installeer de oplossing
Om de TCIA-notebooks op Studio Lab uit te voeren, moet u een account registreren met uw e-mailadres op de Studio Lab-website. Het kan 1 tot 3 dagen duren voordat accountverzoeken worden goedgekeurd.
Daarna kunt u de installatiestappen volgen om aan de slag te gaan:
- Log in bij StudioLab en start een CPU-runtime.
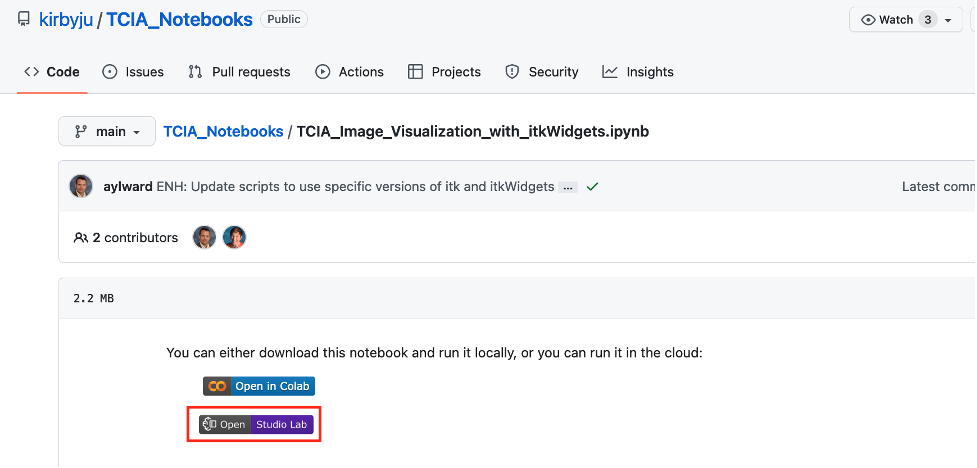
- Navigeer op een apart tabblad naar de TCIA-notebooks GitHub-opslagplaats en kies een notitieblok in de hoofdmap van de repository.
- Kies StudioLab openen om het notitieblok te openen in Studio Lab.
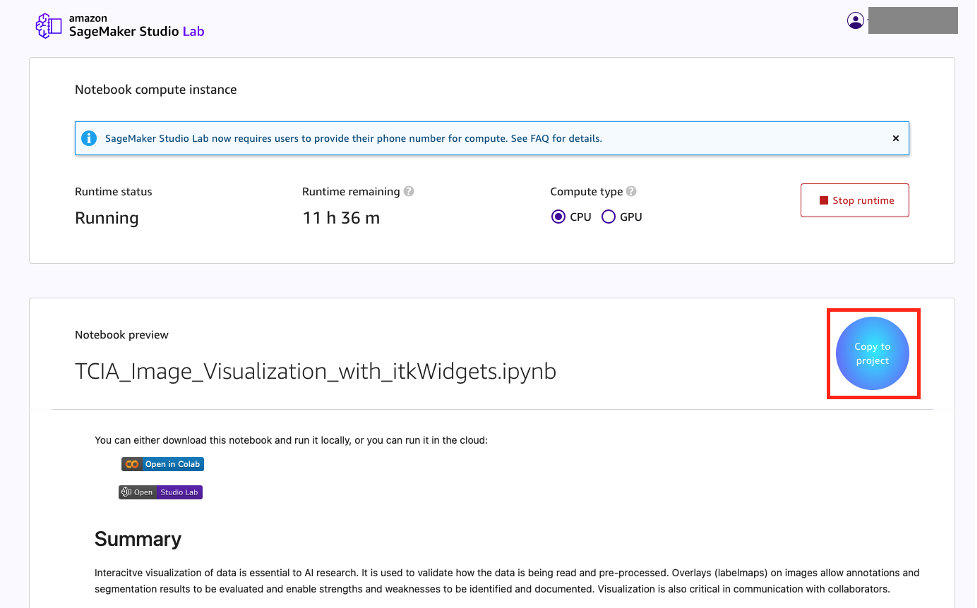
- Terug in Studio Lab, kies Kopiëren naar project.
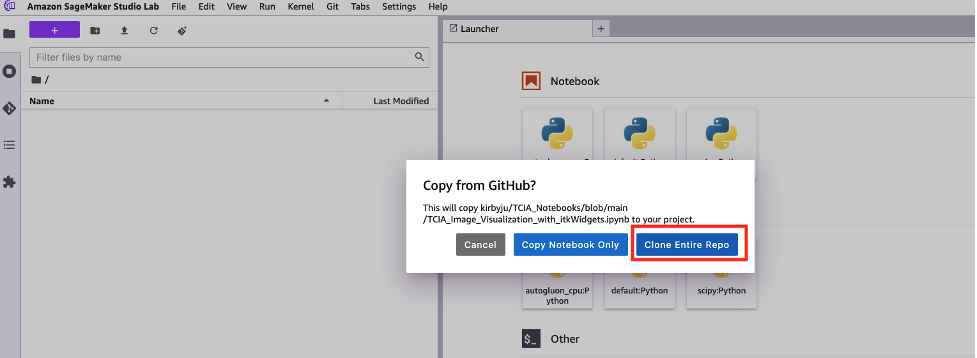
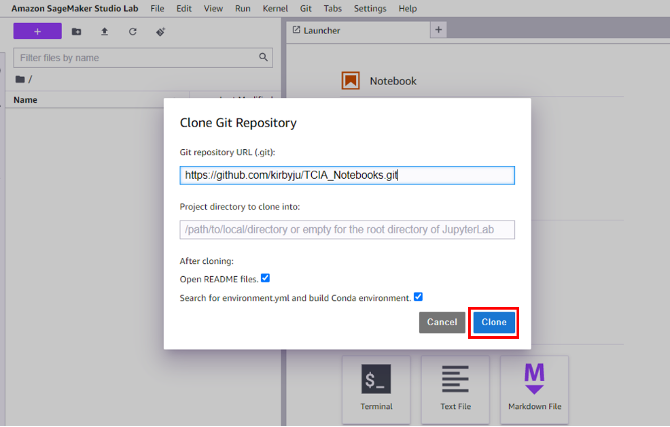
- Kies in de nieuwe pop-up JupyterLab die wordt geopend Kloon de volledige opslagplaats.
- Bewaar in het volgende venster de standaardinstellingen en kies Kloon.
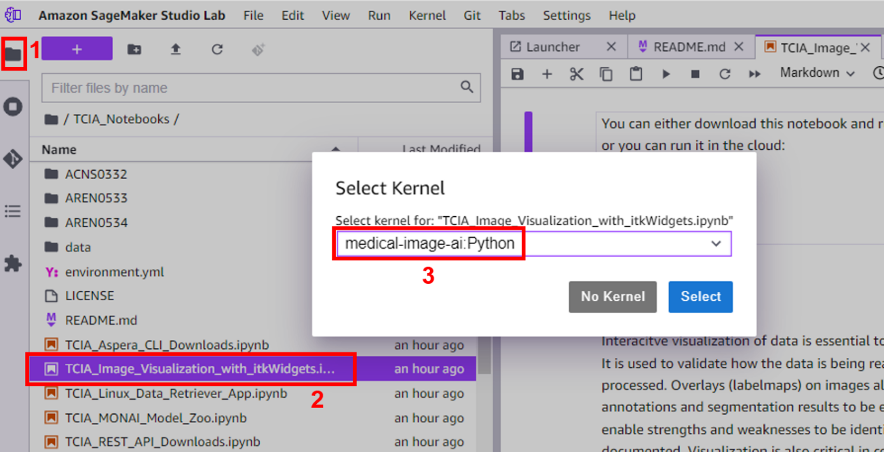
- Kies OK wanneer u wordt gevraagd om te bevestigen dat u de nieuwe Conda-omgeving wilt bouwen (
medical-image-ai). Het bouwen van de Conda-omgeving duurt maximaal 5 minuten.
Het bouwen van de Conda-omgeving duurt maximaal 5 minuten. - Voer in de terminal die in de vorige stap is geopend de volgende opdracht uit om NodeJS te installeren in het
studiolabConda-omgeving, die vereist is om de ImJoy JupyterLab 3-extensie te installeren:conda install -y -c conda-forge nodejs
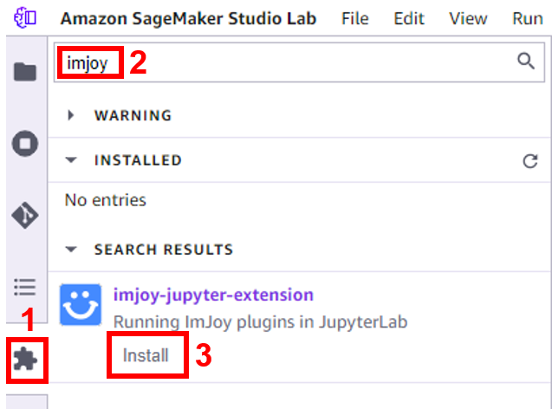
We installeren nu de ImJoy Jupyter-extensie met behulp van de Studio Lab Extension Manager om interactieve visualisaties mogelijk te maken. Met de Imjoy-extensie kunnen itkWidgets en andere data-intensieve processen communiceren met lokale en externe Jupyter-omgevingen, waaronder Jupyter-notebooks, JupyterLab, Studio Lab, enzovoort. - Zoek in Extensiebeheer naar "imjoy" en kies Install.
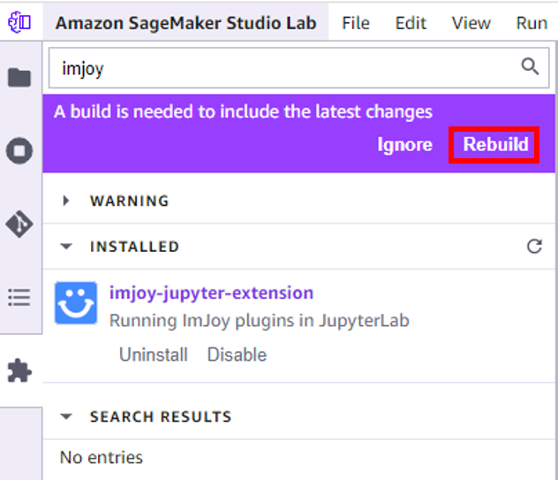
- Bevestig om de kernel opnieuw op te bouwen wanneer daarom wordt gevraagd.
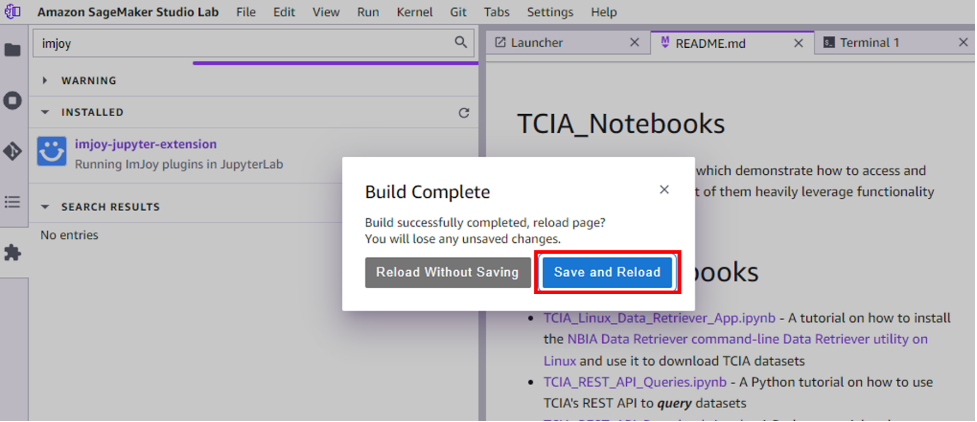
- Kies Opslaan en opnieuw laden als de bouw klaar is.
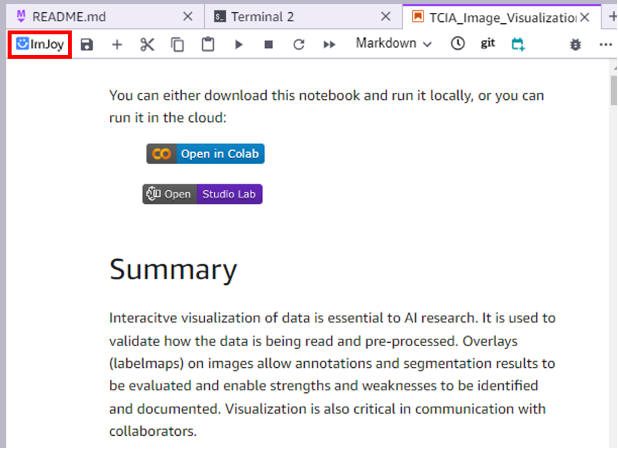
Na de installatie van de ImJoy-extensie ziet u het ImJoy-pictogram in het hoofdmenu van uw notebooks.
Om dit te verifiëren, navigeert u naar de bestandsbrowser, kiest u de TCIA_Image_Visualalization_with_itkWidgets notitieboekje en kies de medical-image-ai kernel om het uit te voeren.
Het ImJoy-pictogram is zichtbaar in de linkerbovenhoek van het notebookmenu.
Met deze installatiestappen hebt u het medical-image-ai Python-kernel en de ImJoy-extensie als voorwaarde om de TCIA-notebooks samen met itkWidgets op Studio Lab uit te voeren.
Test de oplossing
We hebben een set notebooks en een tutorial gemaakt die de integratie van deze AI-technologieën in Studio Lab laten zien. Zorg ervoor dat u de kiest medical-image-ai Python-kernel bij het uitvoeren van de TCIA-notebooks in Studio Lab.
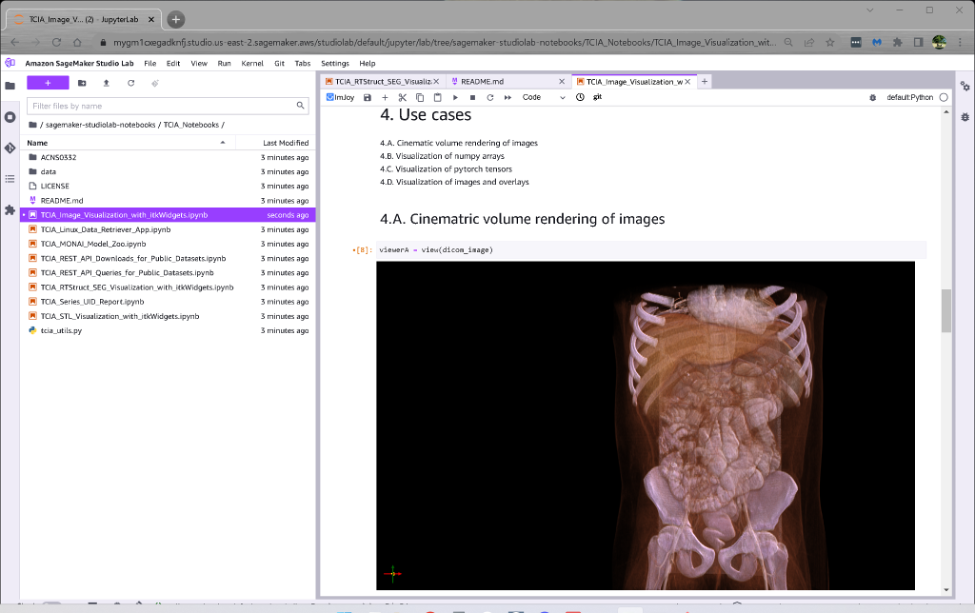
Het eerste SageMaker-notitieboekje laat zien hoe u DICOM-afbeeldingen van TCIA kunt downloaden en deze afbeeldingen kunt visualiseren met behulp van de filmische volumeweergavemogelijkheden van itkWidgets.
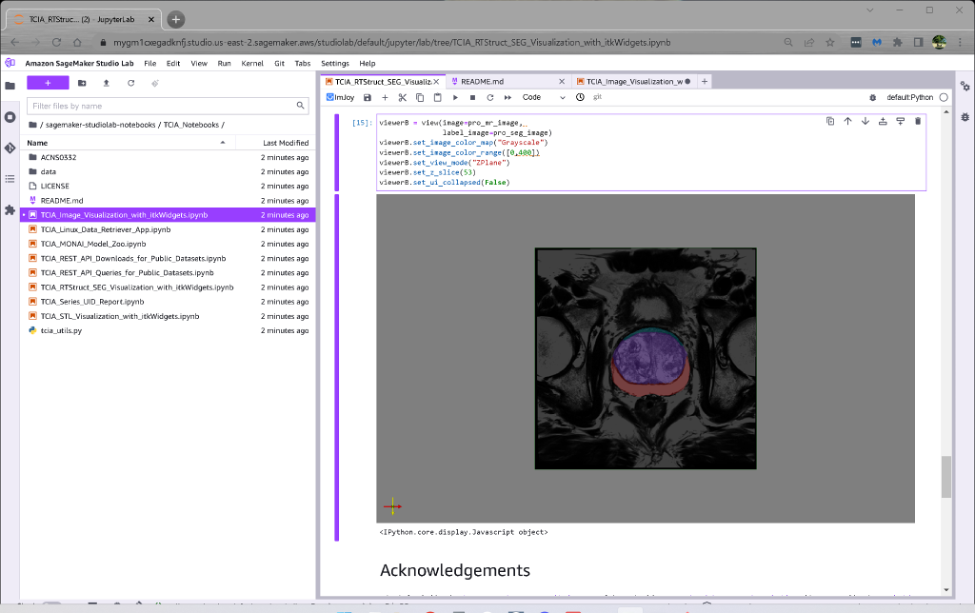
Het tweede notitieboekje laat zien hoe de annotaties van experts die beschikbaar zijn voor honderden onderzoeken naar TCIA kunnen worden gedownload als DICOM SEG- en RTSTRUCT-objecten, gevisualiseerd in 3D of als overlays op 2D-slices, en kunnen worden gebruikt voor training en evaluatie van deep learning-systemen.
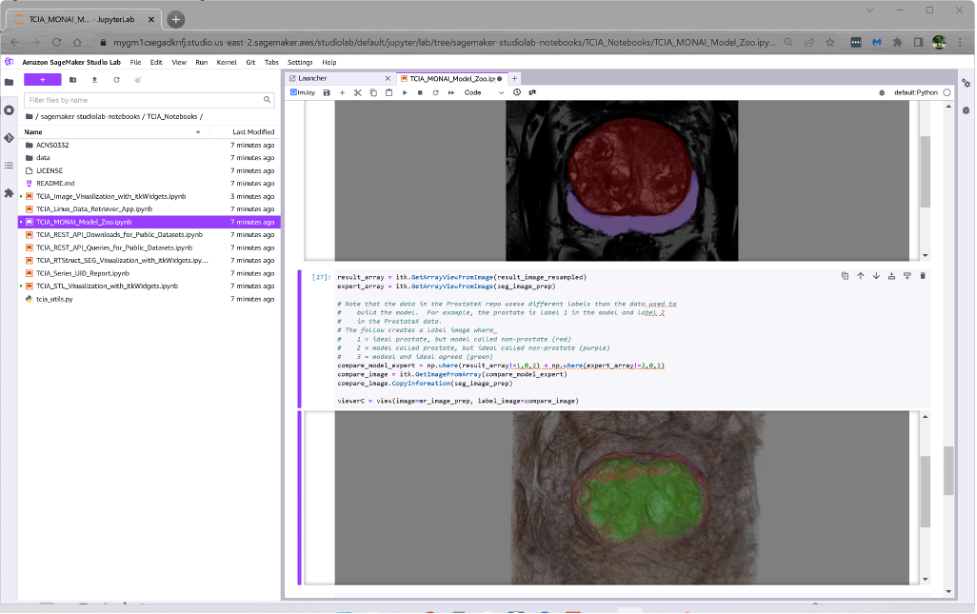
Het derde notitieboekje laat zien hoe vooraf getrainde MONAI deep learning-modellen beschikbaar op MONAI's Model Zoo kunnen worden gedownload en gebruikt om TCIA (of uw eigen) DICOM prostaat MRI-volumes te segmenteren.
Kies StudioLab openen in deze en andere JupyterLab-notebooks om die notebooks te lanceren in de vrij beschikbare Studio Lab-omgeving.
Opruimen
Nadat je de installatiestappen in dit bericht hebt gevolgd en de medical-image-ai Conda-omgeving, wilt u deze misschien verwijderen om opslagruimte te besparen. Gebruik hiervoor de volgende opdracht:
conda remove --name medical-image-ai --allU kunt de ImJoy-extensie ook verwijderen via de Extension Manager. Houd er rekening mee dat u de Conda-omgeving opnieuw moet maken en de ImJoy-extensie opnieuw moet installeren als u later wilt blijven werken met de TCIA-notebooks in uw Studio Lab-account.
Sluit je tabblad en vergeet niet te kiezen Looptijd stoppen op de Studio Lab-projectpagina.
Conclusie
SageMaker Studio Lab is gratis toegankelijk voor medische beeld-AI-onderzoeksgemeenschappen en kan worden gebruikt voor medische beeld-AI-modellering en interactieve medische beeldvisualisatie in combinatie met MONAI en itkWidgets. U kunt de TCIA open data en voorbeeldnotitieboekjes gebruiken met Studio Lab bij trainingsevenementen, zoals hackathons en workshops. Met deze oplossing kunnen wetenschappers en onderzoekers snel experimenteren, samenwerken en innoveren met medische beeld-AI. Als u een AWS-account hebt en een SageMaker Studio-domein hebt ingesteld, kunt u deze notebooks ook op Studio uitvoeren met behulp van de standaard Data Science Python-kernel (met de ImJoy-jupyter-extension geïnstalleerd) terwijl u een keuze maakt uit a verscheidenheid aan typen rekeninstanties.
StudioLab ook heeft een nieuwe functie gelanceerd op AWS re:Invent 2022 om de notebooks die zijn ontwikkeld in Studio Lab te nemen en ze als batchtaken uit te voeren volgens een terugkerend schema in uw AWS-accounts. Daarom kunt u uw ML-experimenten schalen buiten de gratis rekenbeperkingen van Studio Lab en krachtigere rekeninstanties gebruiken met veel grotere datasets op uw AWS-accounts.
Als u meer wilt weten over hoe AWS uw organisatie in de gezondheidszorg of life sciences kan helpen, neem dan contact op met een AWS-vertegenwoordiger. Neem voor meer informatie over MONAI en itkWidgets contact op met Kitgerei. Er worden voortdurend nieuwe gegevens aan TCIA toegevoegd en uw suggesties en bijdragen zijn welkom op de TCIA-website.
Verdere lezing
Over de auteurs
 Stephen Ayward is Senior Director of Strategic Initiatives bij Kitware, adjunct-professor Computer aan de University of North Carolina in Chapel Hill, en een fellow van de MICCAI Society. Dr. Aylward heeft het kantoor van Kitware in North Carolina opgericht, is leider geweest van verschillende open-source-initiatieven en is nu voorzitter van de MONAI-adviesraad.
Stephen Ayward is Senior Director of Strategic Initiatives bij Kitware, adjunct-professor Computer aan de University of North Carolina in Chapel Hill, en een fellow van de MICCAI Society. Dr. Aylward heeft het kantoor van Kitware in North Carolina opgericht, is leider geweest van verschillende open-source-initiatieven en is nu voorzitter van de MONAI-adviesraad.
 Matt McCormick, PhD, is Distinguished Engineer bij Kitware, waar hij leiding geeft aan de ontwikkeling van de Insight Toolkit (ITK), een wetenschappelijke toolkit voor beeldanalyse. Hij was hoofdonderzoeker en mede-onderzoeker van verschillende onderzoekssubsidies van de National Institutes of Health (NIH), leidde samenwerkingen met nationale laboratoria in de Verenigde Staten en leidde verschillende commerciële projecten die geavanceerde software voor medische apparaten leverden. Dr. McCormick is een groot voorstander van door de gemeenschap aangestuurde open-source software, open wetenschap en reproduceerbaar onderzoek.
Matt McCormick, PhD, is Distinguished Engineer bij Kitware, waar hij leiding geeft aan de ontwikkeling van de Insight Toolkit (ITK), een wetenschappelijke toolkit voor beeldanalyse. Hij was hoofdonderzoeker en mede-onderzoeker van verschillende onderzoekssubsidies van de National Institutes of Health (NIH), leidde samenwerkingen met nationale laboratoria in de Verenigde Staten en leidde verschillende commerciële projecten die geavanceerde software voor medische apparaten leverden. Dr. McCormick is een groot voorstander van door de gemeenschap aangestuurde open-source software, open wetenschap en reproduceerbaar onderzoek.
 Brianna majoor is een Research and Development Engineer bij Kitware met een passie voor het ontwikkelen van open source software en tools die de medische en wetenschappelijke gemeenschap ten goede komen.
Brianna majoor is een Research and Development Engineer bij Kitware met een passie voor het ontwikkelen van open source software en tools die de medische en wetenschappelijke gemeenschap ten goede komen.
 Jmet Kirby is technisch projectmanager bij het Frederick National Laboratory for Cancer Research (FNLCR). Zijn werk is gericht op methoden om het delen van gegevens mogelijk te maken met behoud van de privacy van de patiënt om de reproduceerbaarheid en transparantie bij onderzoek naar kankerbeeldvorming te verbeteren. Zijn team richtte in 2010 The Cancer Imaging Archive (TCIA) op, dat de onderzoeksgemeenschap heeft gebruikt om meer dan 200 datasets te publiceren met betrekking tot manuscripten, subsidies, challenge-wedstrijden en belangrijke NCI-onderzoeksinitiatieven. Deze datasets zijn besproken in meer dan 1,500 peer-reviewed publicaties.
Jmet Kirby is technisch projectmanager bij het Frederick National Laboratory for Cancer Research (FNLCR). Zijn werk is gericht op methoden om het delen van gegevens mogelijk te maken met behoud van de privacy van de patiënt om de reproduceerbaarheid en transparantie bij onderzoek naar kankerbeeldvorming te verbeteren. Zijn team richtte in 2010 The Cancer Imaging Archive (TCIA) op, dat de onderzoeksgemeenschap heeft gebruikt om meer dan 200 datasets te publiceren met betrekking tot manuscripten, subsidies, challenge-wedstrijden en belangrijke NCI-onderzoeksinitiatieven. Deze datasets zijn besproken in meer dan 1,500 peer-reviewed publicaties.
 Bende Fu is een Healthcare Solution Architect bij AWS. Hij behaalde een PhD in Farmaceutische Wetenschappen aan de Universiteit van Mississippi en heeft meer dan tien jaar ervaring in technologie en biomedisch onderzoek. Hij is gepassioneerd door technologie en de impact die het kan hebben op de gezondheidszorg.
Bende Fu is een Healthcare Solution Architect bij AWS. Hij behaalde een PhD in Farmaceutische Wetenschappen aan de Universiteit van Mississippi en heeft meer dan tien jaar ervaring in technologie en biomedisch onderzoek. Hij is gepassioneerd door technologie en de impact die het kan hebben op de gezondheidszorg.
 Alex Lemm is Business Development Manager voor medische beeldvorming bij AWS. Alex definieert en voert go-to-market-strategieën uit met beeldvormingspartners en stimuleert de ontwikkeling van oplossingen om op AI/ML gebaseerd onderzoek naar medische beeldvorming in de cloud te versnellen. Hij is gepassioneerd door het integreren van open source ML-frameworks met de AWS AI/ML-stack.
Alex Lemm is Business Development Manager voor medische beeldvorming bij AWS. Alex definieert en voert go-to-market-strategieën uit met beeldvormingspartners en stimuleert de ontwikkeling van oplossingen om op AI/ML gebaseerd onderzoek naar medische beeldvorming in de cloud te versnellen. Hij is gepassioneerd door het integreren van open source ML-frameworks met de AWS AI/ML-stack.
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- Platoblockchain. Web3 Metaverse Intelligentie. Kennis versterkt. Toegang hier.
- Bron: https://aws.amazon.com/blogs/machine-learning/share-medical-image-research-on-amazon-sagemaker-studio-lab-for-free/