この投稿では、最先端のタンパク質言語モデル (pLM) を効率的に微調整して、タンパク質の細胞内局在を予測する方法を説明します。 アマゾンセージメーカー.
タンパク質は体の分子機械であり、筋肉の動きから感染症への対応まで、あらゆることに関与します。この多様性にもかかわらず、すべてのタンパク質はアミノ酸と呼ばれる分子の繰り返し鎖で構成されています。ヒトゲノムは 20 個の標準アミノ酸をコードしており、それぞれがわずかに異なる化学構造を持っています。これらはアルファベットの文字で表すことができるため、タンパク質をテキスト文字列として分析および調査することができます。膨大な数のタンパク質の配列と構造が考えられるため、タンパク質に幅広い用途が与えられます。
タンパク質は、潜在的な標的としてだけでなく治療薬としても、医薬品開発において重要な役割を果たします。次の表に示すように、2022 年に最も売れた医薬品の多くは、タンパク質 (特に抗体)、または体内でタンパク質に翻訳される mRNA などの他の分子でした。このため、多くの生命科学研究者は、タンパク質に関する質問に、より早く、より安く、より正確に答える必要があります。
| 名前 | メーカー | 2022 年の世界売上高 (数十億ドル) | 適応症 |
| コミナティ | ファイザー/ BioNTech | $40.8 | コロナ |
| スパイクバックス | モダン | $21.8 | コロナ |
| ヒュミラ | AbbVie | $21.6 | 関節炎、クローン病など |
| Keytruda | メルク | $21.0 | さまざまながん |
データ ソース: アーカート、L. 2022 年の売上高上位の企業と医薬品。 Nature Reviews Drug Discovery 22、260–260 (2023)。
タンパク質を文字のシーケンスとして表すことができるため、もともと書き言葉用に開発された技術を使用してタンパク質を分析できます。これには、巨大なデータセットで事前トレーニングされた大規模言語モデル (LLM) が含まれており、テキストの要約やチャットボットなどの特定のタスクに適応させることができます。同様に、pLM は、ラベルなしの自己教師あり学習を使用して、大規模なタンパク質配列データベースで事前トレーニングされます。これらを応用して、タンパク質の 3D 構造やタンパク質が他の分子とどのように相互作用するかなどを予測できます。研究者は、pLM を使用して新しいタンパク質をゼロから設計することさえあります。これらのツールは人間の科学的専門知識に代わるものではありませんが、前臨床開発と試験設計をスピードアップする可能性があります。
これらのモデルの課題の 1 つはサイズです。次の図に示すように、LLM と pLM は両方とも、過去数年間で桁違いに成長しました。これは、十分な精度までトレーニングするには長い時間がかかる可能性があることを意味します。また、モデル パラメーターを保存するには、大量のメモリを備えたハードウェア、特に GPU を使用する必要があることも意味します。
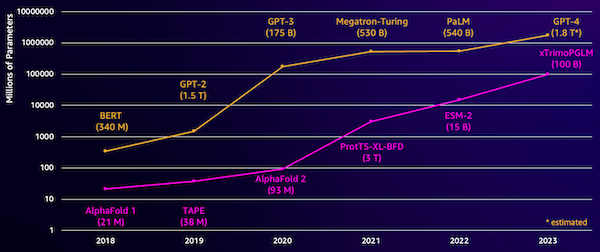
長いトレーニング時間と大規模なインスタンスはコストが高くつくため、多くの研究者にとってこの作業は手の届かないものになる可能性があります。たとえば、2023 年には、 研究チーム 100 個の A768 GPU で 100 億パラメータの pLM を 164 日間トレーニングする方法について説明しました。幸いなことに、多くの場合、既存の pLM を特定のタスクに適応させることで、時間とリソースを節約できます。このテクニックはと呼ばれます 微調整、また、他の種類の言語モデリングから高度なツールを借用することもできます。
ソリューションの概要
この投稿で取り上げる具体的な問題は次のとおりです。 細胞内局在: タンパク質の配列が与えられた場合、それが細胞の外側 (細胞膜) に存在するのか、細胞の内側に存在するのかを予測できるモデルを構築できますか?これは、その機能を理解し、それが適切な薬剤標的となるかどうかを理解するのに役立つ重要な情報です。
まず、次を使用して公開データセットをダウンロードします。 Amazon SageMakerスタジオ。次に、SageMaker を使用して、効率的なトレーニング方法を使用して ESM-2 タンパク質言語モデルを微調整します。最後に、モデルをリアルタイム推論エンドポイントとしてデプロイし、それを使用していくつかの既知のタンパク質をテストします。次の図は、このワークフローを示しています。

次のセクションでは、トレーニング データを準備し、トレーニング スクリプトを作成し、SageMaker トレーニング ジョブを実行する手順を説明します。この投稿で紹介されているコードはすべて、次の場所で入手できます。 GitHubの.
トレーニングデータを準備する
の一部を使用しております。 DeepLoc-2 データセットこれには、実験的に位置が決定された数千の SwissProt タンパク質が含まれています。 100 ~ 512 アミノ酸の間の高品質な配列をフィルターします。
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
次に、シーケンスをトークン化し、トレーニング セットと評価セットに分割します。
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
最後に、処理されたトレーニング データと評価データを次の場所にアップロードします。 Amazon シンプル ストレージ サービス (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)トレーニングスクリプトを作成する
SageMaker スクリプトモード を使用すると、AWS が管理する最適化された機械学習 (ML) フレームワーク コンテナーでカスタム トレーニング コードを実行できます。この例では、 テキスト分類用の既存のスクリプト ハグフェイスより。これにより、トレーニング作業の効率を向上させるためのいくつかの方法を試すことができます。
方法 1: 加重トレーニング クラス
多くの生物学的データセットと同様に、DeepLoc データは不均一に分布しています。これは、膜タンパク質と非膜タンパク質の数が同じではないことを意味します。データをリサンプリングして、大多数のクラスからのレコードを破棄することができます。ただし、これによりトレーニング データの総量が減少し、精度が損なわれる可能性があります。代わりに、トレーニング ジョブ中にクラスの重みを計算し、それを使用して損失を調整します。
トレーニング スクリプトでは、 Trainer からのクラス transformers また、 WeightedTrainer クロスエントロピー損失を計算するときにクラスの重みを考慮するクラス。これは、モデル内のバイアスを防ぐのに役立ちます。
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else loss方法 2: 勾配の累積
勾配累積は、モデルがより大きなバッチ サイズでトレーニングをシミュレートできるようにするトレーニング手法です。通常、バッチ サイズ (1 つのトレーニング ステップで勾配を計算するために使用されるサンプルの数) は、GPU メモリ容量によって制限されます。勾配の累積では、モデルは最初に小さなバッチの勾配を計算します。次に、モデルの重みをすぐに更新するのではなく、複数の小さなバッチにわたって勾配が蓄積されます。累積された勾配が目標のより大きなバッチ サイズに等しい場合、最適化ステップが実行されてモデルが更新されます。これにより、GPU メモリ制限を超えることなく、より大きなバッチでモデルを効果的にトレーニングできるようになります。
ただし、より小さなバッチの前方パスと後方パスには追加の計算が必要です。勾配累積によってバッチ サイズが増加すると、特に累積ステップが多すぎる場合、トレーニングが遅くなる可能性があります。目的は、GPU 使用率を最大化しながら、追加の勾配計算ステップが多すぎることによる過度の速度低下を回避することです。
方法 3: 勾配チェックポイント設定
勾配チェックポイントは、計算時間を適切に保ちながら、トレーニング中に必要なメモリを削減する手法です。大規模なニューラル ネットワークは、バックワード パス中に勾配を計算するために、フォワード パスからのすべての中間値を保存する必要があるため、大量のメモリを消費します。これにより、メモリの問題が発生する可能性があります。解決策の 1 つは、これらの中間値を保存しないことですが、その場合、バックワード パス中に再計算する必要があり、時間がかかります。
勾配チェックポイントは、バランスの取れたアプローチを提供します。と呼ばれる中間値の一部のみを保存します。 チェックポイント、必要に応じてその他を再計算します。したがって、すべてを保存するよりもメモリの使用量が少なくなりますが、すべてを再計算するよりも計算量が少なくなります。勾配チェックポイントにより、どのアクティベーションをチェックポイントにするかを戦略的に選択することで、管理可能なメモリ使用量と計算時間で大規模なニューラル ネットワークをトレーニングできるようになります。この重要な手法により、メモリ制限に遭遇する非常に大規模なモデルのトレーニングが可能になります。
トレーニング スクリプトでは、必要なパラメーターを TrainingArguments オブジェクト:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)方法 4: LLM の低ランク適応
ESM-2 のような大規模な言語モデルには、トレーニングと実行にコストがかかる数十億のパラメーターが含まれる場合があります。 研究者 これらの巨大なモデルをより効率的に微調整するために、Low-Rank Adaptation (LoRA) と呼ばれるトレーニング方法を開発しました。
LoRA の背後にある重要な考え方は、特定のタスクに合わせてモデルを微調整するときに、元のパラメーターをすべて更新する必要がないということです。代わりに、LoRA は、入力と出力を変換する新しい小さな行列をモデルに追加します。微調整中にこれらの小さな行列のみが更新されるため、より高速でメモリの使用量も少なくなります。元のモデルのパラメータは固定されたままになります。
LoRA で微調整した後、適応された小さな行列を元のモデルにマージして戻すことができます。または、以前のタスクを忘れることなく、他のタスク用にモデルをすばやく微調整したい場合は、それらを分離しておくこともできます。全体として、LoRA を使用すると、通常の数分の一のコストで LLM を新しいタスクに効率的に適応させることができます。
トレーニング スクリプトでは、次を使用して LoRA を設定します。 PEFT ハグフェイスのライブラリ:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)SageMaker トレーニング ジョブを送信する
トレーニング スクリプトを定義したら、SageMaker トレーニング ジョブを設定して送信できます。まず、ハイパーパラメータを指定します。
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}次に、トレーニング ログから取得するメトリクスを定義します。
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]最後に、Hugging Face 推定器を定義し、ml.g5.2xlarge インスタンス タイプでのトレーニング用に送信します。これは、多くの AWS リージョンで広く利用できる、コスト効率の高いインスタンス タイプです。
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)次の表は、これまでに説明したさまざまなトレーニング方法と、それらがジョブの実行時間、精度、GPU メモリ要件に及ぼす影響を比較したものです。
| 請求可能時間 (分) | 評価精度 | 最大 GPU メモリ使用量 (GB) | |
| ベースモデル | 28 | 0.91 | 22.6 |
| ベース + GA | 21 | 0.90 | 17.8 |
| ベース + GC | 29 | 0.91 | 10.2 |
| ベース + LoRA | 23 | 0.90 | 18.6 |
いずれの方法でも評価精度の高いモデルが得られました。 LoRA と勾配アクティベーションを使用すると、実行時間 (およびコスト) がそれぞれ 18% と 25% 減少しました。勾配チェックポイントを使用すると、最大 GPU メモリ使用量が 55% 減少しました。制約 (コスト、時間、ハードウェア) によっては、これらのアプローチの XNUMX つが他のアプローチよりも合理的な場合があります。
これらの方法はそれぞれ単独ではうまく機能しますが、組み合わせて使用するとどうなるでしょうか?次の表に結果をまとめます。
| 請求可能時間 (分) | 評価精度 | 最大 GPU メモリ使用量 (GB) | |
| すべての方法 | 12 | 0.80 | 3.3 |
この場合、精度が 12% 低下することがわかります。ただし、実行時間は 57%、GPU メモリの使用量は 85% 削減されました。これは大幅な削減であり、コスト効率の高いさまざまなインスタンス タイプでトレーニングできるようになります。
クリーンアップ
自分の AWS アカウントで作業を進めている場合は、追加料金が発生しないように、作成したリアルタイム推論エンドポイントとデータを削除してください。
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()まとめ
この投稿では、科学的に関連するタスクのために ESM-2 のようなタンパク質言語モデルを効率的に微調整する方法を実証しました。 Transformers および PEFT ライブラリを使用して pLMS をトレーニングする方法の詳細については、投稿を参照してください。 タンパク質を使ったディープラーニング & ESMBind (ESMB): タンパク質結合部位予測のための ESM-2 の低ランク適応 ハグフェイスのブログで。機械学習を使用してタンパク質の特性を予測するその他の例も、次のページで見つけることができます。 AWS での素晴らしいタンパク質分析 GitHubリポジトリ
著者について
 ブライアンロイヤル アマゾン ウェブ サービスのグローバル ヘルスケアおよびライフ サイエンス チームのシニア AI/ML ソリューション アーキテクトです。 バイオテクノロジーと機械学習の分野で 17 年以上の経験があり、顧客がゲノムとプロテオミクスの課題を解決できるよう支援することに情熱を注いでいます。 余暇には、友人や家族と一緒に料理や食事を楽しんでいます。
ブライアンロイヤル アマゾン ウェブ サービスのグローバル ヘルスケアおよびライフ サイエンス チームのシニア AI/ML ソリューション アーキテクトです。 バイオテクノロジーと機械学習の分野で 17 年以上の経験があり、顧客がゲノムとプロテオミクスの課題を解決できるよう支援することに情熱を注いでいます。 余暇には、友人や家族と一緒に料理や食事を楽しんでいます。
- SEO を活用したコンテンツと PR 配信。 今日増幅されます。
- PlatoData.Network 垂直生成 Ai。 自分自身に力を与えましょう。 こちらからアクセスしてください。
- プラトアイストリーム。 Web3 インテリジェンス。 知識増幅。 こちらからアクセスしてください。
- プラトンESG。 カーボン、 クリーンテック、 エネルギー、 環境、 太陽、 廃棄物管理。 こちらからアクセスしてください。
- プラトンヘルス。 バイオテクノロジーと臨床試験のインテリジェンス。 こちらからアクセスしてください。
- 情報源: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



