In diesem Beitrag zeigen wir, wie man ein hochmodernes Protein-Sprachmodell (pLM) effizient verfeinert, um die subzelluläre Lokalisierung von Proteinen vorherzusagen Amazon Sage Maker.
Proteine sind die molekularen Maschinen des Körpers, die für alles verantwortlich sind, von der Bewegung Ihrer Muskeln bis hin zur Reaktion auf Infektionen. Trotz dieser Vielfalt bestehen alle Proteine aus sich wiederholenden Molekülketten, die Aminosäuren genannt werden. Das menschliche Genom kodiert für 20 Standardaminosäuren, jede mit einer leicht unterschiedlichen chemischen Struktur. Diese können durch Buchstaben des Alphabets dargestellt werden, was uns dann ermöglicht, Proteine als Textzeichenfolge zu analysieren und zu erforschen. Die enorme mögliche Anzahl an Proteinsequenzen und -strukturen verleiht Proteinen ihre vielfältigen Einsatzmöglichkeiten.
Auch bei der Arzneimittelentwicklung spielen Proteine eine Schlüsselrolle, als potenzielle Angriffspunkte, aber auch als Therapeutika. Wie aus der folgenden Tabelle hervorgeht, waren viele der meistverkauften Medikamente im Jahr 2022 entweder Proteine (insbesondere Antikörper) oder andere Moleküle wie mRNA, die im Körper in Proteine übersetzt wurden. Aus diesem Grund müssen viele Biowissenschaftsforscher Fragen zu Proteinen schneller, kostengünstiger und genauer beantworten.
| Name und Vorname | Hersteller | Weltweiter Umsatz 2022 (Milliarden US-Dollar) | Indikationen |
| comirnaty | Pfizer / BioNTech | $40.8 | COVID-19 |
| Spikevax | modern | $21.8 | COVID-19 |
| Humira | AbbVie | $21.6 | Arthritis, Morbus Crohn und andere |
| Keytruda | Merck | $21.0 | Verschiedene Krebsarten |
Datenquelle: Urquhart, L. Top-Unternehmen und Medikamente nach Umsatz im Jahr 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Da wir Proteine als Zeichenfolgen darstellen können, können wir sie mit Techniken analysieren, die ursprünglich für die geschriebene Sprache entwickelt wurden. Dazu gehören große Sprachmodelle (LLMs), die auf riesigen Datensätzen vorab trainiert wurden und dann für bestimmte Aufgaben wie Textzusammenfassung oder Chatbots angepasst werden können. In ähnlicher Weise werden pLMs mithilfe von unmarkiertem, selbstüberwachtem Lernen auf großen Proteinsequenzdatenbanken vorab trainiert. Wir können sie anpassen, um Dinge wie die 3D-Struktur eines Proteins vorherzusagen oder wie es mit anderen Molekülen interagieren kann. Forscher haben pLMs sogar verwendet, um neuartige Proteine von Grund auf zu entwickeln. Diese Tools ersetzen nicht das wissenschaftliche Fachwissen des Menschen, haben aber das Potenzial, die präklinische Entwicklung und das Studiendesign zu beschleunigen.
Eine Herausforderung bei diesen Modellen ist ihre Größe. Sowohl LLMs als auch pLMs sind in den letzten Jahren um Größenordnungen gewachsen, wie die folgende Abbildung zeigt. Das bedeutet, dass es lange dauern kann, sie auf eine ausreichende Genauigkeit zu trainieren. Dies bedeutet auch, dass Sie Hardware, insbesondere GPUs, mit großer Speicherkapazität zum Speichern der Modellparameter verwenden müssen.
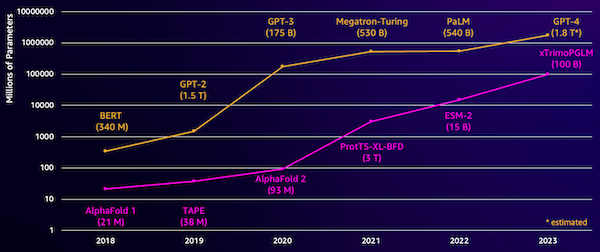
Lange Trainingszeiten und große Instanzen bedeuten hohe Kosten, die diese Arbeit für viele Forscher unerschwinglich machen können. Zum Beispiel im Jahr 2023, a Forschungsgruppe beschrieb das Training eines 100-Milliarden-Parameter-pLM auf 768 A100-GPUs für 164 Tage! Glücklicherweise können wir in vielen Fällen Zeit und Ressourcen sparen, indem wir ein vorhandenes pLM an unsere spezifische Aufgabe anpassen. Diese Technik heißt Feintuningund ermöglicht es uns auch, erweiterte Tools aus anderen Arten der Sprachmodellierung auszuleihen.
Lösungsüberblick
Das spezifische Problem, das wir in diesem Beitrag ansprechen, ist subzelluläre Lokalisation: Können wir anhand einer gegebenen Proteinsequenz ein Modell erstellen, das vorhersagen kann, ob es sich außerhalb (Zellmembran) oder innerhalb einer Zelle befindet? Dies ist eine wichtige Information, die uns helfen kann, die Funktion zu verstehen und herauszufinden, ob es sich um ein gutes Angriffsziel für Medikamente handelt.
Wir beginnen mit dem Herunterladen eines öffentlichen Datensatzes mit Amazon SageMaker-Studio. Dann verwenden wir SageMaker, um das ESM-2-Proteinsprachenmodell mithilfe einer effizienten Trainingsmethode zu verfeinern. Schließlich stellen wir das Modell als Echtzeit-Inferenzendpunkt bereit und verwenden es zum Testen einiger bekannter Proteine. Das folgende Diagramm veranschaulicht diesen Arbeitsablauf.

In den folgenden Abschnitten gehen wir die Schritte durch, um Ihre Trainingsdaten vorzubereiten, ein Trainingsskript zu erstellen und einen SageMaker-Trainingsjob auszuführen. Der gesamte in diesem Beitrag vorgestellte Code ist auf verfügbar GitHub.
Bereiten Sie die Trainingsdaten vor
Wir nutzen einen Teil davon DeepLoc-2-Datensatz, das mehrere tausend SwissProt-Proteine mit experimentell bestimmten Standorten enthält. Wir filtern nach hochwertigen Sequenzen zwischen 100 und 512 Aminosäuren:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Als nächstes tokenisieren wir die Sequenzen und teilen sie in Trainings- und Bewertungssätze auf:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Abschließend laden wir die verarbeiteten Trainings- und Bewertungsdaten auf hoch Amazon Simple Storage-Service (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Erstellen Sie ein Trainingsskript
SageMaker-Skriptmodus ermöglicht Ihnen die Ausführung Ihres benutzerdefinierten Trainingscodes in optimierten Framework-Containern für maschinelles Lernen (ML), die von AWS verwaltet werden. Für dieses Beispiel passen wir an an vorhandenes Skript zur Textklassifizierung von Hugging Face. Dies ermöglicht es uns, verschiedene Methoden auszuprobieren, um die Effizienz unserer Schulungsarbeit zu verbessern.
Methode 1: Gewichteter Trainingskurs
Wie viele biologische Datensätze sind auch die DeepLoc-Daten ungleichmäßig verteilt, was bedeutet, dass es nicht die gleiche Anzahl an Membran- und Nichtmembranproteinen gibt. Wir könnten unsere Daten erneut abtasten und Datensätze aus der Mehrheitsklasse verwerfen. Dies würde jedoch die gesamten Trainingsdaten reduzieren und möglicherweise unsere Genauigkeit beeinträchtigen. Stattdessen berechnen wir die Klassengewichte während des Trainingsjobs und verwenden sie zur Anpassung des Verlusts.
In unserem Trainingsskript unterteilen wir die Trainer Klasse von transformers mit WeightedTrainer Klasse, die Klassengewichte bei der Berechnung des Kreuzentropieverlusts berücksichtigt. Dies trägt dazu bei, Verzerrungen in unserem Modell zu vermeiden:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMethode 2: Gradientenakkumulation
Die Gradientenakkumulation ist eine Trainingstechnik, die es Modellen ermöglicht, das Training für größere Batchgrößen zu simulieren. Typischerweise ist die Batch-Größe (die Anzahl der Proben, die zur Berechnung des Gradienten in einem Trainingsschritt verwendet werden) durch die GPU-Speicherkapazität begrenzt. Bei der Gradientenakkumulation berechnet das Modell zunächst Gradienten für kleinere Chargen. Anstatt die Modellgewichte sofort zu aktualisieren, werden die Gradienten dann über mehrere kleine Chargen akkumuliert. Wenn die akkumulierten Gradienten der angestrebten größeren Chargengröße entsprechen, wird der Optimierungsschritt durchgeführt, um das Modell zu aktualisieren. Dadurch können Modelle mit effektiv größeren Batches trainieren, ohne das GPU-Speicherlimit zu überschreiten.
Für kleinere Vorwärts- und Rückwärtsdurchläufe sind jedoch zusätzliche Berechnungen erforderlich. Erhöhte Batchgrößen durch Gradientenakkumulation können das Training verlangsamen, insbesondere wenn zu viele Akkumulationsschritte verwendet werden. Ziel ist es, die GPU-Auslastung zu maximieren, aber übermäßige Verlangsamungen durch zu viele zusätzliche Gradientenberechnungsschritte zu vermeiden.
Methode 3: Gradienten-Checkpointing
Gradient Checkpointing ist eine Technik, die den während des Trainings benötigten Speicher reduziert und gleichzeitig die Rechenzeit angemessen hält. Große neuronale Netze beanspruchen viel Speicher, da sie alle Zwischenwerte des Vorwärtsdurchlaufs speichern müssen, um die Gradienten beim Rückwärtsdurchlauf zu berechnen. Dies kann zu Speicherproblemen führen. Eine Lösung besteht darin, diese Zwischenwerte nicht zu speichern, sie müssen dann aber beim Rückwärtsdurchlauf neu berechnet werden, was viel Zeit in Anspruch nimmt.
Gradient Checkpointing bietet einen ausgewogenen Ansatz. Es speichert nur einige der Zwischenwerte, genannt Checkpointsund berechnet die anderen nach Bedarf neu. Daher verbraucht es weniger Speicher als das Speichern von allem, aber auch weniger Rechenaufwand als das Neuberechnen von allem. Durch die strategische Auswahl der zu prüfenden Aktivierungen ermöglicht Gradient Checkpointing das Training großer neuronaler Netze mit überschaubarem Speicherverbrauch und Rechenzeit. Diese wichtige Technik macht es möglich, sehr große Modelle zu trainieren, die andernfalls an Speicherbeschränkungen stoßen würden.
In unserem Trainingsskript aktivieren wir die Gradientenaktivierung und das Checkpointing, indem wir die erforderlichen Parameter hinzufügen TrainingArguments Objekt:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Methode 4: Low-Rank-Anpassung von LLMs
Große Sprachmodelle wie ESM-2 können Milliarden von Parametern enthalten, deren Training und Ausführung teuer ist. Forscher hat eine Trainingsmethode namens Low-Rank Adaptation (LoRA) entwickelt, um die Feinabstimmung dieser riesigen Modelle effizienter zu gestalten.
Der Schlüsselgedanke hinter LoRA besteht darin, dass Sie bei der Feinabstimmung eines Modells für eine bestimmte Aufgabe nicht alle ursprünglichen Parameter aktualisieren müssen. Stattdessen fügt LoRA dem Modell neue kleinere Matrizen hinzu, die die Ein- und Ausgänge transformieren. Bei der Feinabstimmung werden nur diese kleineren Matrizen aktualisiert, was viel schneller ist und weniger Speicher verbraucht. Die ursprünglichen Modellparameter bleiben eingefroren.
Nach der Feinabstimmung mit LoRA können Sie die kleinen angepassten Matrizen wieder in das Originalmodell einbinden. Oder Sie können sie getrennt halten, wenn Sie das Modell schnell für andere Aufgaben optimieren möchten, ohne vorherige zu vergessen. Insgesamt ermöglicht LoRA eine effiziente Anpassung von LLMs an neue Aufgaben zu einem Bruchteil der üblichen Kosten.
In unserem Trainingsskript konfigurieren wir LoRA mithilfe von PEFT Bibliothek von Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Senden Sie einen SageMaker-Schulungsauftrag
Nachdem Sie Ihr Trainingsskript definiert haben, können Sie einen SageMaker-Trainingsjob konfigurieren und übermitteln. Geben Sie zunächst die Hyperparameter an:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Definieren Sie als Nächstes, welche Metriken aus den Trainingsprotokollen erfasst werden sollen:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Definieren Sie abschließend einen Hugging Face-Schätzer und senden Sie ihn zum Training für einen ml.g5.2xlarge-Instanztyp. Dies ist ein kostengünstiger Instanztyp, der in vielen AWS-Regionen weit verbreitet ist:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)In der folgenden Tabelle werden die verschiedenen von uns besprochenen Trainingsmethoden und ihre Auswirkungen auf die Laufzeit, Genauigkeit und GPU-Speicheranforderungen unseres Jobs verglichen.
| Konfiguration | Abrechenbare Zeit (Min.) | Bewertungsgenauigkeit | Maximale GPU-Speichernutzung (GB) |
| Basismodell | 28 | 0.91 | 22.6 |
| Basis + GA | 21 | 0.90 | 17.8 |
| Basis + GC | 29 | 0.91 | 10.2 |
| Basis + LoRA | 23 | 0.90 | 18.6 |
Alle Methoden führten zu Modellen mit hoher Bewertungsgenauigkeit. Der Einsatz von LoRA und der Gradientenaktivierung verringerte die Laufzeit (und die Kosten) um 18 % bzw. 25 %. Die Verwendung von Gradient Checkpointing verringerte die maximale GPU-Speichernutzung um 55 %. Abhängig von Ihren Einschränkungen (Kosten, Zeit, Hardware) kann einer dieser Ansätze sinnvoller sein als ein anderer.
Jede dieser Methoden funktioniert für sich genommen gut, aber was passiert, wenn wir sie in Kombination verwenden? Die folgende Tabelle fasst die Ergebnisse zusammen.
| Konfiguration | Abrechenbare Zeit (Min.) | Bewertungsgenauigkeit | Maximale GPU-Speichernutzung (GB) |
| Alle Methoden | 12 | 0.80 | 3.3 |
In diesem Fall sehen wir eine Verringerung der Genauigkeit um 12 %. Allerdings haben wir die Laufzeit um 57 % und die GPU-Speichernutzung um 85 % reduziert! Dies ist eine enorme Reduzierung, die es uns ermöglicht, auf einer breiten Palette kostengünstiger Instanztypen zu trainieren.
Aufräumen
Wenn Sie mit Ihrem eigenen AWS-Konto mitverfolgen, löschen Sie alle von Ihnen erstellten Echtzeit-Inferenzendpunkte und Daten, um weitere Kosten zu vermeiden.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Zusammenfassung
In diesem Beitrag haben wir gezeigt, wie Proteinsprachmodelle wie ESM-2 effizient für eine wissenschaftlich relevante Aufgabe optimiert werden können. Weitere Informationen zur Verwendung der Transformers- und PEFT-Bibliotheken zum Trainieren von pLMS finden Sie in den Beiträgen Deep Learning mit Proteinen und ESMBind (ESMB): Low-Rank-Adaption von ESM-2 zur Vorhersage der Proteinbindungsstelle auf dem Hugging Face-Blog. Weitere Beispiele für die Verwendung von maschinellem Lernen zur Vorhersage von Proteineigenschaften finden Sie im Fantastische Proteinanalyse auf AWS GitHub-Repository.
Über den Autor
 Brian Treue ist Senior AI/ML Solutions Architect im Team Global Healthcare and Life Sciences bei Amazon Web Services. Er verfügt über mehr als 17 Jahre Erfahrung in Biotechnologie und maschinellem Lernen und unterstützt Kunden leidenschaftlich gerne bei der Lösung genomischer und proteomischer Herausforderungen. In seiner Freizeit kocht und isst er gerne mit seinen Freunden und seiner Familie.
Brian Treue ist Senior AI/ML Solutions Architect im Team Global Healthcare and Life Sciences bei Amazon Web Services. Er verfügt über mehr als 17 Jahre Erfahrung in Biotechnologie und maschinellem Lernen und unterstützt Kunden leidenschaftlich gerne bei der Lösung genomischer und proteomischer Herausforderungen. In seiner Freizeit kocht und isst er gerne mit seinen Freunden und seiner Familie.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- Quelle: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



