Bu yazıda, protein hücre altı lokalizasyonunu tahmin etmek için son teknoloji ürünü bir protein dil modeline (pLM) verimli bir şekilde ince ayar yapmanın nasıl yapılacağını gösteriyoruz. Amazon Adaçayı Yapıcı.
Proteinler vücudun moleküler makineleridir ve kaslarınızı hareket ettirmekten enfeksiyonlara yanıt vermeye kadar her şeyden sorumludur. Bu çeşitliliğe rağmen tüm proteinler, amino asit adı verilen moleküllerin tekrarlanan zincirlerinden oluşur. İnsan genomu, her biri biraz farklı kimyasal yapıya sahip 20 standart amino asidi kodlar. Bunlar alfabenin harfleriyle temsil edilebilir, bu da proteinleri bir metin dizisi olarak analiz etmemize ve keşfetmemize olanak tanır. Protein dizilerinin ve yapılarının mümkün olan çok büyük sayısı, proteinlere geniş kullanım alanı sağlayan şeydir.
Proteinler aynı zamanda ilaç geliştirmede potansiyel hedefler olarak ve aynı zamanda terapötik olarak da önemli bir rol oynamaktadır. Aşağıdaki tabloda gösterildiği gibi, 2022'de en çok satan ilaçların çoğu ya proteinler (özellikle antikorlar) ya da vücutta proteinlere çevrilen mRNA gibi diğer moleküllerdi. Bu nedenle birçok yaşam bilimi araştırmacısının proteinlerle ilgili soruları daha hızlı, daha ucuz ve daha doğru yanıtlaması gerekiyor.
| Name | Üretici firma | 2022 Küresel Satışlar (milyar ABD Doları) | Endikasyonları |
| Komünite | Pfizer / Biontech | $40.8 | Covid-19 |
| Sivrisinek | modern | $21.8 | Covid-19 |
| Humira | AbbVie | $21.6 | Artrit, Crohn hastalığı ve diğerleri |
| Keytruda | Merck | $21.0 | Çeşitli kanserler |
Veri kaynağı: Urquhart, L. 2022'de satışlara göre en iyi şirketler ve ilaçlar. Nature Reviews İlaç Keşfi 22, 260–260 (2023).
Proteinleri karakter dizileri olarak temsil edebildiğimiz için, onları orijinal olarak yazılı dil için geliştirilen teknikleri kullanarak analiz edebiliriz. Buna, büyük veri kümeleri üzerinde önceden eğitilmiş ve daha sonra metin özetleme veya sohbet robotları gibi belirli görevlere uyarlanabilen büyük dil modelleri (LLM'ler) dahildir. Benzer şekilde pLM'ler, etiketlenmemiş, kendi kendini denetleyen öğrenme kullanılarak büyük protein dizisi veritabanlarında önceden eğitilir. Bunları bir proteinin 3 boyutlu yapısı veya diğer moleküllerle nasıl etkileşime girebileceği gibi şeyleri tahmin etmek için uyarlayabiliriz. Araştırmacılar sıfırdan yeni proteinler tasarlamak için pLM'leri bile kullandılar. Bu araçlar insanın bilimsel uzmanlığının yerini almaz ancak klinik öncesi geliştirmeyi ve deneme tasarımını hızlandırma potansiyeline sahiptirler.
Bu modellerdeki zorluklardan biri boyutlarıdır. Aşağıdaki şekilde gösterildiği gibi, hem LLM'ler hem de pLM'ler son birkaç yılda büyük miktarlarda büyüdü. Bu, onları yeterli doğrulukta eğitmenin uzun zaman alabileceği anlamına gelir. Bu aynı zamanda model parametrelerini depolamak için büyük miktarda belleğe sahip donanım, özellikle GPU'lar kullanmanız gerektiği anlamına da gelir.
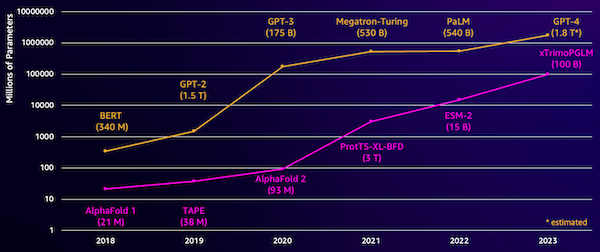
Uzun eğitim süreleri artı büyük örnekler yüksek maliyet anlamına gelir ve bu da bu çalışmayı birçok araştırmacının erişemeyeceği bir yere koyabilir. Örneğin 2023 yılında araştırma ekibi 100 gün boyunca 768 A100 GPU'da 164 milyar parametreli bir pLM'nin eğitilmesini anlattı! Neyse ki çoğu durumda mevcut bir pLM'yi özel görevimize uyarlayarak zamandan ve kaynaklardan tasarruf edebiliriz. Bu tekniğe denir ince ayarve aynı zamanda diğer dil modelleme türlerinden gelişmiş araçları ödünç almamıza da olanak tanır.
Çözüme genel bakış
Bu yazıda ele aldığımız spesifik sorun hücre altı lokalizasyonu: Bir protein dizisi verildiğinde, onun hücrenin dışında mı (hücre zarı) yoksa içinde mi yaşadığını tahmin edebilecek bir model oluşturabilir miyiz? Bu, işlevi ve iyi bir ilaç hedefi olup olmayacağını anlamamıza yardımcı olabilecek önemli bir bilgi parçasıdır.
Aşağıdakileri kullanarak genel bir veri kümesini indirerek başlıyoruz: Amazon SageMaker Stüdyosu. Daha sonra etkili bir eğitim yöntemi kullanarak ESM-2 protein dili modeline ince ayar yapmak için SageMaker'ı kullanıyoruz. Son olarak modeli gerçek zamanlı bir çıkarım uç noktası olarak dağıtıyoruz ve onu bilinen bazı proteinleri test etmek için kullanıyoruz. Aşağıdaki şemada bu iş akışı gösterilmektedir.

Aşağıdaki bölümlerde eğitim verilerinizi hazırlamak, bir eğitim komut dosyası oluşturmak ve bir SageMaker eğitim işi yürütmek için gerekli adımları inceleyeceğiz. Bu yazıda yer alan kodun tamamı şu adreste mevcuttur: GitHub.
Eğitim verilerini hazırlayın
Bir kısmını kullanıyoruz DeepLoc-2 veri kümesiDeneysel olarak belirlenmiş konumlara sahip birkaç bin SwissProt proteini içerir. 100-512 amino asit arasındaki yüksek kaliteli dizileri filtreliyoruz:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Daha sonra dizileri tokenize edip eğitim ve değerlendirme setlerine ayırıyoruz:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Son olarak işlenen eğitim ve değerlendirme verilerini şuraya yüklüyoruz: Amazon Basit Depolama Hizmeti (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Bir eğitim betiği oluşturun
SageMaker komut dosyası modu özel eğitim kodunuzu AWS tarafından yönetilen optimize edilmiş makine öğrenimi (ML) çerçeve kapsayıcılarında çalıştırmanıza olanak tanır. Bu örnek için, metin sınıflandırması için mevcut komut dosyası Hugging Face'den. Bu, eğitim işimizin verimliliğini artırmak için çeşitli yöntemler denememize olanak tanır.
Yöntem 1: Ağırlıklı eğitim sınıfı
Birçok biyolojik veri seti gibi DeepLoc verileri de eşit olmayan bir şekilde dağılmıştır; bu, eşit sayıda membran ve membran dışı protein olmadığı anlamına gelir. Verilerimizi yeniden örnekleyebilir ve çoğunluk sınıfındaki kayıtları atabiliriz. Ancak bu, toplam eğitim verilerini azaltacak ve potansiyel olarak doğruluğumuza zarar verecektir. Bunun yerine eğitim işi sırasında sınıf ağırlıklarını hesaplıyoruz ve bunları kaybı ayarlamak için kullanıyoruz.
Eğitim senaryomuzda, aşağıdakileri alt sınıflara ayırıyoruz: Trainer sınıftan transformers Birlikte WeightedTrainer Çapraz entropi kaybını hesaplarken sınıf ağırlıklarını dikkate alan sınıf. Bu, modelimizde önyargının önlenmesine yardımcı olur:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossYöntem 2: Gradyan birikimi
Gradyan birikimi, modellerin daha büyük parti boyutlarında eğitimi simüle etmesine olanak tanıyan bir eğitim tekniğidir. Tipik olarak parti boyutu (bir eğitim adımında eğimi hesaplamak için kullanılan örnek sayısı) GPU bellek kapasitesi ile sınırlıdır. Gradyan birikimiyle model, öncelikle daha küçük gruplardaki gradyanları hesaplar. Daha sonra model ağırlıklarını hemen güncellemek yerine degradeler birden fazla küçük grup halinde toplanır. Birikmiş degradeler hedefin daha büyük toplu boyutuna eşit olduğunda, modeli güncellemek için optimizasyon adımı gerçekleştirilir. Bu, modellerin GPU bellek sınırını aşmadan etkili bir şekilde daha büyük gruplarla eğitilmesine olanak tanır.
Ancak daha küçük toplu ileri ve geri geçişler için ekstra hesaplama gereklidir. Gradyan birikimi yoluyla artan parti boyutları, özellikle çok fazla birikim adımı kullanıldığında eğitimi yavaşlatabilir. Amaç, GPU kullanımını en üst düzeye çıkarmak ancak çok fazla ekstra gradyan hesaplama adımından kaynaklanan aşırı yavaşlamaları önlemektir.
Yöntem 3: Degrade denetim noktası oluşturma
Gradyan kontrol noktası oluşturma, hesaplama süresini makul tutarken eğitim sırasında ihtiyaç duyulan belleği azaltan bir tekniktir. Büyük sinir ağları çok fazla bellek kaplar çünkü geri geçiş sırasındaki gradyanları hesaplamak için ileri geçişteki tüm ara değerleri depolamak zorundadırlar. Bu hafıza sorunlarına neden olabilir. Çözümlerden biri, bu ara değerleri saklamamaktır, ancak daha sonra çok fazla zaman alan geri geçiş sırasında bunların yeniden hesaplanması gerekir.
Gradyan kontrol noktası oluşturma dengeli bir yaklaşım sağlar. Adı verilen ara değerlerden yalnızca bazılarını kaydeder. kontrol noktalarıve diğerlerini gerektiği gibi yeniden hesaplar. Bu nedenle, her şeyi depolamaktan daha az bellek kullanır, aynı zamanda her şeyi yeniden hesaplamaktan daha az hesaplama kullanır. Gradyan kontrol noktası oluşturma, hangi aktivasyonların kontrol noktası olacağını stratejik olarak seçerek, büyük sinir ağlarının yönetilebilir bellek kullanımı ve hesaplama süresi ile eğitilmesine olanak tanır. Bu önemli teknik, aksi takdirde bellek sınırlamalarıyla karşılaşacak çok büyük modellerin eğitilmesini mümkün kılar.
Eğitim senaryomuzda, gerekli parametreleri ekleyerek degrade aktivasyonunu ve kontrol noktası oluşturmayı açıyoruz. TrainingArguments nesne:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Yöntem 4: Yüksek Lisans Derecelerinin Düşük Sıralı Uyarlanması
ESM-2 gibi büyük dil modelleri, eğitilmesi ve çalıştırılması pahalı olan milyarlarca parametre içerebilir. Araştırmacılar bu devasa modellere ince ayar yapmayı daha verimli hale getirmek için Düşük Sıralı Adaptasyon (LoRA) adı verilen bir eğitim yöntemi geliştirdi.
LoRA'nın arkasındaki temel fikir, bir modele belirli bir görev için ince ayar yaparken tüm orijinal parametreleri güncellemenize gerek olmamasıdır. Bunun yerine LoRA, modele girdileri ve çıktıları dönüştüren yeni, daha küçük matrisler ekler. İnce ayar sırasında yalnızca bu daha küçük matrisler güncellenir; bu, çok daha hızlıdır ve daha az bellek kullanır. Orijinal model parametreleri donmuş halde kalır.
LoRA ile ince ayar yaptıktan sonra uyarlanmış küçük matrisleri orijinal modele geri birleştirebilirsiniz. Veya önceki görevleri unutmadan diğer görevler için modele hızlı bir şekilde ince ayar yapmak istiyorsanız bunları ayrı tutabilirsiniz. Genel olarak LoRA, LLM'lerin normal maliyetin çok altında bir maliyetle yeni görevlere verimli bir şekilde uyarlanmasına olanak tanır.
Eğitim senaryomuzda LoRA'yı aşağıdakileri kullanarak yapılandırıyoruz: PEFT Hugging Face'den kütüphane:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Bir SageMaker eğitim işi gönderin
Eğitim komut dosyanızı tanımladıktan sonra bir SageMaker eğitim işini yapılandırabilir ve gönderebilirsiniz. İlk önce hiperparametreleri belirtin:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Daha sonra, eğitim günlüklerinden hangi ölçümlerin yakalanacağını tanımlayın:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Son olarak, bir Hugging Face tahmincisi tanımlayın ve bunu bir ml.g5.2xlarge bulut sunucusu tipi üzerinde eğitim için gönderin. Bu, birçok AWS Bölgesinde yaygın olarak kullanılabilen, uygun maliyetli bir bulut sunucusu türüdür:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)Aşağıdaki tablo, tartıştığımız farklı eğitim yöntemlerini ve bunların işimizin çalışma süresi, doğruluğu ve GPU bellek gereksinimleri üzerindeki etkilerini karşılaştırmaktadır.
| yapılandırma | Faturalandırılabilir Süre (dk) | Değerlendirme Doğruluğu | Maksimum GPU Bellek Kullanımı (GB) |
| Temel Model | 28 | 0.91 | 22.6 |
| Taban + GA | 21 | 0.90 | 17.8 |
| Baz + GC | 29 | 0.91 | 10.2 |
| Temel + LoRA | 23 | 0.90 | 18.6 |
Yöntemlerin tümü yüksek değerlendirme doğruluğuna sahip modeller üretti. LoRA ve degrade aktivasyonunun kullanılması, çalışma süresini (ve maliyetini) sırasıyla %18 ve %25 oranında azalttı. Gradyan kontrol noktası kullanımı, maksimum GPU bellek kullanımını %55 oranında azalttı. Kısıtlamalarınıza (maliyet, zaman, donanım) bağlı olarak bu yaklaşımlardan biri diğerinden daha anlamlı olabilir.
Bu yöntemlerin her biri tek başına iyi performans gösteriyor ancak bunları bir arada kullandığımızda ne olur? Aşağıdaki tablo sonuçları özetlemektedir.
| yapılandırma | Faturalandırılabilir Süre (dk) | Değerlendirme Doğruluğu | Maksimum GPU Bellek Kullanımı (GB) |
| Tüm yöntemler | 12 | 0.80 | 3.3 |
Bu durumda doğrulukta %12 oranında bir azalma görüyoruz. Ancak çalışma süresini %57 ve GPU bellek kullanımını %85 oranında azalttık! Bu, çok çeşitli uygun maliyetli bulut sunucusu türleri üzerinde eğitim almamıza olanak tanıyan çok büyük bir azalmadır.
Temizlemek
Kendi AWS hesabınızdan takip ediyorsanız daha fazla ücretlendirmeyi önlemek için oluşturduğunuz tüm gerçek zamanlı çıkarım uç noktalarını ve verileri silin.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Sonuç
Bu yazıda, bilimsel açıdan anlamlı bir görev için ESM-2 gibi protein dili modellerine verimli bir şekilde ince ayar yapmanın nasıl yapılacağını gösterdik. pLMS'yi eğitmek için Transformers ve PEFT kitaplıklarını kullanma hakkında daha fazla bilgi için gönderilere göz atın Proteinlerle Derin Öğrenme ve ESMBind (ESMB): Protein Bağlanma Bölgesi Tahmini için ESM-2'nin Düşük Sıralı Adaptasyonu Hugging Face blogunda. Ayrıca protein özelliklerini tahmin etmek için makine öğrenimini kullanmanın daha fazla örneğini şurada bulabilirsiniz: AWS'de Müthiş Protein Analizi GitHub deposu.
Yazar Hakkında
 Brian Sadık Amazon Web Services'ta Küresel Sağlık Hizmetleri ve Yaşam Bilimleri ekibinde Kıdemli Yapay Zeka/Makine Öğrenmesi Çözümleri Mimarıdır. Biyoteknoloji ve makine öğreniminde 17 yıldan fazla deneyime sahiptir ve müşterilerin genomik ve proteomik zorlukları çözmelerine yardımcı olma konusunda tutkuludur. Boş zamanlarında arkadaşları ve ailesi ile yemek yapmaktan ve yemek yemekten keyif alıyor.
Brian Sadık Amazon Web Services'ta Küresel Sağlık Hizmetleri ve Yaşam Bilimleri ekibinde Kıdemli Yapay Zeka/Makine Öğrenmesi Çözümleri Mimarıdır. Biyoteknoloji ve makine öğreniminde 17 yıldan fazla deneyime sahiptir ve müşterilerin genomik ve proteomik zorlukları çözmelerine yardımcı olma konusunda tutkuludur. Boş zamanlarında arkadaşları ve ailesi ile yemek yapmaktan ve yemek yemekten keyif alıyor.
- SEO Destekli İçerik ve Halkla İlişkiler Dağıtımı. Bugün Gücünüzü Artırın.
- PlatoData.Network Dikey Üretken Yapay Zeka. Kendine güç ver. Buradan Erişin.
- PlatoAiStream. Web3 Zekası. Bilgi Genişletildi. Buradan Erişin.
- PlatoESG. karbon, temiz teknoloji, Enerji, Çevre, Güneş, Atık Yönetimi. Buradan Erişin.
- PlatoSağlık. Biyoteknoloji ve Klinik Araştırmalar Zekası. Buradan Erişin.
- Kaynak: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



