Neste post, demonstramos como ajustar com eficiência um modelo de linguagem de proteínas (pLM) de última geração para prever a localização subcelular de proteínas usando Amazon Sage Maker.
As proteínas são as máquinas moleculares do corpo, responsáveis por tudo, desde movimentar os músculos até responder a infecções. Apesar desta variedade, todas as proteínas são feitas de cadeias repetidas de moléculas chamadas aminoácidos. O genoma humano codifica 20 aminoácidos padrão, cada um com uma estrutura química ligeiramente diferente. Estas podem ser representadas por letras do alfabeto, o que nos permite analisar e explorar proteínas como uma sequência de texto. O enorme número possível de sequências e estruturas de proteínas é o que dá às proteínas sua ampla variedade de usos.
As proteínas também desempenham um papel fundamental no desenvolvimento de medicamentos, como alvos potenciais, mas também como terapêutica. Conforme mostrado na tabela a seguir, muitos dos medicamentos mais vendidos em 2022 eram proteínas (especialmente anticorpos) ou outras moléculas como o mRNA traduzido em proteínas no corpo. Por causa disso, muitos pesquisadores de ciências biológicas precisam responder perguntas sobre proteínas de forma mais rápida, barata e precisa.
| Nome | Fabricante | Vendas globais em 2022 (US$ bilhões) | Indicações |
| Comunidade | Pfizer / BioNTech | $40.8 | Covid-19 |
| Spikevax | Moderno | $21.8 | Covid-19 |
| Humira | AbbVie | $21.6 | Artrite, doença de Crohn e outras |
| keytruda | Merck | $21.0 | Vários cânceres |
Fonte de dados: Urquhart, L. Principais empresas e medicamentos por vendas em 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Como podemos representar proteínas como sequências de caracteres, podemos analisá-las utilizando técnicas originalmente desenvolvidas para a linguagem escrita. Isto inclui grandes modelos de linguagem (LLMs) pré-treinados em enormes conjuntos de dados, que podem então ser adaptados para tarefas específicas, como resumo de texto ou chatbots. Da mesma forma, os pLMs são pré-treinados em grandes bancos de dados de sequências de proteínas usando aprendizado auto-supervisionado e não rotulado. Podemos adaptá-los para prever coisas como a estrutura 3D de uma proteína ou como ela pode interagir com outras moléculas. Os pesquisadores até usaram pLMs para projetar novas proteínas do zero. Estas ferramentas não substituem o conhecimento científico humano, mas têm o potencial de acelerar o desenvolvimento pré-clínico e o desenho dos ensaios.
Um desafio desses modelos é seu tamanho. Tanto os LLMs quanto os pLMs cresceram ordens de magnitude nos últimos anos, conforme ilustrado na figura a seguir. Isso significa que pode levar muito tempo para treiná-los com precisão suficiente. Isso também significa que você precisa usar hardware, principalmente GPUs, com grande quantidade de memória para armazenar os parâmetros do modelo.
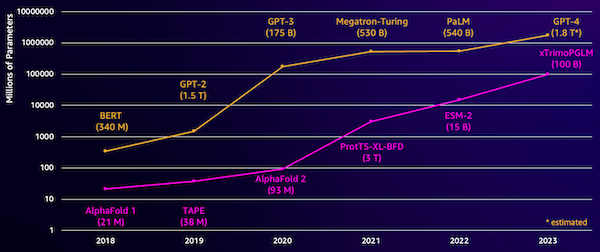
Longos tempos de treinamento, somados a grandes instâncias, equivalem a custos elevados, o que pode colocar este trabalho fora do alcance de muitos pesquisadores. Por exemplo, em 2023, um equipe de pesquisa descreveu o treinamento de um pLM de 100 bilhões de parâmetros em 768 GPUs A100 por 164 dias! Felizmente, em muitos casos podemos poupar tempo e recursos adaptando um pLM existente à nossa tarefa específica. Essa técnica é chamada afinaçãoe também nos permite emprestar ferramentas avançadas de outros tipos de modelagem de linguagem.
Visão geral da solução
O problema específico que abordamos nesta postagem é localização subcelular: Dada uma sequência de proteína, podemos construir um modelo que possa prever se ela vive fora (membrana celular) ou dentro de uma célula? Esta é uma informação importante que pode nos ajudar a entender a função e se ela seria um bom alvo para o medicamento.
Começamos baixando um conjunto de dados público usando Estúdio Amazon SageMaker. Em seguida, usamos o SageMaker para ajustar o modelo de linguagem da proteína ESM-2 usando um método de treinamento eficiente. Finalmente, implantamos o modelo como um endpoint de inferência em tempo real e o usamos para testar algumas proteínas conhecidas. O diagrama a seguir ilustra esse fluxo de trabalho.

Nas seções a seguir, percorremos as etapas para preparar seus dados de treinamento, criar um script de treinamento e executar um trabalho de treinamento do SageMaker. Todo o código apresentado nesta postagem está disponível em GitHub.
Preparar os dados de treinamento
Usamos parte do Conjunto de dados DeepLoc-2, que contém vários milhares de proteínas SwissProt com localizações determinadas experimentalmente. Filtramos sequências de alta qualidade entre 100–512 aminoácidos:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
A seguir, tokenizamos as sequências e as dividimos em conjuntos de treinamento e avaliação:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Finalmente, carregamos os dados de treinamento e avaliação processados para Serviço de armazenamento simples da Amazon (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Crie um roteiro de treinamento
Modo de script SageMaker permite que você execute seu código de treinamento personalizado em contêineres de estrutura de aprendizado de máquina (ML) otimizados gerenciados pela AWS. Para este exemplo, adaptamos um script existente para classificação de texto de Abraçando o Rosto. Isto permite-nos experimentar vários métodos para melhorar a eficiência do nosso trabalho de formação.
Método 1: aula de treinamento ponderado
Como muitos conjuntos de dados biológicos, os dados do DeepLoc são distribuídos de forma desigual, o que significa que não há um número igual de proteínas de membrana e não-membrana. Poderíamos reamostrar nossos dados e descartar os registros da classe majoritária. No entanto, isso reduziria o total de dados de treinamento e potencialmente prejudicaria nossa precisão. Em vez disso, calculamos os pesos das classes durante o trabalho de treinamento e os usamos para ajustar a perda.
Em nosso script de treinamento, subclassificamos o Trainer classe de transformers com uma WeightedTrainer classe que leva em consideração os pesos das classes ao calcular a perda de entropia cruzada. Isso ajuda a evitar preconceitos em nosso modelo:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMétodo 2: acumulação de gradiente
A acumulação de gradiente é uma técnica de treinamento que permite que os modelos simulem o treinamento em lotes maiores. Normalmente, o tamanho do lote (o número de amostras usadas para calcular o gradiente em uma etapa de treinamento) é limitado pela capacidade de memória da GPU. Com o acúmulo de gradiente, o modelo calcula primeiro os gradientes em lotes menores. Então, em vez de atualizar os pesos do modelo imediatamente, os gradientes são acumulados em vários lotes pequenos. Quando os gradientes acumulados são iguais ao tamanho de lote maior alvo, a etapa de otimização é executada para atualizar o modelo. Isso permite que os modelos treinem com lotes efetivamente maiores sem exceder o limite de memória da GPU.
No entanto, é necessário um cálculo extra para as passagens de avanço e retrocesso de lotes menores. O aumento do tamanho dos lotes por meio do acúmulo de gradiente pode retardar o treinamento, especialmente se muitas etapas de acumulação forem usadas. O objetivo é maximizar o uso da GPU, mas evitar lentidão excessiva devido a muitas etapas extras de cálculo de gradiente.
Método 3: ponto de verificação de gradiente
O checkpoint de gradiente é uma técnica que reduz a memória necessária durante o treinamento, mantendo o tempo computacional razoável. Grandes redes neurais ocupam muita memória porque precisam armazenar todos os valores intermediários da passagem para frente para calcular os gradientes durante a passagem para trás. Isso pode causar problemas de memória. Uma solução é não armazenar esses valores intermediários, mas então eles terão que ser recalculados durante o retrocesso, o que leva muito tempo.
O checkpoint de gradiente fornece uma abordagem equilibrada. Ele salva apenas alguns dos valores intermediários, chamados pontos de controlee recalcula os outros conforme necessário. Portanto, usa menos memória do que armazenar tudo, mas também menos computação do que recalcular tudo. Ao selecionar estrategicamente quais ativações devem ser verificadas, o checkpoint de gradiente permite que grandes redes neurais sejam treinadas com uso de memória e tempo de computação gerenciáveis. Esta importante técnica torna viável treinar modelos muito grandes que, de outra forma, teriam limitações de memória.
Em nosso script de treinamento, ativamos a ativação de gradiente e o checkpoint adicionando os parâmetros necessários ao TrainingArguments objeto:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Método 4: Adaptação de baixo nível de LLMs
Grandes modelos de linguagem como o ESM-2 podem conter bilhões de parâmetros cujo treinamento e execução são caros. Pesquisadores desenvolveu um método de treinamento chamado Low-Rank Adaptation (LoRA) para tornar o ajuste fino desses modelos enormes mais eficiente.
A ideia principal por trás do LoRA é que, ao ajustar um modelo para uma tarefa específica, você não precisa atualizar todos os parâmetros originais. Em vez disso, LoRA adiciona novas matrizes menores ao modelo que transformam as entradas e saídas. Somente essas matrizes menores são atualizadas durante o ajuste fino, que é muito mais rápido e utiliza menos memória. Os parâmetros do modelo original permanecem congelados.
Após o ajuste fino com LoRA, você pode mesclar as pequenas matrizes adaptadas de volta ao modelo original. Ou você pode mantê-los separados se quiser ajustar rapidamente o modelo para outras tarefas, sem esquecer as anteriores. No geral, o LoRA permite que os LLMs sejam adaptados de forma eficiente a novas tarefas por uma fração do custo normal.
Em nosso script de treinamento, configuramos LoRA usando o PEFT biblioteca de Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Envie um trabalho de treinamento do SageMaker
Depois de definir seu script de treinamento, você poderá configurar e enviar um trabalho de treinamento do SageMaker. Primeiro, especifique os hiperparâmetros:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}A seguir, defina quais métricas capturar dos logs de treinamento:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Por fim, defina um estimador Hugging Face e envie-o para treinamento em um tipo de instância ml.g5.2xlarge. Este é um tipo de instância econômico que está amplamente disponível em muitas regiões da AWS:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)A tabela a seguir compara os diferentes métodos de treinamento que discutimos e seus efeitos no tempo de execução, na precisão e nos requisitos de memória GPU de nosso trabalho.
| Configuração | Tempo faturável (min) | Precisão da avaliação | Uso máximo de memória GPU (GB) |
| Modelo Base | 28 | 0.91 | 22.6 |
| Base + GA | 21 | 0.90 | 17.8 |
| Base + CG | 29 | 0.91 | 10.2 |
| Base + LoRA | 23 | 0.90 | 18.6 |
Todos os métodos produziram modelos com alta precisão de avaliação. O uso de LoRA e ativação gradiente diminuiu o tempo de execução (e custo) em 18% e 25%, respectivamente. O uso do checkpoint de gradiente diminuiu o uso máximo de memória da GPU em 55%. Dependendo das suas restrições (custo, tempo, hardware), uma dessas abordagens pode fazer mais sentido do que outra.
Cada um desses métodos funciona bem por si só, mas o que acontece quando os usamos em combinação? A tabela a seguir resume os resultados.
| Configuração | Tempo faturável (min) | Precisão da avaliação | Uso máximo de memória GPU (GB) |
| Todos os métodos | 12 | 0.80 | 3.3 |
Neste caso, vemos uma redução de 12% na precisão. No entanto, reduzimos o tempo de execução em 57% e o uso de memória da GPU em 85%! Esta é uma redução enorme que nos permite treinar em uma ampla variedade de tipos de instâncias econômicas.
limpar
Se você estiver acompanhando em sua própria conta da AWS, exclua todos os endpoints e dados de inferência em tempo real que você criou para evitar cobranças adicionais.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Conclusão
Neste post, demonstramos como ajustar com eficiência modelos de linguagem de proteínas como o ESM-2 para uma tarefa cientificamente relevante. Para obter mais informações sobre o uso das bibliotecas Transformers e PEFT para treinar pLMS, confira as postagens Aprendizado profundo com proteínas e ESMBind (ESMB): Adaptação de baixa classificação do ESM-2 para previsão do local de ligação de proteínas no blog Hugging Face. Você também pode encontrar mais exemplos de uso de aprendizado de máquina para prever propriedades de proteínas no Análise de proteína incrível na AWS Repositório GitHub.
Sobre o autor
 Brian Leal é arquiteto sênior de soluções de IA/ML da equipe global de saúde e ciências biológicas da Amazon Web Services. Ele tem mais de 17 anos de experiência em biotecnologia e aprendizado de máquina e é apaixonado por ajudar os clientes a resolver desafios genômicos e proteômicos. Em seu tempo livre, ele gosta de cozinhar e comer com seus amigos e familiares.
Brian Leal é arquiteto sênior de soluções de IA/ML da equipe global de saúde e ciências biológicas da Amazon Web Services. Ele tem mais de 17 anos de experiência em biotecnologia e aprendizado de máquina e é apaixonado por ajudar os clientes a resolver desafios genômicos e proteômicos. Em seu tempo livre, ele gosta de cozinhar e comer com seus amigos e familiares.
- Conteúdo com tecnologia de SEO e distribuição de relações públicas. Seja amplificado hoje.
- PlatoData.Network Gerativa Vertical Ai. Capacite-se. Acesse aqui.
- PlatoAiStream. Inteligência Web3. Conhecimento Amplificado. Acesse aqui.
- PlatãoESG. Carbono Tecnologia Limpa, Energia, Ambiente, Solar, Gestão de resíduos. Acesse aqui.
- PlatoHealth. Inteligência em Biotecnologia e Ensaios Clínicos. Acesse aqui.
- Fonte: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



