倫理
この研究はヘルシンキ宣言の原則に従って実施されました。 ヒトiPS細胞の使用は、慶応義塾大学医学部の倫理委員会によって承認されています(承認番号:20130146および20030092)。
動物
すべての動物実験は、実験動物中央研究所(CIEA、神奈川県)の実験動物の管理と使用に関するガイドに従って行われました。 実験プロトコールは、CIEA Animal Care Committee (許可番号: 11029A) および慶応義塾大学医学部 (東京、日本) (許可番号: 16-096-25) によって承認されました。
細胞培養
ゼノフリーおよびフィーダーフリー条件下で末梢血単核細胞から樹立された組込みフリーのhiPSC株(1210B2および1231A3)は、iPS細胞研究応用センター(CiRA:京都)よりご提供いただきました。19,45。 真皮線維芽細胞から樹立されたhiPSC株201B746 山中伸弥博士のご厚意により提供していただきました。 HiPSC 株 WD39 は真皮線維芽細胞から樹立されました35。 フィーダーフリーの hiPSC は、ヒト組換えラミニンフラグメント iMatrix-511 (Nippi) およびゼノフリー培地 StemFit®AK03 (味の素) を使用して維持されました。19,45。 hiPSC-NS/PC 誘導では、TrypLE Select (Thermo Fisher Scientific) を使用してフィーダーフリー hiPSC を単一細胞に解離し、965000 ウェル低細胞接着プレート (Sumilon PrimeSurface プレート) を使用して再凝集して胚様体 (EB) を形成しました。 M;住友ベークライト)、100nM LDN−193189(Stemgent)および500nM A83−01(Stemgent)を補充した、FGF2を含まないStemFit(登録商標)AS200(味の素)中で5000細胞/ウェル(100μl)の密度で。 培地は毎日交換した。 100日目に、EBを200μg/cm 2 でプレコートした組織培養プレートに付着させた。2 StemFit®AS511 の iMatrix-200。 その後の 7 日間の培養中に、神経ロゼット構造が発達しました。 12日目に、それらを顕微鏡下で針を用いて手動で選択し、StemFit® AS3で200日間浮遊培養で維持しました。 15日目に、浮遊神経ロゼット由来の球体を同じ培地中の組織培養プレートに播種した。 18 日目に、付着した神経ロゼットを TrypLE Select で単一細胞に解離し、StemFit® AS200 の PO/ラミニンでコーティングされたプレートにプレーティングしました。 培地は一日おきに交換した。 細胞は 3 ~ 4 日ごとに継代されました16,20。 201B7-Neurospheres は岡田洋平博士のご厚意により提供されました。4。 1210B2-EB-NS/PC は金村米弘博士のご厚意により提供されました。9。 AF22 と AF23 は、Austin Smith 博士のご厚意により提供されました。20。 hiPSC-NS/PC のニューロンおよびグリア分化のために、細胞を PO/ラミニンでコーティングされた 8 ウェル チャンバー ガラス スライド上に 2.5 × 10 の密度でプレーティングしました。4 セル/cm2 2% B27 および 1% GlutaMAX (Gibco; Thermo Fisher Scientific) を含む Neurobasal 培地 (Gibco; Thermo Fisher Scientific) 中で16。 骨形成分化および脂肪形成分化は、製造業者の指示に従って実施した(ヒト間葉系幹細胞(hMSC)分化ブレットキット、Lonza)。 骨形成分化の場合、合計 7.0 × 103 細胞を24ウェルプレートに移し、培地中で一晩培養した。 接着細胞は、3 日ごとに交換される骨形成分化培地 (Lonza) で培養されました。 15日後、これらの細胞の骨芽細胞への分化をアリザリンレッドS染色(Millipore)によって評価した。47。 脂肪生成分化の場合、7.0 × 104 細胞を24ウェルプレートに移し、細胞がコンフルエントに達するまで培養した。 100%のコンフルエンスで、誘導/維持の 3 サイクルを実行しました。 各サイクルは、細胞に脂肪生成誘導培地 (Lonza) を供給し、2 日間培養し、その後、脂肪生成維持培地 (Lonza) で 3 日間培養することから構成されます。 7 つの完全なサイクルの後、細胞を脂肪生成維持培地とともに 22 日間さらにインキュベートし、培地を週に XNUMX 回交換しました。 22日後、オイルレッドO染色(武藤純薬)を行い、細胞が脂肪細胞に分化したかどうかを調べた。
hiPSC (1210B2) からの hiPSC-NCC 誘導では、フィーダーフリーの hiPSC を 2 mg/ml コラゲナーゼ IV (Thermo Fisher Scientific) とともにインキュベートしました。 剥がれた iPSC コロニーを穏やかなピペッティングで細かく砕き、約 200 個の細胞からなるクラスターを 100 mm ペトリ皿 (Beckton Dickinson) 上の NCC 誘導用培地に播種しました。30。 NCC 誘導用の培地は、1:1 神経基礎培地 (Thermo Fisher Scientific) と、12x GlutaMax (Thermo Fisher Scientific)、1 mg/ml インスリン (Sigma-Aldrich)、5% ペニシリンおよびストレプトマイシンを含む DMEM/F-0.5 培地で構成されました。 0.5x GEM 21 NeuroPlex 無血清サプリメント (Gemini Bio Products, West Sacramento, CA)、0.5x N2 サプリメント、20 μg/ml ヒト組換え EGF (Peprotech) および 20 μg/ml FGF2 を補充。 培地は一日おきに交換した。 播種から 7 日後、付着した細胞塊から遊走性 NCC が観察されました。
MSCおよびWBMの調製用48、ヒト骨髄単核細胞 (BM-MNC) (Lonza、2M-125C) を、モノクローナル抗体 [LNGFR-PE (Miltenyi Biotec; 30:1) および THY-40-APC (BD Pharmingen)] を用いて氷上で 1 分間染色しました。 ; 1:200)]。 死細胞を除外するためにヨウ化プロピジウム (PI) (Sigma) を使用しました。 コントロールのフローサイトメトリー分析により、PI(-)LNGFR(+)THY-1(+) BM-MNC (MSC) のゲーティングおよびソーティングの設定が決定されました。 PI 陰性細胞を全骨髄細胞 (WBM) として分類しました。 フローサイトメトリー分析および選別手順は、FACSAriaセルソーター(BD Biosciences)を使用して実行されました。 選別された細胞をMSC培地[20%FBSおよびFGF2(5ng/ml)を含むDMEM(ナカライテスク)]で洗浄し、培養プレートに播種した。 コンフルエントに達する前に、20% トリプシン-EDTA (Gibco) を使用して細胞を継代しました。 実験には3継代した細胞を使用した。
単一細胞のクローニング
hiPSC-NS/PC (NS/PC-B) は、TrypLE Select (Thermo Fisher Scientific) を使用して解離されました。 解離した細胞をStemFit AS200(味の素社)に懸濁した。 単一細胞の選別は、SH800 フローサイトメーター (Sony) を使用して実行されました。 選別された細胞は、Matrigel (Corning) でコーティングされた 200 ウェル プレート (Greiner Bio-One) 内の StemFit AS96 で培養されました。 単一の選別された細胞に由来する各クローンをコンフルエントで継代した。 クローンは分析のためにさらに拡張されました。
免疫細胞化学
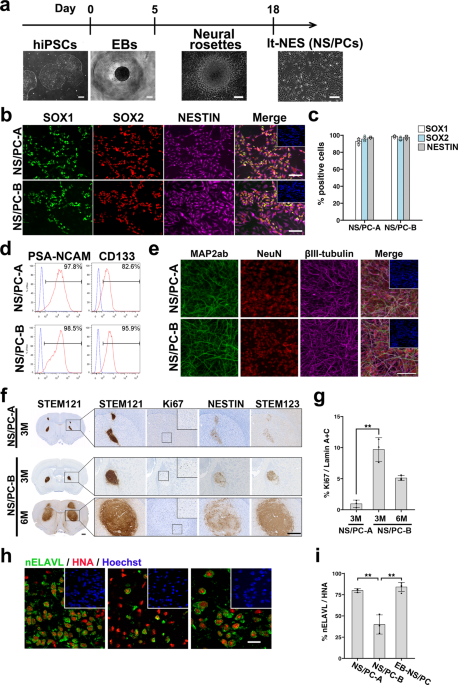
細胞を 4% PFA/PBS 中で室温で 15 ~ 20 分間固定し、PBS で 5 回洗浄しました。 次に細胞を 0.3% ウシ胎児血清および 100% Triton X-1 を含む PBS で室温で 4 時間透過処理およびブロックし、一次抗体: 抗 SOX1 (R&D Systems、AF3369; 1) とともに 500 °C で一晩インキュベートしました。 :2)、抗 SOX2018 (R&D Systems、MAB1; 500:18741)、抗ヒト Nestin (Immuno-Biological Laboratories Co.、1; 500:5326 および Millipore、MAB1; 200:9)、抗 SOX20095 (Santa) Cruz Biotechnology、sc-1; 200:8660)、抗βIII-チューブリン (Sigma-Aldrich、T1; 500:2)、抗 AP12726α (Santa Cruz Biotechnology、sc-1; 50:177487)、抗 NeuN ( abcam、ab1; 500:2)、抗 MAP1406ab (Sigma-Aldrich、M1; 500:342)、抗 GalC (Millipore、MAB1; 500:13)、抗 GFAP (Thermo Scientific、0300-1; 500: 67)および抗Ki15580(abcam、ab1; 1000:3)。 PBS で 1 回洗浄した後、細胞を Alexa Fluor 488、555、および 647 結合二次抗体 (Thermo Fisher Scientific; 1:1000) とともに室温で 1 時間インキュベートしました。 細胞核を33258μg/ml Hoechst 2 (Sigma-Aldrich)で対比染色した。 画像は、Apotome 蛍光顕微鏡 (Carl Zeiss)、Axio Imager Z6000 (Carl Zeiss)、または IN Cell Analyzer 2 (Cytiva) で取得しました。 SOXXNUMXの定量分析に+ そしてネスチン+ 図のセル。 1c9 フィールドがランダムに選択され、フィールドあたり 150 個以上の細胞が分析されました。 陽性細胞の数は、IN Cell Analyzer Workstation (Cytiva) のマルチターゲット分析モジュールによって定量化され、総核数に対して正規化されました。
免疫不全マウスへのhiPSC-NS/PCの移植
線条体内移植は、9 週齢の雌 NOG マウス(日本クレア)の線条体に hiPSC-NS/PCs を両側から注射することにより行われました(1.0 × 106 サイトごとのセル)。 生後9週目のNOD/SCIDマウスの中等度挫傷(IHインパクター、60~70kdyn)の9日後に、損傷した脊髄中心部にhiPSC-NS/PCを注入することにより脊髄内移植を行った(チャールズリバーラボラトリーズジャパン) (5.0×105 サイトごとのセル)9。 解剖した脳を100%エタノールで脱水し、キシレンで透明にし、パラフィンに包埋した。 次に、厚さ 5 μm の脳の連続冠状切片と脊髄の矢状切片を調製し、ヘマトキシリン エオシン (H&E) 染色および免疫組織化学のために処理しました。
免疫組織化学
3,3-ジアミノベンジジン染色は、Bond-Max自動染色システム(Leica)を使用して、製造業者の指示に従って実施した。 パラフィン包埋脳切片を脱パラフィンして再水和し、続いて BOND Epitope Retrieval Solution10 (Leica) 中で 100 °C で 1 分間加熱することにより抗原を賦活化しました。 切片を一次抗体とともに室温で 30 分間インキュベートしました。 抗ヒト細胞質 (STEM121、タカラバイオ、Y40410; 1:1000)、抗 Ki67 (Dako、M7240; 1:50 および Leica Biosystems、KI67; 1:100)、抗ラミン A + C (abcam、ab108595; 1:400)、抗ヒト NESTIN (IBL、18741; 1:100)、抗ヒト GFAP (STEM123、タカラバイオ、Y40420; 1:1000)、抗 NeuN (abcam、ab177487; 1:250)、抗-切断型カスパーゼ 3 (Cell Signaling Technology、9661; 1:250)、抗ヒト シナプトフィジン (R&D Systems、AF5555; 1:250)、抗 RUNX2 (abcam、ab192256; 1:500)。
蛍光免疫組織化学では、パラフィン包埋脳切片を脱パラフィンして再水和し、続いて Target Retrieval Solution (Dako、S10) 中で 121 °C で 1699 分間加熱することにより抗原を回復させました。 次に、切片を Blocking One (Nacalai Tesque) で室温で 1 時間ブロックし、一次抗体: 抗 nELAVL (Thermo Fisher Scientific、A4; 21271:1)、抗ヒト核抗原 (HNA) とともに 50 °C で一晩インキュベートしました。 ; Millipore、MAB4383; 1:100)、抗ヒト細胞質 (STEM121、Takara Bio、Y40410; 1:100)、抗 SOX1 (R&D Systems、AF3369; 1:200)、抗 SOX9 (Santa Cruz Biotechnology、sc) -20095; 1:100)、抗 AP2α (Santa Cruz Biotechnology、sc-12726; 1:50)、抗ヒト Vimentin NL493 結合ラット IgG2a、および抗ヒト Snail NL557 結合ヤギ IgG (Human EMT 3-カラー免疫細胞化学キット、R&D Systems、SC026)。 Alexa Fluor 488、555、および 647 結合二次抗体を 1:1000 で使用しました。 細胞核を 1 μg/ml Hoechst 33258 で対比染色しました。画像は、Apotome 蛍光顕微鏡、Axio Imager Z2、共焦点レーザー走査顕微鏡 (LSM700; Carl Zeiss)、または共焦点画像サイトメーター (CQ1; 横河電機株式会社) で取得しました。 蛍光画像は、20 倍の一次対物レンズを使用して取得されました。 HNAなどの陽性細胞の数+、SOX1+、SOX9+、AP2α+、およびnELAVL+ 細胞を各セクションでカウントした。
骨の染色
フォン・コッサ染色は、カルシウム染色キット(ScyTek Laboratories, Inc.、CVK-1)に基づいて実施した。 組織切片を5%硝酸銀溶液中で紫外線下で60分間インキュベートした。 インキュベーション後、切片を蒸留水で3回洗浄した。 次に、スライドを5%チオ硫酸ナトリウム溶液に2分間浸し、流水で2分間洗浄し、蒸留水で2回洗浄する。 切片をさらに核色素溶液で 5 分間染色しました。 最後に、切片をエタノールで脱水し、観察のためにマウントしました。 アリザリンレッドS染色の場合、アリザリンレッドS(ナカライテスク)を蒸留水で最終濃度1%に希釈して染色液を調製した。 溶液のpHをアンモニア水で60~5に調整した。 組織切片をアリザリンレッドS染色溶液中で室温で2分間インキュベートした。
Lyoplateを用いた細胞表面マーカーのスクリーニング
細胞は、BD Lyoplate™ ヒト細胞表面マーカー スクリーニング パネル BD Biosciences を使用して分析されました。 560747)、製造元のプロトコルに若干の変更を加えたものに従います。 単一細胞懸濁液を 0.5 ~ 1.0 × 10 で調製しました6 細胞/100μl、0.5μg/20μLの一次抗体とともに30℃で4分間インキュベートしました。 BD Pharmingen Stain Buffer (BD Biosciences、2)、554656 mM EDTA、および 5 μM Y-10 (Wako) を含む FACS Buffer で 27632 回洗浄した後、細胞を Alexa Fluor 647 結合二次抗体とともに 4 °C で 30 分間インキュベートしました。暗闇の中で341081分。 FACSバッファーで洗浄した後、BD CellFIX(BD Biosciences、7.6)で細胞を固定した。 フローサイトメトリー分析は、LSRFortessaフローサイトメーター(BD Biosciences)で実施した。 データは、FlowJo バージョン XNUMX (TreeStar) を使用して分析されました。
フローサイトメトリー
細胞を 0.5% ウシ血清アルブミンおよび 2 mM EDTA (pH 8.0) を含む PBS に 3.0 × 10 で懸濁しました。5 細胞/50 μl および蛍光色素結合抗体を用いて暗所で氷上で 30 分間染色: PSA-NCAM (Millipore、MAB5324; 1:50)、PSA-NCAM-APC (Miltenyi Biotec、130-093-273; 1) :50)、CD133-APC (Miltenyi Biotec、130-090-826; 1:50)、CD15-Brilliant Violet 421 (BioLegend、323039; 1:50)、CD49α-FITC (BioLegend、328308; 1:50)、 CD73-PE-Cy7、(BioLegend、127224; 1:50)、および CD105-APC (BioLegend、323208; 1:50)。 さらに、7-AAD (BD Biosciences、559925; 1:1000) を生死識別に適用しました。 アイソタイプコントロールを使用してバックグラウンド蛍光を差し引いた。 フローサイトメトリー分析は、FACSVerseフローサイトメーター(BD Biosciences)で実施した。 細胞選別は、FACSAriaセルソーター(BD Biosciences)で実施した。 データは FlowJo バージョン 7.6 を使用して分析されました。 ゲート戦略は補足図に示されています。 13.
インビボ評価のための NS/PC-B からの CD15 + CD73- CD105- NS/PC の精製
hiPSC-NS/PC (NS/PC-B) を 0.5% ウシ血清アルブミンおよび 2 mM EDTA (pH 8.0) を含む PBS に 3.0 × 10 で懸濁しました。5 細胞/50 μl、および蛍光色素結合抗体を用いて暗所の氷上で 30 分間:CD15-Brilliant Violet 421 (BioLegend、323039)、CD73-PE-Cy7、(BioLegend、127224)、および CD105-APC (BioLegend、 323208)。 7-AAD (BD Biosciences、559925) も生死の識別に使用されました。 アイソタイプコントロールを使用してバックグラウンド蛍光を差し引いた。 細胞選別はFACSAriaセルソーター(BD Biosciences)で行い、選別した細胞をMatrigel(Corning)でコーティングした200ウェルプレート(Greiner Bio-One)のAS9に播種して培養した。 XNUMX週齢のNOGマウス(日本クレア、インビボサイエンス株式会社)への線条体内移植は、前述のように行われました。9。 移植後 0.1 ~ 4 週間で、すべてのマウスを麻酔し、7% PFA を含む 10 M PBS の経心臓灌流によって安楽死させました。 解剖した脳をさらに 4%PFA で 24 時間固定し、免疫組織化学分析のために処理しました。
マイクロアレイ解析
全RNAをRNAeasy Kit (Qiagen)を用いて抽出した。 RNA の品質は、Agilent 2100 Bioanalyzer (Agilent Technologies) を使用して評価しました。 総RNA (200 ng) を逆転写し、Illumina Expression BeadChip 用 Target Amp-Nano Labeling Kit (Epicentre、Illumina) を使用してビオチンで標識し、メーカーの指示に従って HumanHT-12_v4_BeadChip (Illumina) にハイブリダイズさせました。 アレイを洗浄し、イルミナ遺伝子発現キットを使用して染色した。 生の強度値は、iScan マイクロアレイ スキャナー (Illumina) を使用して取得しました。 生のプローブ強度ファイルは、Illumina GenomeStudio 遺伝子発現ソフトウェア (v1.9.0) を使用してエクスポートされ、バックグラウンド補正、分位点正規化、および limma パッケージによる対数 (基数 2) 変換のために R にロードされました。 最後に、遺伝子セットを発現レベルによってフィルタリングして、すべてのサンプルで発現されなかった遺伝子を除去しました。
NS/PC 様 scNS/PC と NCC 様 scNS/PC の間で 1.2 倍異なる発現を示す遺伝子が抽出され、DAVID Bioinformatics Resources (http://david.ncifcrf.gov)。 遺伝子発現の箱ひげ図評価は BoxPlotR を使用して実行されました (http://shiny.chemgrid.org)49.
RNA シーケンス
RNA-seq 用のサンプルは、製造元のプロトコルに従って TruSeq RNA サンプル調製キット (Illumina) を使用して調製しました。 配列決定ライブラリーは、HiSeq 2500 (Illumina) で配列決定されました。 ベース コールと貞操フィルタリングは、リアルタイム分析ソフトウェア バージョン 1.18.61 を使用して実行されました。 デフォルト設定のセイルフィッシュ (v19) を使用して、生のリードを参照ゲノム hg0.7.6 にマッピングしました。 バショウカジキからのカウント行列データを R ソフトウェア (v4.1.1) にロードし、下流分析を実行しました。
単一細胞 RNA 配列
hiPSC-NS/PCをSH96フローサイトメーターによって800ウェルプレートに選別し、細胞溶解緩衝液(0.5% NP40)で溶解した。 これらの溶液をベンチトップミキサーを使用して 2,500 rpm、4 °C で 15 秒間混合し、その後 3000 × で混合しました。 g 4℃で10秒間。 0.8回目の遠心分離の直後、1.5μlのプライミングバッファー(MgCl2を含む41.67×PCRバッファー、TaKaRa Bio)、4pmol/lのRTプライマー(TATAGAATTCGCGGCCGCTCGCGATAATACGACTCACTATAGGGCGTTTTTTTTTTTTTTTTTTTTTT)、50U/μlのRNase阻害剤(Promega Corp)、および2500μmol /l dNTPを各ウェルに加えた。 溶液を 4 rpm、15 °C で 1000 秒間混合しました。 変性とプライミングは、サーマルサイクラー (C1000 および S70; BioRad Laboratories) を使用して、90 °C で 35 秒間、および 15 °C で 96 秒間実行されました。 次に、0 ウェル プレートを 0.8 °C のアルミニウム PCR ラックに置きました。 その後、1μlのRT緩衝液(25×PCR緩衝液、12.5U/μl逆転写酵素(SuperScript III; Life Technologies)、および35mmol/l DTT)を各ウェルに添加し、5℃で逆転写を行った。 45分間、20℃で70分間。 反応を 10 °C で 96 分間加熱不活化し、0 ウェル プレートを再度 3000 °C のアルミニウム PCR ラックに置きました。 XNUMX×遠心後 g 4℃で10秒間、1μlのエキソヌクレアーゼ溶液(1×エキソヌクレアーゼ緩衝液および1.5U/μlエキソヌクレアーゼI;両方ともTaKaRa Bio)を各ウェルに添加した。 プライマー消化は 37 °C で 30 分間実行され、反応は 80 °C で 10 分間熱不活化されました。 反応プレートをアルミニウム PCR ラックに 0 °C で置きました。 3000×遠心後 g および 4 °C で 30 秒間、2.5 μl のポリ A テーリング バッファー (1× PCR バッファー、3 mmol/l dATP、33.6 U/μl ターミナルトランスフェラーゼ (Roche Applied Science)、および 0.048 U/μl RNase H (Invitrogen) ) をアルミ PCR ラック内の各チューブに 0 °C で加え、反応プレートを 2500 rpm、4 °C で 15 秒間混合し、すぐに 3000 × 遠心分離しました。 g 0 °C で 10 秒間反応させた後、反応プレートをサーマル サイクラー ブロックに置き、0 °C に事前冷却しました。 続いて、ポリ A テーリング反応を 37 °C で 50 秒間実行し、65 °C で 10 分間熱不活化しました。 次に、反応プレートを 0 °C のアルミニウム PCR ラックに置きました。 3000×遠心後 g 4℃で30秒間反応させた後、反応プレートを0℃のアルミニウム製PCRラックに置きました。 次に、23μlの第1.09鎖バッファー(2×MightyAmpバッファーv70(TaKaRa)、0.054pmol/lタギングプライマー(TATAGAATTCGCGGCCGCTCGCGATTTTTTTTTTTTTTTTTTTTTT)、および2500U/μl MightyAmp DNAポリメラーゼ(TaKaRa))を各ウェルに加えました。 反応プレートを 4 rpm、15 °C で 3000 秒間混合しました。 XNUMX×遠心後 g 4℃で10秒間、第98鎖合成は130℃で40秒間、1℃で68分間、5℃で0分間実施した。 続いて、反応プレートをすぐに同じく25℃に冷却したアルミニウムPCRラックに移し、1μlのPCRバッファー(2×MightyAmpバッファーバージョン1.9および2μmol/lサプレッションPCRプライマー(NH3000)-GTATAGAATTCGCGGCCGCTCGCGAT)を加えた。が追加されました。 XNUMX×遠心後 g および 4 °C で 10 秒間、PCR 濃縮は、サイクルあたり次の条件を使用して合計 21 PCR サイクルで実行されました: 98 °C 10 秒、65 °C 15 秒、および 68 °C 5 分間。 PCR ステップの後、反応プレートを 68 °C で 5 分間インキュベートしました。 次に、反応プレートを 25 °C のアルミニウム PCR ラックに置きました。 増幅されたcDNAをPCR精製ビーズシステム(Agencourt AMPure XP; Beckman Coulter Inc)を使用して精製した。 増幅されたcDNAは、Nextera XT Library Prep Kit (Illumina)を使用してライブラリー調製のために処理されました。 DNA シーケンシング ライブラリは、超並列シーケンサー HiSeq 2500 で分析されました。生のリードは、Trimmomatic ソフトウェア (v0.33) を使用してリード品質とリード長によってトリミングされました。 トリムされたリードは、デフォルト設定のセイルフィッシュ (v19) を使用して参照ゲノム hg0.7.6 にアライメントされました。 サンプルは次のパラメータでフィルタリングされ、分析に使用されました: リード数 > 1 万、アライメント率 > 70%、検出された遺伝子数 > 5000。
差次的に発現される遺伝子の相関解析
個々の細胞内で差次的に発現される遺伝子と、代表的な組織または細胞の公的に利用可能なデータセットとの相関関係は、ExAtlas (https://lgsun.irp.nia.nih.gov/exatlas/)27。 体細胞組織のデータセットは ExAtlas にプリロードされています。 比較に使用した他のデータセットは次のとおりです: iPSC 由来の NS/PC (GSM1553289、GSM1553290、GSM1553291、および GSM2030405、GSM2040306)、iPSC 由来の NCC (GSM1470883、GSM1470884、および GSM1470885)、iPSC-派生 NCMSC (GSM1470886、GSM1470887、およびGSM1470888)、およびPSA-NCAM+またはPSA-NCAM-胚性幹細胞由来NS/PC(GSE67383)。
RNA-seqデータの再解析
新皮質および脊髄由来のヒト神経上皮幹細胞の RNA-seq データは、Gene Expression Omnibus (GEO) データベース (GSE107514) から取得しました。 SRA 生データはダウンロードされ、SRA Toolkit を使用して fastq データに変換されました。 デフォルト設定の kallisto (v38) を使用して、生のリードを参照ゲノム hg0.46.2 にマッピングしました。 kallisto からのカウント行列データを R ソフトウェア (v4.1.1) にロードし、ダウンストリーム分析を実行しました。
統計と再現性
すべてのデータは平均値 ± SD として表示されます。 統計的有意性はスチューデントの t-テスト。 サンプルサイズを決定するための統計的テストはありませんでした。 サンプルサイズは、同様の実験を行った以前のレポートに基づいて決定されました。 各実験のサンプルサイズは図の凡例の部分に記載されています。 統計的有意性の程度はアスタリスク、* で表されます。P <0.05; ****P <0.01; ***P < 0.001。 単一細胞クローニングを除くインビトロ実験は少なくとも XNUMX 回実施され、同様の結果が得られました。
レポートの概要
研究デザインの詳細については、 ネイチャー ポートフォリオ レポートの概要 この記事にリンクされています。
- SEO を活用したコンテンツと PR 配信。 今日増幅されます。
- EVMファイナンス。 分散型金融のための統一インターフェイス。 こちらからアクセスしてください。
- クォンタムメディアグループ。 IR/PR増幅。 こちらからアクセスしてください。
- プラトアイストリーム。 Web3 データ インテリジェンス。 知識増幅。 こちらからアクセスしてください。
- 情報源: https://www.nature.com/articles/s42003-023-04995-9



