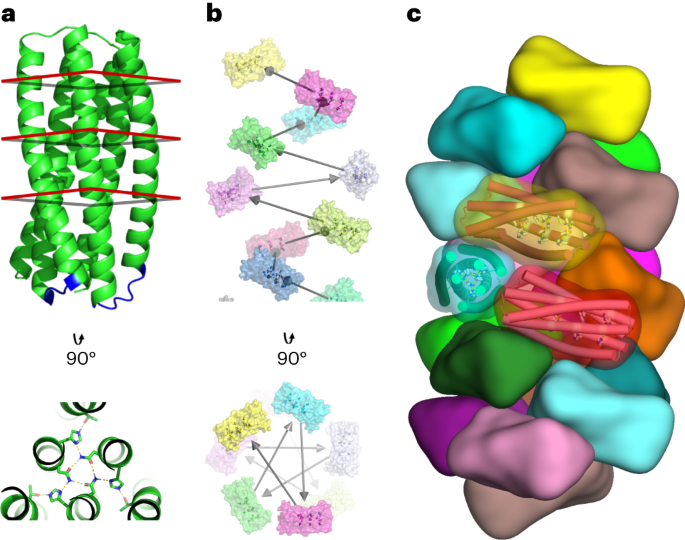
Chiến lược thiết kế tính toán
Các vòng ngắn để kết nối các vòng xoắn pRO-2.3 thành một chuỗi duy nhất được thiết kế bằng cơ sở dữ liệu đầy đủ về các mẫu xương sống bao gồm các mảnh trải dài hai vùng xoắn ốc được DSSP xác định trong các cấu trúc tinh thể có độ phân giải cao (như đã mô tả trước đây).14). Các vòng lặp được xác định trong cơ sở dữ liệu này thông qua việc căn chỉnh cứng nhắc các phần dư cuối cùng của đoạn và mục tiêu bằng thuật toán xếp chồng được tối ưu hóa15. Các ứng cử viên đáp ứng dung sai căn chỉnh 0.35 Å RMSD được căn chỉnh với xương sống mục tiêu thông qua tọa độ không gian xoắn và các ràng buộc tọa độ mềm đối với tọa độ nguyên tử nặng của xương sống ứng viên được căn chỉnh. Sau đó, các trình tự vòng lặp ứng viên được thiết kế theo các ràng buộc về cấu hình trình tự được tạo ra thông qua việc căn chỉnh đường trục vòng lặp với cơ sở dữ liệu cấu trúc nguồn. Các ứng viên có điểm thấp nhất được chọn vào thiết kế vòng cuối cùng.
Phương pháp thiết kế và lắp ghép xoắn ốc7 đã được áp dụng cho pRO-2.3 được liên kết để tạo ra các mô hình thiết kế dây tóc xoắn ốc. Các tiêu chí sau đây đã lọc các quỹ đạo thiết kế riêng lẻ: sự khác biệt vượt quá −15.0 đơn vị năng lượng Rosetta giữa trạng thái liên kết (polyme) và không liên kết (monomeric), diện tích bề mặt giao diện vượt quá 700 Å2, độ bổ sung hình dạng Rosetta vượt quá 0.62 và số lượng dư lượng cực không thỏa mãn được tính dưới 5. Các thiết kế đáp ứng các tiêu chí này đã trải qua quá trình sàng lọc thủ công, liên quan đến việc đảo ngược một điểm đối với các đột biến được coi là không góp phần ổn định trạng thái ràng buộc của giao diện. Sau đó, thiết kế đạt điểm cao nhất cho từng cấu hình được gắn đế sẽ được tích hợp vào bộ protein cuối cùng để xác thực bằng thực nghiệm.
Biểu hiện và tinh chế protein
Các gen tổng hợp cho tổng số 18 kiểu dáng đã được tối ưu hóa để biểu hiện trong Escherichia coli và thu được từ IDT, sau đó chèn vào vị trí nhân bản đa dạng của vectơ pET29b+ giữa các vị trí hạn chế NdeI và XhoI. Các cấu trúc này đã được đưa vào BL21* (DE3) E. coli tế bào có thẩm quyền. Các chất biến đổi được nuôi cấy trong 50 ml môi trường Terrific Broth bổ sung 200 mg l-1 kanamycin. Biểu hiện, dưới sự kiểm soát của chất kích thích T7, được tiến hành trong 24 giờ ở 37 ° C bằng cách sử dụng tính năng tự động cảm ứng của Studier16 cho đến khi mẫu cấy được thu hoạch bằng cách ly tâm. Các viên tế bào được treo lại trong dung dịch muối Tris-buffered (TBS) và được lọc bằng chất tẩy Bugbuster. Phần hòa tan, được làm rõ bằng cách ly tâm, được tinh chế qua Ni2+ sắc ký ái lực kim loại cố định sử dụng nhựa Ni-NTA Superflow. Nhựa có lysate tế bào liên kết được rửa bằng mười thể tích cột 40 mM imidazole và 500 mM NaCl, sau đó rửa giải bằng 400 mM imidazole và 75 mM NaCl. Các phần hòa tan và không hòa tan được phân tích bằng điện di trên gel polyacrylamide SDS. Các mẫu có dải protein ở trọng lượng phân tử chính xác được chọn để sàng lọc bằng kính hiển vi điện tử. Các thiết kế đã chọn được tăng tỷ lệ lên tới 0.5 l để mô tả thêm đặc tính, với quá trình biểu hiện lại tiếp tục trong 24 giờ ở 37 ° C bằng cách sử dụng tính năng tự động cảm ứng của Studier16 trước khi thu hoạch bằng phương pháp ly tâm. Các viên tế bào được treo lại trong TBS và được ly giải bằng phương pháp vi lỏng, sau đó được tinh chế như mô tả ở trên.
Vết âm tính EM
Các phần hòa tan được cô đặc trong TBS (dung dịch đệm Tris 25 mM, NaCl 75 mM, pH 8) để sàng lọc bằng kính hiển vi điện tử. Một giọt 6 µl (1 µl mẫu được pha loãng ngay lập tức với 5 µl dung dịch đệm) được nhỏ lên lưới đồng 200 lưới được phủ carbon, phóng điện phát sáng âm, rửa bằng nước Milli-Q và nhuộm màu bằng uranyl formate 0.75% (pH 4.0). ) hoặc Nano-W (pH 6.8) được mua từ Nanoprobes, Inc. như mô tả trước đây17. Việc sàng lọc được tiến hành bằng kính hiển vi điện tử truyền qua Morgagni M100 (FEI) 268 kV hoặc kính hiển vi điện tử truyền qua Talos L120C 120 kV (ThermoFisher). Hình ảnh được chụp bằng hệ thống camera Teitz CMOS 4k gắn phía dưới và được xử lý để tăng độ tương phản bằng phần mềm Fiji (phiên bản: 2.14.0/1.54f)18 cho rõ ràng.
Độ dài sợi được định lượng bằng thuật toán dò sợi trong cryoSPARC8. Phương pháp này xác định các sợi bằng cách tương quan chéo với một lớp mẫu và truy tìm các sợi liền kề từ các hạt đã xác định. Một lớp mẫu được tạo từ DpHF19 đã được sử dụng cho tất cả các sợi được đo. Sợi được lọc theo độ cong trung bình (<0.0005 Å-1) và tương quan chéo chuẩn hóa trung bình (> 0.5) trên mỗi sợi. Đối với DpHF18, chúng tôi sử dụng 5, 2, 3, 20, 28 và 21 hình ảnh tương ứng cho pH 3, 3.5, 4.2, 5, 8 và 3 đến 8. Đối với DpHF19, chúng tôi sử dụng hình ảnh 7, 8, 8, 28, 4 và 5 tương ứng cho độ pH 3, 3.5, 4.2, 5, 8 và 3 đến 8. Đối với DpHF19_9his, chúng tôi sử dụng hình ảnh 6, 6, 8, 14, 15, 8 và 4 tương ứng cho pH 3, 3.5, 4.2, 5, 6, 8 và 3 đến 8.
Cryo-EM
Các mẫu Cryo-EM được chuẩn bị bằng cách áp dụng protein vào lưới Holey-carbon CFLAT, loại bỏ chất lỏng và nhúng lưới vào ethane lỏng bằng Vitrobot (ThermoFisher). Đối với DpHF19, video được thu trên kính hiển vi Glacios (ThermoFisher) được trang bị camera K-2 Summit Direct Detect (Gatan Inc.) hoạt động ở chế độ đếm, với kích thước pixel là 1.16 Å mỗi pixel, 50 khung hình và tổng liều điện tử của 65 Å-2. Đối với DpHF18 và DpHF7, video được thu trên Titan Krios (ThermoFisher) được trang bị camera K-2 Summit Direct Detect (Gatan Inc.) hoạt động ở chế độ siêu phân giải, với kích thước pixel là 0.525 Å mỗi pixel, 50 khung hình và tổng liều điện tử là 90 Å-2. Việc thu thập dữ liệu tự động được thực hiện bằng Leginon19 phiên bản 3.4. Xử lý dữ liệu được thực hiện bằng cryoSPARC8và quy trình công việc được tóm tắt trong Hình bổ sung. 10–12. Các video được căn chỉnh bằng cách vá lỗi hiệu chỉnh chuyển động, với các video có độ phân giải siêu cao được đặt ở kích thước pixel là 1.05 Å. Các tham số của hàm truyền tương phản (CTF) được ước tính bằng cách sử dụng bản vá CTF. Việc dò tìm sợi không có mẫu được thực hiện trên một tập hợp con hình ảnh và các hạt thu được phải được phân loại 2D. Các lớp 2D đã chọn sau đó được sử dụng làm mẫu để theo dõi dây tóc dựa trên mẫu trên bộ dữ liệu đầy đủ. Sau nhiều vòng phân loại 2D, các hạt được chọn sẽ được sàng lọc 3D với tính năng đối xứng xoắn ốc được áp dụng và kích hoạt sàng lọc không đồng nhất. Đối với DpHF19, chúng tôi đã áp đặt tính đối xứng xoắn ốc một điểm liên quan đến các tiểu đơn vị riêng lẻ, không tiếp xúc, thay vì các tham số đối xứng xoắn ốc hai điểm bắt đầu. Đối với DpHF7 và DpHF19, độ lệch tiêu trên từng hạt, độ nghiêng chùm tia và quang sai hình cầu cũng được tinh chỉnh. Việc sửa đổi mật độ được thực hiện bằng ResolveCryoEM trong Phenix20,21 phiên bản phenix-1.20.1. Các mô hình nguyên tử cho DpHF18 và DpHF19 đã được tinh chỉnh thành bản đồ cryo-EM bằng ISOLDE22, tiếp theo là sàng lọc không gian thực trong Phenix, với các hạn chế rotamer và Ramachandran bị vô hiệu hóa và với các hạn chế tham chiếu do mô hình khởi động đầu vào áp đặt. Việc làm sáng tỏ mô hình cho DpHF7 đã sử dụng giao thức xây dựng mô hình de novo trên mật độ đơn vị bất đối xứng cryo-EM được phân đoạn23. Việc kết hợp và sàng lọc dư lượng sau đó đã đạt được bằng cách sử dụng RosettaCM24 phiên bản 2019.31, tận dụng tính đối xứng trên bản đồ cryo-EM không phân đoạn để có các giao diện phù hợp với mật độ và nội bộ dây tóc tối ưu. Vòng sàng lọc không gian thực cuối cùng đã được thực hiện trong Phenix, như được mô tả ở trên cho DpHF18 và DpHF19. Thống kê thu thập, sàng lọc và xác nhận dữ liệu Cryo-EM được tóm tắt trong Bảng bổ sung 1.
TIRFM
Lắp ráp sợi
Để tạo hình ảnh quá trình tạo mầm của các sợi phản ứng với pH, sợi DpHF18 được dán nhãn bằng hai loại fluorophor liên hợp maleimide khác nhau là Oregon488 và sulfo-Cy5. Các sợi được dán nhãn với lượng dư 10× mol, trong PBS + 1 mM TCEP trong 4 giờ ở nhiệt độ phòng, trước khi trao đổi đệm thành TBS (25 mM Tris, 100 mM NaCl, pH 8.0) trên cột quay Zeba và nồng độ đến 30 μM . Các sợi màu xanh lá cây ở mức 30 μM được tách rời bằng cách bổ sung 1 M citrate (0.6 μl citrate vào 20 μl chất xơ) để giảm độ pH xuống 3.0. Dung dịch này được ủ trong 5 phút trước khi bổ sung Tris (3.6 μl dung dịch 1 M) để đưa độ pH trở lại 8.0; 1 μl sợi DpHF18–Cy5 đã lắp ráp ở mức 30 μM đã được thêm vào dung dịch. Dung dịch này sau đó được ủ ở nhiệt độ phòng trước khi ly tâm ở tốc độ 13,000 g trong 2 phút trong máy ly tâm để bàn. Các sợi được treo lại trong TBS và được chụp ảnh bằng TIRFM.
Tháo gỡ sợi
Hình ảnh TIRFM nhanh của các sợi được tháo rời ở độ pH thấp được thực hiện trên hệ thống TIRF được chế tạo tùy chỉnh dựa trên chân đế Nikon Ti được trang bị hệ thống lấy nét hoàn hảo cùng với khả năng lấy nét nhanh. Z Giai đoạn áp điện (ASI), đèn chiếu sáng TIRF phương vị (iLas2, Roper France) với trường nhìn mở rộng tùy chỉnh (Cairn) và vật kính PLAN Apo 1.45 NA ×100. Hình ảnh được thu được bằng máy ảnh sCMOS chiếu sáng sau Photometrics Prime 95B chạy ở chế độ màn trập toàn cầu giả, được đồng bộ hóa với chiếu sáng phương vị. Hệ thống được vận hành bởi Metamorph 7.10.1.161. Các sợi có nhãn Sulfo-Cy5 maleimide được chụp ảnh bằng tia laser 630nm (OBIS Coherent 150 mW được gắn trong máy phóng laser Cairn) và được chụp ảnh bằng bộ lọc Chroma ET655lp gắn trong bánh xe Cairn Optospin với tốc độ khung hình là 1 khung hình cứ sau 16 ms.
Các sợi được chụp ảnh trong bộ đệm hình ảnh (25 mM Tris pH 8.0, 100 mM NaCl) trong tế bào dòng Ibidi được gắn trên các tấm phủ cấp phòng sạch (tùy chỉnh, 25 × 75 mm2, Nexterion) và được thụ động hóa bằng PLL-PEG (0.1 mg ml-1 trong 20 mM Hepes, pH 7.6; 5 phút). Các sợi được phép lắng đọng trên lớp phủ trong 5 phút trước khi các sợi không liên kết được loại bỏ bằng bộ đệm hình ảnh. Trong quá trình thu nhận nhanh, độ pH được giảm bằng cách cho dòng đệm có độ pH thấp (25 mM Tris, 100 mM NaCl, pH 3.0).
Để đo độ phân tách sợi trong dung dịch khối, các sợi được tạo hình trước trong ống Eppendorf 1.5 ml được trao đổi vào dung dịch đệm citrate ở độ pH thấp hơn để kích thích quá trình phân tách. Một phần của mỗi phản ứng pH được loại bỏ tại các thời điểm khác nhau và được thêm vào đĩa 96 giếng trong 10 phút để cho phép các sợi lắng xuống và bám dính vào chất nền thủy tinh. Đối với mỗi điều kiện và thời điểm, chín trường quan sát đã thu được trên kính hiển vi IN Cell Phân tích 2500HS (Thiết bị phân tử) sử dụng vật kính không khí Nikon ×60 PLAN Apo 0.95 NA và nguồn kích thích LED 631nm, thời gian phơi sáng 150 ms với lượng phát xạ được thu thập thông qua bộ lọc thông dải 684 ± 24 nm. Hình ảnh được định lượng bằng cách sử dụng tập lệnh CellProfiler tùy chỉnh để phân đoạn các sợi bằng thuật toán ngưỡng Otsu25. Giới hạn trên và dưới của ngưỡng, cũng như cửa sổ thích ứng cho ID đối tượng, được điều chỉnh cho đến khi các sợi được xác định chính xác so với tín hiệu nền. Độ dài trục chính của các đối tượng được xác định bằng quy trình CellProfiler được vẽ biểu đồ theo thời gian ủ cho từng điều kiện pH.
AFM pha lỏng
Chuẩn bị mẫu
Chúng tôi đã ủ 10 µl dung dịch poly-lysine 0.01% trọng lượng trên bề mặt mica muscovite mới cắt (12 mm, Ted Pella Inc.) trong 2 phút. Dung dịch dư được loại bỏ và bề mặt được rửa sạch bằng nước và làm khô bằng N2 xăng7. Sau đó, 30 µl dung dịch protein 10 µM trong đệm hình ảnh (25 mM Tris-HCl, 400 mM NaCl ở pH 8) được ủ trên mica phủ poly-lysine trong 30 phút và rửa bằng đệm hình ảnh để loại bỏ protein dư thừa. Độ pH của dung dịch đệm tách (25 mM Tris-HCl, 400 mM NaCl, pH 4.1, 4.4, 4, 5 hoặc 4.7) được điều chỉnh bằng NaOH 10 M hoặc axit xitric 1 M và được lọc bằng bộ lọc PVDF cỡ lỗ 0.1 µm trước khi sử dụng . Đối với các thí nghiệm quang axit, dung dịch protein 10 µM trong 25 mM Tris-HCl pH 8 được ủ trên mica trần trong 30 phút và rửa bằng 25 mM Tris-HCl pH 5.5; bước lắng đọng và rửa bổ sung được thực hiện nếu mật độ số lượng sợi trên bề mặt thấp. Chúng tôi cũng đã chuẩn bị mới 1 mM 2-nitrobenzaldehyde (Sigma-Aldrich) trong 25 mM Tris-HCl pH 5.5 và sử dụng ngay mà không cần tiếp xúc với ánh sáng ở bất kỳ giai đoạn nào26. Các phép đo quang phổ và độ pH chỉ ra rằng 2-nitrobenzaldehyde có thể kích hoạt giữa các bước sóng 200 và 405 nm và làm giảm độ pH từ 5.5 xuống 2.7, đồng thời cường độ laser cao hơn dẫn đến tiêu thụ và axit hóa nhanh hơn.
Hình ảnh
Đối với nghiên cứu động học ở thành phần không đổi, chất nền mica poly-lysine được phủ protein được đặt dưới tế bào chất lỏng AFM (Bruker Multimode8). Hình ảnh được chụp trong bộ đệm hình ảnh bằng cách sử dụng công cụ đúc hẫng silicon nitride sạch (Bruker, SNL-10, hằng số lò xo: 0.12 N m-1, UV ozon hóa trong 5 phút) ở chế độ khai thác ở nhiệt độ phòng (25°C). Trước khi chạy bộ đệm tháo rời, các sợi được chụp ảnh liên tục trong 10 phút để tối ưu hóa các tham số (256 dòng quét, tốc độ quét 1.5 Hz, mức tăng tích phân cao (3–4) và biên độ tự do 50–100 mV). Sau khi xác nhận rằng không có hư hỏng do công xôn gây ra, bộ đệm tháo rời được bơm liên tục ở mức 25 µl phút-1. Việc thiết lập dòng chảy qua được tối ưu hóa để mang lại thời gian lưu trú không đáng kể và chuyển đổi pH nhanh10.
Đối với nghiên cứu quang axit, mica phủ protein với 25 mM Tris-HCl pH 5.5 được đặt dưới tế bào chất lỏng của Cypher VRS AFM (Nghiên cứu tị nạn) được trang bị laser BlueDrive (bộ lọc cường độ × 0.3, bước sóng 405nm) với van thông hơi mở và vận hành ở chế độ khai thác. Sau khi xác nhận độ che phủ bề mặt cao của các sợi, bộ đệm hình ảnh được thay thế bằng 1 mM 2-nitrobenzaldehyde trong 25 mM Tris-HCl pH 5.5, hoạt động mà không tiếp xúc với ánh sáng nền khả kiến và chụp ảnh lại. Sau đó, công xôn được rút lại và BlueDrive được bật và quét liên tục trên các khu vực được chọn trước bằng cách sử dụng kính hiển vi quang học cơ giới của AFM. Tổng thời gian tiếp xúc với tia cực tím trong raster/dwell đối với các mẫu điểm và đường không quá 10 phút, sau đó công xôn được di chuyển trở lại khu vực tiếp xúc và chụp ảnh. Đối với sự thay đổi độ pH toàn cầu, cửa sổ thạch anh của tế bào chất lỏng AFM tiếp xúc với dung dịch axit quang được tiếp xúc với đèn UV cầm tay (bước sóng 364nm) trong 7 phút, sau đó chụp ảnh.
Hình ảnh được xử lý bằng phần mềm phân tích dữ liệu Gwyddion SPM v2.62 và phân tích bằng phần mềm Fiji v1.53s18. Đối với động học, tổng chiều dài sợi được đo và bất kỳ mảnh nào được coi là đã được tháo rời đều bị loại khỏi phép đo chiều dài. Để đo tốc độ tháo gỡ ở mỗi đầu của từng sợi riêng lẻ (Hình bổ sung. 8), tâm của sợi (một nửa chiều dài ban đầu) được chỉ định làm đầu thứ hai để đo chiều dài, trong khi đối với các đoạn sợi, tâm của đoạn được đo là đầu thứ hai.
- Phân phối nội dung và PR được hỗ trợ bởi SEO. Được khuếch đại ngay hôm nay.
- PlatoData.Network Vertical Generative Ai. Trao quyền cho chính mình. Truy cập Tại đây.
- PlatoAiStream. Thông minh Web3. Kiến thức khuếch đại. Truy cập Tại đây.
- Trung tâmESG. Than đá, công nghệ sạch, Năng lượng, Môi trường Hệ mặt trời, Quản lý chất thải. Truy cập Tại đây.
- PlatoSức khỏe. Tình báo thử nghiệm lâm sàng và công nghệ sinh học. Truy cập Tại đây.
- nguồn: https://www.nature.com/articles/s41565-024-01641-1



