Kiểm soát chất lượng, căn chỉnh và gọi SNP
Kiểu gen dựa trên trình tự ddRAD của 58 cá thể thuộc sáu giống gia súc bản địa; Gia súc Gir, Sahiwal, Tharparkar, Rathi, Red Sindhi và Kankrej với sự phân bố địa lý và sinh thái của chúng (Hình XNUMX). 1) bao gồm mục đích sản xuất, màu lông, vùng khí hậu đại diện, đường sinh sản, tọa độ địa lý của từng đường sinh sản cùng với ID động vật và giới tính của từng cá thể được trình bày trong Bảng bổ sung S1; dẫn đến 138.59 triệu lượt đọc thô tương ứng với 23 triệu lượt đọc cho mỗi giống và 2.2 triệu lượt đọc cho mỗi con vật. Sau khi lọc ban đầu trên cơ sở chất lượng đọc và loại bỏ bộ điều hợp, phần lớn số lần đọc (138.58 triệu lượt đọc; 99.9%) được giữ lại (Bảng bổ trợ S2). Tỷ lệ đọc cao (94.53%) được ánh xạ tới bos taurus (ARS-UCD1.2) tổ hợp tham chiếu (Bảng bổ trợ S2). Trong nghiên cứu này, nỗ lực được thực hiện để chỉ phân tích SNP trên các giống gia súc khác nhau, do đó, tất cả các biến thể khác không được xem xét trong phân tích tiếp theo. Số lượng SNPs trong 6 giống gia súc nằm trong khoảng từ 8,42,768 đến 3,81,966 sau khi gọi biến thể riêng lẻ. Số lượng SNP tối đa được quan sát thấy trong SAC (8,42,768), tiếp theo là GIC (8,34,780), KAC (8,10,279), RAC (8,05,020), RSC (6,72,632) và THC (3,81,966) (Bàn 1). Bộ dữ liệu kết hợp trên 6 giống gia súc đã tạo ra tổng cộng 43,47,445 SNP. Sau đó, tệp VCF được xử lý theo cách từng bước để lọc ra các SNP chất lượng thấp. Đầu tiên, các SNP được lọc ở độ sâu đọc 2 (RD 2), độ sâu đọc 5 (RD 5) và độ sâu đọc 10 (RD 10). Để phân tích sâu hơn, bộ dữ liệu gồm 9,82,174 SNP được xác định ở RD là 5, được sử dụng để phân tích tiếp theo (Bảng 1). Tất cả những SNP có mặt ở mức độ phù hợp thấp (RD <5) đã bị xóa khỏi tập dữ liệu. Các SNP được xác định ở RD trên 5 đã được lọc thêm bằng nhiều tiêu chí khác nhau, chẳng hạn như tỷ lệ kiểu gen bị thiếu, tần số alen nhỏ và Cân bằng Hardy Weinberg (HWE). Một loạt lọc dẫn đến tổng số 84,027 SNP chất lượng cao. Sau quá trình lọc, số lượng SNP giữa các giống khác nhau đáng kể. Số lượng SNP cao nhất được quan sát thấy trong GIC (34,743), tiếp theo là RSC (13,092), KAC (12,812), SAC (8956), THC (7356) và RAC (7068) (Bảng 2).
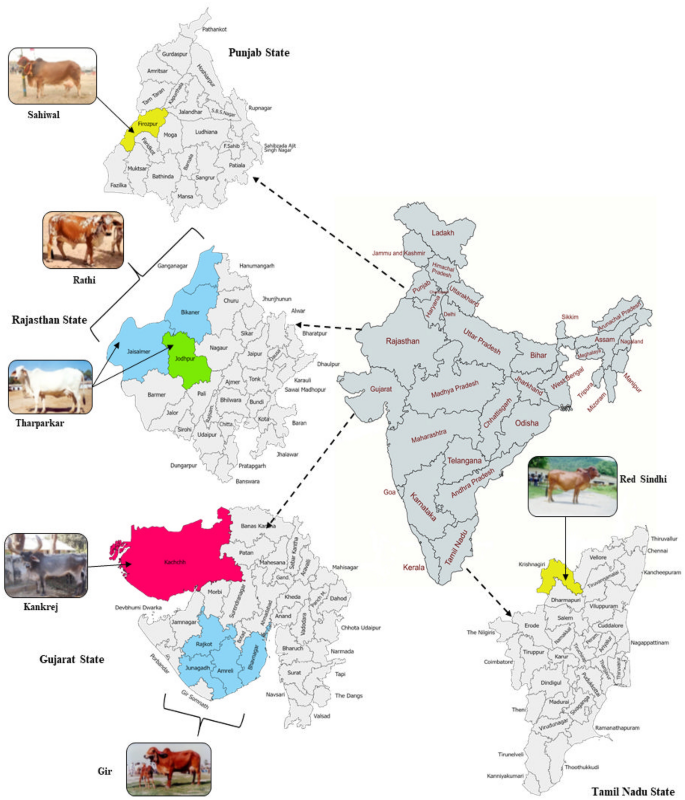
Phân bố địa lý của sáu giống gia súc được đưa vào nghiên cứu này (Bản đồ được tạo bằng các trang web Map Chart https://www.mapchart.net/ và Vẽ bản đồ https://paintmaps.com/).
Chú thích chức năng của các biến thể
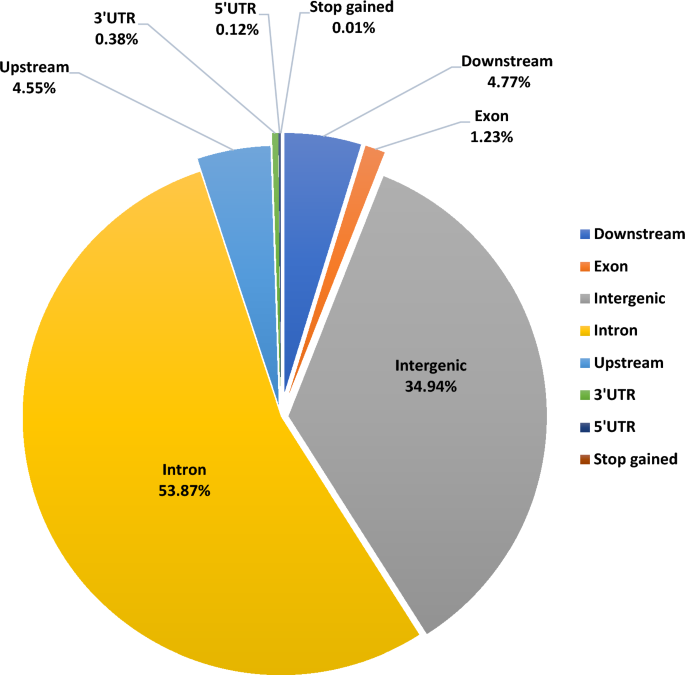
Bộ dữ liệu SNPs chất lượng cao được hợp nhất của tất cả 6 giống bò sữa đã được chú thích thành bos taurus (ARS-UCD1.2) bộ gen tham khảo. Đối với sự phân bố của chúng trong bộ gen, một số lượng lớn SNP được chú thích đã được dự đoán là ở khu vực phức tạp (41,372 SNP, 53.87%), tiếp theo là các khu vực xen kẽ (26,834 SNP, 34.94%). Chỉ có 948 SNP (1.23%) được phân phối ở các vùng exonic. Hơn nữa, có 3497 SNP (4.55%) nằm trong vùng 5 Kb ngược dòng và 3661 SNP (4.77%) ở hạ nguồn của vị trí bắt đầu phiên mã. Phân tích cũng cho kết quả 93 SNP (0.121%) nằm ở vùng 5'UTR, 293 SNP (0.38%) ở vùng 3'UTR. Tổng cộng có 8 SNP (0.01%) được dự đoán là gây ra codon dừng sớm cũng đã được xác định (Hình. 2).
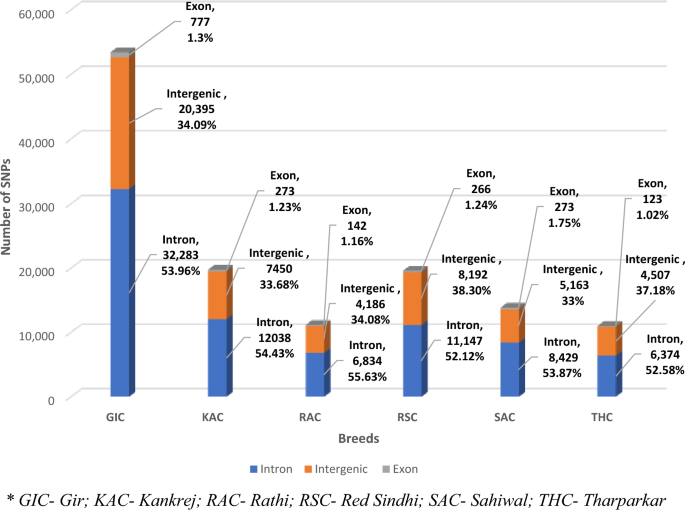
Trên cơ sở tác động của SNPs đối với gen mã hóa protein, SNPs được phân loại là có tác động cao (10 SNPs; 0.01%), tác động vừa phải (298 SNPs; 0.39%) và tác động thấp (697 SNPs; 0.91%). . Phần lớn các SNP (75,801; 98.69%) được xác định là công cụ sửa đổi (Bảng bổ trợ S3). Ngoài ra, tỷ lệ cao SNP (65.74%) có bản chất im lặng, tiếp theo là tên lửa (33.37%) và vô nghĩa (0.89%), với tỷ lệ tên lửa/im lặng trung bình là 0.507 (Bảng bổ trợ S4). Ngoài ra, trong số tất cả các kiểu gen thay thế được xác định trong nghiên cứu hiện tại, kiểu gen C/T và G/A được cho là chiếm ưu thế, trong khi kiểu gen A/T được tìm thấy ở tỷ lệ thấp nhất (Bảng bổ trợ S5). Đối với từng giống riêng lẻ, kết quả chú thích được tóm tắt trong Hình. 3 và bảng bổ trợ S6. Trong GIC, số lượng SNP cao nhất 32,283 (53.96%) được dự đoán là ở khu vực nghiêm trọng, tiếp theo là khu vực xen kẽ 20,395 (34.09%). Chỉ có 777 (1.3%) được phát hiện ở vùng exonic. Tương tự như GIC, số lượng SNP cao nhất được phân phối ở khu vực phức tạp, tiếp theo là khu vực xen kẽ và ngoại lai trong tất cả các giống gia súc khác. Ví dụ: trong SAC, 53.87% SNP (8429) được dự đoán ở khu vực khắc nghiệt, tiếp theo là khu vực xen kẽ 33% (5163 SNP) và chỉ 1.75% (273 SNP) ở khu vực exonic. Một xu hướng tương tự đã được quan sát thấy đối với các giống gia súc RAC, RSC, KAC và THC với 6834 (55.63%), 11,147 (52.12%), 8429 (53.87%), 6374 (52.58%) SNP tương ứng ở khu vực khắc nghiệt, 4186 (34.08) %), 8192 (38.30%), 5163 (33%), 4507 (37.18%) SNP tương ứng ở khu vực liên gen và chỉ 142 (1.16%), 266 (1.24%), 273 (1.75%), 123 (1.02) %) đã được dự đoán ở các khu vực exonic (Hình. 3). Số biến thể đồng nghĩa được xác định trong GIC, KAC, RAC, RSC, SAC và THC lần lượt là 570, 190, 101, 172, 213 và 87. Mặt khác, số biến thể không đồng nghĩa được phát hiện đối với 6 giống gia súc lần lượt là 165, 64, 31, 82, 53 và 30. chữ TS/TV tỷ lệ quan sát được trong GIC, KAC, RAC RSC SAC và THC lần lượt là 2.55, 2.64, 2.33, 2.43, 2.51 và 2.19 (Bảng bổ trợ S6).
Số lượng SNP liên gen là 4,639,873 (68.1%) và 1,676,710 (24.6%) là nghiêm trọng. Có 230,365 (3.4%) SNP nằm trong phạm vi 5 kb ngược dòng và 197,827 (2.9%) ở hạ lưu của vị trí bắt đầu phiên mã; 12,428 SNP được đặt trong 5′ UTR và 2613 trong 3′ UTR. Tổng cộng có 4356 SNP được đặt tại các vị trí nối của 2966 gen: 142 nằm trong các vị trí cho mối nối, 142 là các vị trí chấp nhận mối nối và 4072 nằm trong khu vực của vị trí nối. Chúng tôi đã xác định được 45,776 SNP ảnh hưởng đến trình tự mã hóa của 11,538 gen. Có 221 SNP được dự đoán sẽ gây ra codon dừng sớm và 17 gây ra mức tăng trong chuỗi mã hóa. Số lượng SNP được dự đoán là không đồng nghĩa là 20,828. Số lượng SNP liên gen là 4,639,873 (68.1%) và 1,676,710 (24.6%) là nghiêm trọng. Có 230,365 (3.4%) SNP nằm trong phạm vi 5 kb ngược dòng và 197,827 (2.9%) ở hạ lưu của vị trí bắt đầu phiên mã; 12,428 SNP được đặt trong 5′ UTR và 2613 trong 3′ UTR. Tổng cộng có 4356 SNP được đặt tại các vị trí nối của 2966 gen: 142 nằm trong các vị trí cho mối nối, 142 là các vị trí chấp nhận mối nối và 4072 nằm trong khu vực của vị trí nối. Số lượng SNP liên gen là 4,639,873 (68.1%) và 1,676,710 (24.6%) là nghiêm trọng. Có 230,365 (3.4%) SNP nằm trong phạm vi 5 kb ngược dòng và 197,827 (2.9%) ở hạ lưu của vị trí bắt đầu phiên mã; 12,428 SNP được đặt trong 5′ UTR và 2613 trong 3′ UTR. Tổng cộng có 4356 SNP được đặt tại các vị trí nối của 2966 gen: 142 nằm trong các vị trí cho mối nối, 142 là các vị trí chấp nhận mối nối và 4072 nằm trong khu vực của vị trí nối. Chúng tôi đã xác định được 45,776 SNP ảnh hưởng đến trình tự mã hóa của 11,538 gen. Có 221 SNP được dự đoán sẽ gây ra codon dừng sớm và 17 gây ra mức tăng trong chuỗi mã hóa. Số lượng SNP được dự đoán là không đồng nghĩa là 20,828. Số lượng SNP liên gen là 4,639,873 (68.1%) và 1,676,710 (24.6%) là nghiêm trọng. Có 230,365 (3.4%) SNP nằm trong phạm vi 5 kb ngược dòng và 197,827 (2.9%) ở hạ lưu của vị trí bắt đầu phiên mã; 12,428 SNP được đặt trong 5′ UTR và 2613 trong 3′ UTR. Tổng cộng có 4,356 SNP được đặt tại các vị trí nối của 2966 gen: 142 nằm trong các vị trí cho mối nối, 142 là các vị trí chấp nhận mối nối và 4072 nằm trong khu vực của vị trí nối. Chúng tôi đã xác định được 45,776 SNP ảnh hưởng đến trình tự mã hóa của 11,538 gen. Có 221 SNP được dự đoán sẽ gây ra codon dừng sớm và 17 gây ra mức tăng trong chuỗi mã hóa. Số lượng SNP được dự đoán là không đồng nghĩa là 20,828.
Trong đa dạng giống
Độ đa dạng nucleotide (π) cao nhất ở THC (π = 0.458), tiếp theo là RSC (π = 0.364), SAC (π = 0.363), GIC (π = 0.356), KAC (π = 0.348) và RAC (π = 0.347 ). Giá trị đa dạng nucleotide trung bình là 0.373 (Bảng 3). Giá trị D của Tajima là âm đối với 4 giống gia súc viz., RSC, RAC, SAC và THC ngoại trừ GIC và SAC có quan sát thấy giá trị D dương. Giá trị D của Tajima âm cao nhất được quan sát thấy ở THC (-1.194), tiếp theo là RSC (- 1.088), RAC (- 0.295) và KAC (- 0.279).
Tỷ lệ dị hợp tử quan sát được (HO) nằm trong khoảng từ 0.464 đến 0.551 trong khi tỷ lệ dị hợp tử dự kiến (HE) dao động từ 0.448 đến 0.535. Các giá trị dị hợp tử quan sát được cao nhất được quan sát thấy ở THC (HO = 0.551) theo sau là RAC (HO = 0.523), RSC (HO = 0.5184), SẮC (HO = 0.5180), GIC (HO = 0.499) và KAC (HO = 0.464) (Bảng 4). F trung bìnhIS (hệ số cận huyết) dao động từ -0.253 trong THC đến 0.0513 trong KAC. FIS ước tính trong số sáu giống gia súc có hàm lượng THC cao nhất (FIS = − 0.253) theo sau là RAC (FIS = − 0.105), trong khi F thấp nhấtIS ước tính đã được quan sát thấy trong KAC (FIS = 0.0513) theo sau là GIC (FIS = − 0.00063). tổng thể FIS phân tích cho thấy có quá nhiều dị hợp tử đối với tất cả các giống gia súc ngoại trừ KAC (Bảng 4). dị hợp tử và FIS ước tính cho thấy sự hiện diện của đủ đa dạng trong sáu giống gia súc.
Giữa đa dạng giống
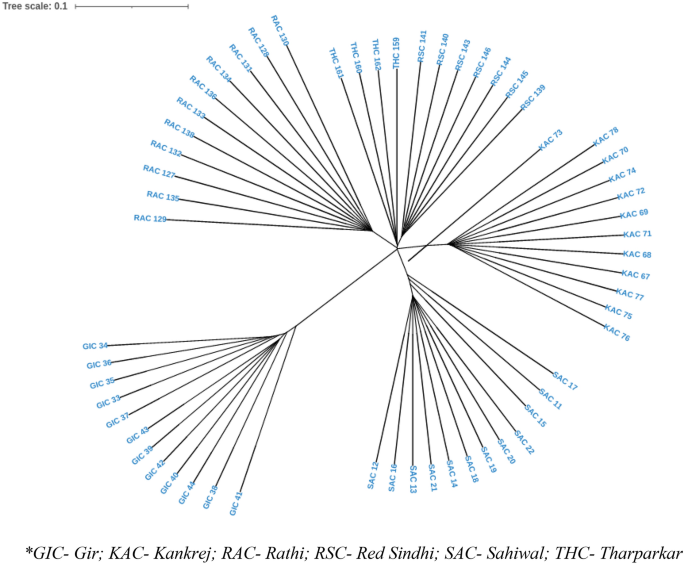
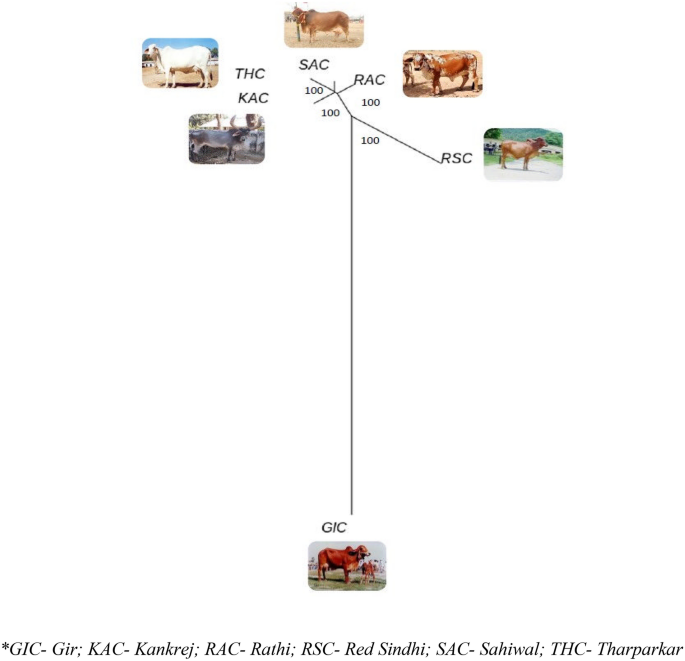
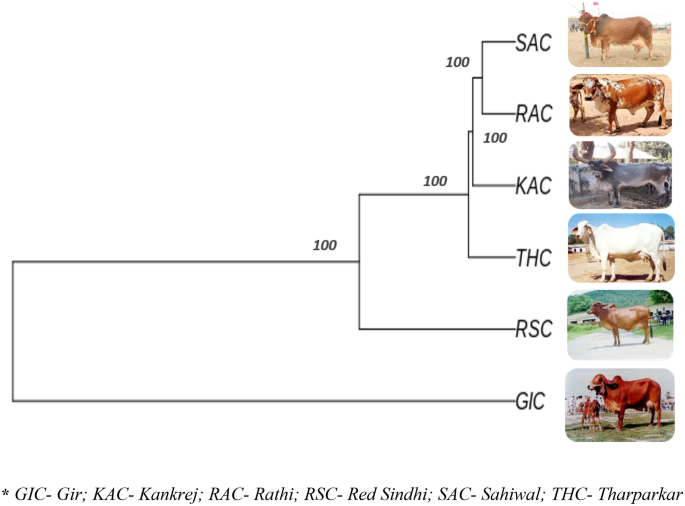
Sự phân hóa di truyền trên cơ sở chỉ số cố định (FST) dao động từ 0.2840 đến 0.3905, cho thấy đủ giữa đa dạng giống. Sự phân kỳ cao nhất được quan sát thấy giữa cặp RAC-SAC (FST = 0.3905), tiếp theo là cặp giống RSC-RAC (FST = 0.3790), cặp giống RSC-SAC (FST = 0.3751). Sự phân kỳ ít nhất đã được quan sát thấy đối với cặp giống KAC-THC (FST = 0.2840) (Bảng 5). Cây dựa trên Neighbor Joining (NJ) được xây dựng, nhóm các động vật riêng lẻ của 6 giống gia súc theo liên kết giống của chúng với GIC và RSC là giống đa dạng nhất trong số 6 giống gia súc được nghiên cứu. Mối quan hệ phát sinh gen ở cấp độ cá nhân được thể hiện trong Hình. 4. Cây NJ khôn ngoan của giống được mô tả trong hình. 5, ít nhiều được chứng thực bằng cây cấp độ riêng lẻ. Hơn nữa, cây phát sinh gen dựa trên UPGMA đã được xây dựng ở cấp độ giống bằng cách sử dụng gói “phangorn” trong nền tảng R với 100 giá trị bootstrap. Các giá trị bootstrap của mỗi nút gần bằng 100% cho thấy độ bền cao của cây được xây dựng. Cây phát sinh gen dựa trên UPGMA phản ánh mối quan hệ di truyền tương tự như được tiết lộ bởi sự khác biệt di truyền dựa trên NJ (khôn ngoan của từng cá nhân và ở cấp độ giống) trong đó GIC và RSC xuất hiện như những giống khác biệt nhất. GIC xuất hiện trên nút chính và được nhóm lại thành một nhóm trong khi các quần thể khác tạo thành hai nhóm với RSC được nhóm trên một nút và RAC, THC, SAC và KAC tạo thành các cụm phụ khác (Hình XNUMX). 6).
Phân tích cơ cấu dân số
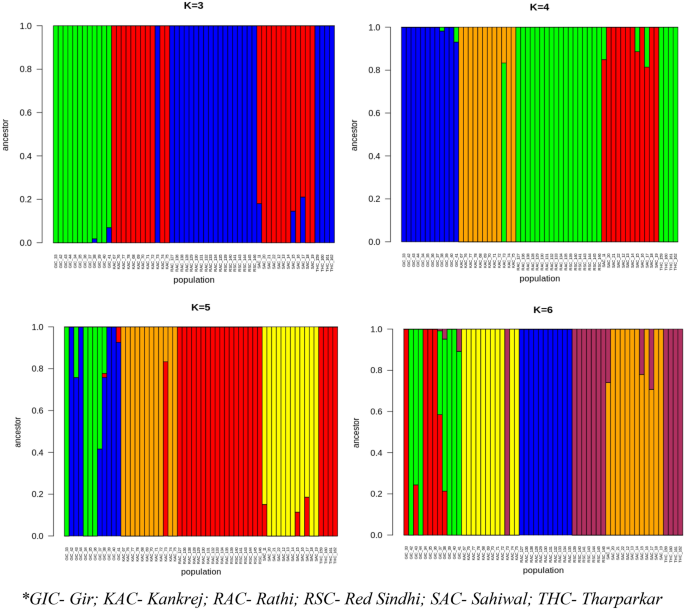
Phân tích hỗn hợp được thực hiện bằng cách phân vùng bộ gen của từng cá nhân thành một cụm được xác định trước. Việc phân tích được thực hiện ở K = 3, 4, 5 và 6 (Hình XNUMX). 7). Các cá thể không thể được nhóm lại ở K = 3 theo giống tương ứng của chúng. Chỉ GIC mới có thể được phân biệt rõ ràng trong khi các cá nhân của KAC và SAC xuất hiện dưới dạng một nhóm và RAC, THC và RSC được nhóm lại với nhau biểu thị tổ tiên chung của chúng. Ở K = 4 và thậm chí ở K = 5, THC, RAC và RSC tập hợp lại với nhau cho thấy tổ tiên chung mạnh mẽ của chúng, trong khi tất cả các cá thể khác tập hợp thành giống tương ứng của riêng chúng. K tốt nhất trong phân tích cấu trúc quần thể là K = 6, theo đó hầu hết tất cả các loài động vật được nhóm theo giống tương ứng của chúng, cho thấy rõ ràng tổ tiên của chúng, ngoại trừ RSC và THC vẫn nhóm lại với nhau. Sự gần gũi về mặt di truyền giữa RSC và THC có thể được tiết lộ bằng các nghiên cứu chuyên sâu hơn và bằng cách tăng số lượng mẫu.
Phân tích dựa trên PCA cũng phân nhóm 6 giống gia súc riêng biệt và củng cố thực tế rằng đây là những giống gia súc riêng biệt (Hình bổ sung. S1). Các cá thể của KAC được nhóm lại với nhau trong một góc phần tư, trong khi các cá thể của các giống gia súc SAC RAC, THC và RSC rơi vào một góc phần tư khác. Các cá thể của giống gia súc GIC xuất hiện như một quần thể riêng biệt.
- Phân phối nội dung và PR được hỗ trợ bởi SEO. Được khuếch đại ngay hôm nay.
- EVM tài chính. Giao diện hợp nhất cho tài chính phi tập trung. Truy cập Tại đây.
- Tập đoàn truyền thông lượng tử. Khuếch đại IR/PR. Truy cập Tại đây.
- PlatoAiStream. Thông minh dữ liệu Web3. Kiến thức khuếch đại. Truy cập Tại đây.
- nguồn: https://www.nature.com/articles/s41598-023-32418-6