I det här inlägget visar vi hur man effektivt finjusterar en toppmodern proteinspråksmodell (pLM) för att förutsäga proteinsubcellulär lokalisering med hjälp av Amazon SageMaker.
Proteiner är kroppens molekylära maskiner, ansvariga för allt från att röra dina muskler till att svara på infektioner. Trots denna variation är alla proteiner gjorda av upprepade kedjor av molekyler som kallas aminosyror. Det mänskliga genomet kodar för 20 standardaminosyror, var och en med en något olika kemisk struktur. Dessa kan representeras av bokstäver i alfabetet, som sedan låter oss analysera och utforska proteiner som en textsträng. Det enorma möjliga antalet proteinsekvenser och strukturer är det som ger proteiner deras många olika användningsområden.
Proteiner spelar också en nyckelroll i läkemedelsutveckling, som potentiella mål men också som terapeutiska medel. Som visas i följande tabell var många av de mest sålda läkemedlen under 2022 antingen proteiner (särskilt antikroppar) eller andra molekyler som mRNA som översatts till proteiner i kroppen. På grund av detta behöver många life science-forskare svara på frågor om proteiner snabbare, billigare och mer exakt.
| Namn | Tillverkare | Global försäljning 2022 (miljarder USD) | Indikationer |
| Komirnati | Pfizer / BioNTech | $40.8 | Covid-19 |
| Spikevax | modern | $21.8 | Covid-19 |
| Humira | Abbvie | $21.6 | Artrit, Crohns sjukdom och andra |
| Keytruda | Merck | $21.0 | Olika cancerformer |
Datakälla: Urquhart, L. Toppföretag och läkemedel efter försäljning 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Eftersom vi kan representera proteiner som sekvenser av tecken, kan vi analysera dem med hjälp av tekniker som ursprungligen utvecklats för skriftspråk. Detta inkluderar stora språkmodeller (LLM) som är förtränade på enorma datamängder, som sedan kan anpassas för specifika uppgifter, som textsammanfattning eller chatbots. På liknande sätt är pLMs förtränade på stora proteinsekvensdatabaser med hjälp av omärkt, självövervakad inlärning. Vi kan anpassa dem för att förutsäga saker som 3D-strukturen hos ett protein eller hur det kan interagera med andra molekyler. Forskare har till och med använt pLM för att designa nya proteiner från grunden. Dessa verktyg ersätter inte mänsklig vetenskaplig expertis, men de har potential att påskynda preklinisk utveckling och utformning av försök.
En utmaning med dessa modeller är deras storlek. Både LLM och pLM har vuxit i storleksordningar under de senaste åren, vilket illustreras i följande figur. Detta innebär att det kan ta lång tid att träna upp dem till tillräcklig noggrannhet. Det betyder också att du behöver använda hårdvara, särskilt GPU:er, med stora mängder minne för att lagra modellparametrarna.
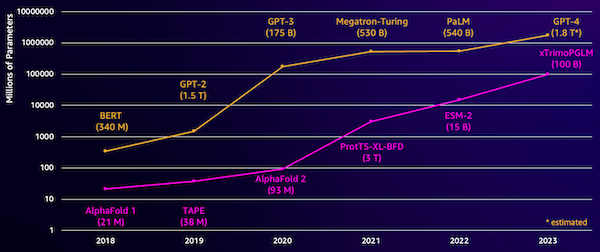
Långa utbildningstider, plus stora instanser, är lika med höga kostnader, vilket kan göra detta arbete utom räckhåll för många forskare. Till exempel, 2023, en forskargrupp beskrev träning av en pLM med 100 miljarder parametrar på 768 A100 GPU:er i 164 dagar! Som tur är kan vi i många fall spara tid och resurser genom att anpassa en befintlig pLM till vår specifika uppgift. Denna teknik kallas finjustering, och låter oss även låna avancerade verktyg från andra typer av språkmodellering.
Lösningsöversikt
Det specifika problemet vi tar upp i det här inlägget är subcellulär lokalisering: Givet en proteinsekvens, kan vi bygga en modell som kan förutsäga om den lever på utsidan (cellmembranet) eller inuti en cell? Detta är en viktig del av information som kan hjälpa oss att förstå funktionen och om den skulle vara ett bra läkemedelsmål.
Vi börjar med att ladda ner en offentlig datauppsättning med hjälp av Amazon SageMaker Studio. Sedan använder vi SageMaker för att finjustera ESM-2-proteinspråkmodellen med hjälp av en effektiv träningsmetod. Slutligen distribuerar vi modellen som en slutpunkt i realtid och använder den för att testa några kända proteiner. Följande diagram illustrerar detta arbetsflöde.

I följande avsnitt går vi igenom stegen för att förbereda din träningsdata, skapa ett träningsskript och köra ett SageMaker-utbildningsjobb. All kod som visas i det här inlägget är tillgänglig på GitHub.
Förbered träningsdata
Vi använder en del av DeepLoc-2 dataset, som innehåller flera tusen SwissProt-proteiner med experimentellt bestämda platser. Vi filtrerar efter högkvalitativa sekvenser mellan 100–512 aminosyror:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Därefter tokeniserar vi sekvenserna och delar upp dem i tränings- och utvärderingsset:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Slutligen laddar vi upp den bearbetade utbildnings- och utvärderingsdatan till Amazon enkel lagringstjänst (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Skapa ett träningsmanus
SageMaker skriptläge låter dig köra din anpassade utbildningskod i ramverkscontainrar för optimerad maskininlärning (ML) som hanteras av AWS. För detta exempel anpassar vi en befintligt skript för textklassificering från Hugging Face. Detta gör att vi kan prova flera metoder för att förbättra effektiviteten i vårt träningsjobb.
Metod 1: Viktad träningsklass
Liksom många biologiska datamängder är DeepLoc-data ojämnt fördelade, vilket innebär att det inte finns lika många membranproteiner som icke-membranproteiner. Vi kunde omsampla våra data och slänga poster från majoritetsklassen. Detta skulle dock minska den totala träningsdatan och potentiellt skada vår noggrannhet. Istället räknar vi ut klassvikterna under träningsjobbet och använder dem för att justera förlusten.
I vårt träningsmanus underklassar vi Trainer klass från transformers med en WeightedTrainer klass som tar hänsyn till klassvikter vid beräkning av korsentropiförlust. Detta hjälper till att förhindra partiskhet i vår modell:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMetod 2: Gradientackumulering
Gradientackumulering är en träningsteknik som gör att modeller kan simulera träning på större batchstorlekar. Vanligtvis begränsas batchstorleken (antalet prover som används för att beräkna gradienten i ett träningssteg) av GPU-minneskapaciteten. Med gradientackumulering beräknar modellen först gradienter på mindre partier. Sedan, istället för att uppdatera modellvikterna direkt, ackumuleras gradienterna över flera små batcher. När de ackumulerade gradienterna är lika med målets större batchstorlek, utförs optimeringssteget för att uppdatera modellen. Detta låter modeller träna med effektivt större partier utan att överskrida GPU-minnesgränsen.
Det krävs dock extra beräkning för de mindre partierna framåt och bakåt. Ökade batchstorlekar via gradientackumulering kan bromsa träningen, speciellt om för många ackumuleringssteg används. Syftet är att maximera GPU-användningen men att undvika överdrivna nedgångar från för många extra steg för beräkning av gradientberäkningar.
Metod 3: Gradientkontroll
Gradient checkpointing är en teknik som minskar minnet som behövs under träning samtidigt som beräkningstiden hålls rimlig. Stora neurala nätverk tar upp mycket minne eftersom de måste lagra alla mellanvärden från det framåtgående passet för att kunna beräkna gradienterna under det bakåtgående passet. Detta kan orsaka minnesproblem. En lösning är att inte lagra dessa mellanvärden utan då måste de räknas om under bakåtpassningen, vilket tar mycket tid.
Gradient checkpointing ger ett balanserat tillvägagångssätt. Den sparar bara några av de mellanliggande värdena, kallad checkpoints, och räknar om de andra efter behov. Därför använder den mindre minne än att lagra allt, men också mindre beräkning än att räkna om allt. Genom att strategiskt välja vilka aktiveringar som ska kontrolleras, gör gradientkontrollpunkten det möjligt för stora neurala nätverk att tränas med hanterbar minnesanvändning och beräkningstid. Denna viktiga teknik gör det möjligt att träna mycket stora modeller som annars skulle stöta på minnesbegränsningar.
I vårt träningsskript aktiverar vi gradientaktivering och checkpointing genom att lägga till nödvändiga parametrar till TrainingArguments objekt:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Metod 4: Lågrankad anpassning av LLM
Stora språkmodeller som ESM-2 kan innehålla miljarder parametrar som är dyra att träna och köra. Forskare utvecklat en träningsmetod som heter Low-Rank Adaptation (LoRA) för att göra finjusteringen av dessa enorma modeller mer effektiv.
Nyckeltanken bakom LoRA är att när du finjusterar en modell för en specifik uppgift behöver du inte uppdatera alla ursprungliga parametrar. Istället lägger LoRA till nya mindre matriser till modellen som transformerar in- och utdata. Endast dessa mindre matriser uppdateras under finjustering, vilket är mycket snabbare och använder mindre minne. De ursprungliga modellparametrarna förblir frusna.
Efter finjustering med LoRA kan du slå ihop de små anpassade matriserna tillbaka till den ursprungliga modellen. Eller så kan du hålla dem åtskilda om du snabbt vill finjustera modellen för andra uppgifter utan att glömma tidigare. Sammantaget tillåter LoRA att LLM:er effektivt kan anpassas till nya uppgifter till en bråkdel av den vanliga kostnaden.
I vårt träningsskript konfigurerar vi LoRA med hjälp av PEFT bibliotek från Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Skicka in ett SageMaker-utbildningsjobb
När du har definierat ditt träningsskript kan du konfigurera och skicka in ett SageMaker-utbildningsjobb. Ange först hyperparametrarna:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Definiera sedan vilka mätvärden som ska hämtas från träningsloggarna:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Definiera slutligen en Hugging Face-estimator och skicka in den för träning på en ml.g5.2xlarge-instanstyp. Detta är en kostnadseffektiv instanstyp som är allmänt tillgänglig i många AWS-regioner:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)Följande tabell jämför de olika träningsmetoderna vi diskuterade och deras effekt på körtid, noggrannhet och GPU-minneskrav för vårt jobb.
| konfiguration | Fakturerbar tid (min) | Utvärderingsnoggrannhet | Max GPU-minneanvändning (GB) |
| Basmodell | 28 | 0.91 | 22.6 |
| Bas + GA | 21 | 0.90 | 17.8 |
| Bas + GC | 29 | 0.91 | 10.2 |
| Bas + LoRA | 23 | 0.90 | 18.6 |
Alla metoder producerade modeller med hög utvärderingsnoggrannhet. Genom att använda LoRA och gradientaktivering minskade körtiden (och kostnaden) med 18 % respektive 25 %. Användning av gradientcheckpointing minskade den maximala GPU-minnesanvändningen med 55 %. Beroende på dina begränsningar (kostnad, tid, hårdvara) kan en av dessa metoder vara mer meningsfull än en annan.
Var och en av dessa metoder fungerar bra i sig, men vad händer när vi använder dem i kombination? Följande tabell sammanfattar resultaten.
| konfiguration | Fakturerbar tid (min) | Utvärderingsnoggrannhet | Max GPU-minneanvändning (GB) |
| Alla metoder | 12 | 0.80 | 3.3 |
I det här fallet ser vi en minskning av noggrannheten med 12 %. Vi har dock minskat körtiden med 57 % och GPU-minnesanvändningen med 85 %! Detta är en massiv minskning som gör att vi kan träna på ett brett utbud av kostnadseffektiva instanstyper.
Städa upp
Om du följer med i ditt eget AWS-konto, radera eventuella slutpunkter och data i realtid som du skapat för att undvika ytterligare avgifter.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Slutsats
I det här inlägget visade vi hur man effektivt finjusterar proteinspråksmodeller som ESM-2 för en vetenskapligt relevant uppgift. För mer information om att använda Transformers- och PEFT-biblioteken för att träna pLMS, kolla in inläggen Djupt lärande med proteiner och ESMBind (ESMB): Lågrankad anpassning av ESM-2 för förutsägelse av proteinbindningsställen på bloggen Hugging Face. Du kan också hitta fler exempel på att använda maskininlärning för att förutsäga proteinegenskaper i Fantastisk proteinanalys på AWS GitHub-förvar.
Om författaren
 Brian Loyal är senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet på Amazon Web Services. Han har mer än 17 års erfarenhet av bioteknik och maskininlärning, och brinner för att hjälpa kunder att lösa genomiska och proteomiska utmaningar. På fritiden tycker han om att laga mat och äta med sina vänner och familj.
Brian Loyal är senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet på Amazon Web Services. Han har mer än 17 års erfarenhet av bioteknik och maskininlärning, och brinner för att hjälpa kunder att lösa genomiska och proteomiska utmaningar. På fritiden tycker han om att laga mat och äta med sina vänner och familj.
- SEO-drivet innehåll och PR-distribution. Bli förstärkt idag.
- PlatoData.Network Vertical Generative Ai. Styrka dig själv. Tillgång här.
- PlatoAiStream. Web3 Intelligence. Kunskap förstärkt. Tillgång här.
- Platoesg. Kol, CleanTech, Energi, Miljö, Sol, Avfallshantering. Tillgång här.
- PlatoHealth. Biotech och kliniska prövningar Intelligence. Tillgång här.
- Källa: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



