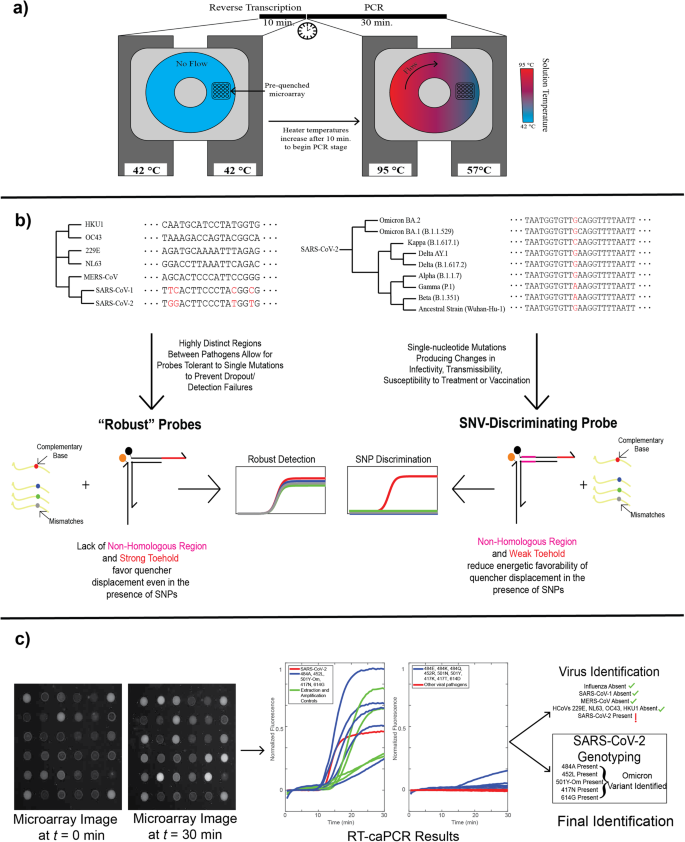
Integratie van reverse transcriptie in caPCR
We introduceerden eerst een RT-stap in de standaard caPCR-test. capPCR wordt uitgevoerd met behulp van een set van 4 verwarmingselementen die aan weerszijden van een patroon klemmen met daarin een toroïdale kamer gevuld met het PCR-reactiemengsel4. Door verwarmingselementen aan de ene kant van de kamer in te stellen op de PCR-annealtemperatuur en de andere op de denaturatietemperatuur, wordt een gradiënt tot stand gebracht, waardoor een passieve convectieve stroom wordt geproduceerd die PCR-amplificatie aanstuurt. De vooraf uitgedoofde reeks sondes wordt geïmmobiliseerd op het oppervlak van de cartridge en reageert via TMSD terwijl specifieke PCR-producten worden gesynthetiseerd. Om een initiële omgekeerde transcriptiestap mogelijk te maken, hebben we de verwarmers geprogrammeerd om gedurende 42 minuten een constante temperatuur van 10 ° C te handhaven voor cDNA-synthese, waarna de verwarmers zich aanpassen aan respectievelijk 57 ° C en 95 ° C om convectieve stroming te initiëren (Fig. 2a). Er wordt gebruik gemaakt van een RT-PCR-benadering met één pot om het aantal handmatige handelingen in de workflow te verminderen. Toehold probe-gemedieerde detectie op het caPCR-platform functioneert optimaal onder asymmetrische PCR-omstandigheden: een overmaat van de primer die de streng produceert die de quencher verdringt, genereert een sterker signaal op de fluorescente probe-array dan een 1:1 voorwaartse naar achterwaartse primerverhouding (Afb. S1). We hebben ervoor gezorgd dat dezelfde primer ook de cDNA-synthese van RNA-doelen medieert om de noodzaak te vermijden om afzonderlijke primers te introduceren voor reverse transcriptie- en PCR-stadia.
a De caPCR-workflow kan worden uitgebreid met een omgekeerde transcriptiestap (RT-caPCR) door de toevoeging van een reverse transcriptase-enzym en een speciale RT-fase waarbij alle apparaatverwarmers zijn ingesteld op de RT-temperatuur (42 °C). De temperaturen van de verwarming worden vervolgens verhoogd tot verschillende temperaturen voor denaturatie (95 °C) en annealing (57 °C), waardoor convectieve stroming en PCR-amplificatie van de cDNA-matrijzen worden geïnduceerd. b Het energetische ontwerpschema van probes, gebaseerd op DNA-bindende thermodynamica, kan worden gebruikt om tegemoet te komen aan twee behoeften in de diagnostiek van infectieziekten: I) De detectie van virale pathogenen moet robuust zijn tegen kleine veranderingen in de nucleotidesequentie om uitval te voorkomen als er nieuwe varianten ontstaan. II) Variantdiscriminatie vereist resolutie van één nucleotide. Variatie van de energieën van het steunpunt, het domein en de niet-homologe regio (NHR) kunnen worden gebruikt om aan beide vereisten te voldoen. c Voorbeeldgegevensuitvoer van een donut RT-caPCR-run. Beelden van de probe-array worden elke 30 seconden vastgelegd gedurende 30 minuten caPCR. Fluorescentiesporen worden geëxtraheerd en geanalyseerd om te identificeren welke luchtwegpathogenen aanwezig zijn en, indien van toepassing, welke variant van SARS-CoV-2 aanwezig is.
Om afstembare probes te ontwikkelen met programmeerbare sequentieselectiviteit, variërend van mutatiegevoelig tot mutatietolerant, hebben we twee sets energetische criteria voor teenhold-probes gebruikt (Fig. 2b). Robuuste probes hebben een sterker teensteungebied en een overeenkomstig zwakker domein om de algehele invloed van een enkele mutatie op de initiatie van de bindingsreactie en de daaropvolgende verplaatsing van de quencher-arm te verminderen. Voor SNV-discriminatie wordt de lengte van de steun ingekort en wordt een niet-homologe regio (NHR) geïntroduceerd die geen complementariteit met het doelwit heeft. Deze combinatie dient om de algehele energetische voorkeur voor het binden van de teen en de verplaatsing van de quencher-arm te verminderen, waardoor extra gewicht wordt gegeven aan afzonderlijke mismatches in de doelsequentie.
We hebben RT-caPCR toegepast met zowel robuuste als SNV-onderscheidende sondes voor de detectie van respiratoire virussen. Probe-arraybeelden van het RT-caPCR-systeem worden verwerkt met behulp van een aangepast MATLAB-script om fluorescentiesporen te produceren die vervolgens worden geïnterpreteerd om zowel de aanwezige virale doelwitten als, indien van toepassing, de variant van SARS-CoV-2 te identificeren. Een voorbeeld wordt getoond voor de SARS-CoV-2 Omicron-variant in Fig. 2c. Onze RT-caPCR-test met één pot demonstreert dus zijn vermogen om tegelijkertijd zowel gevoelige detectie met robuuste probes als single-nucleotide resolutie met SNV-discriminerende probes uit te voeren om in een enkele reactie nauwkeurig RNA-doelen van belang te identificeren.
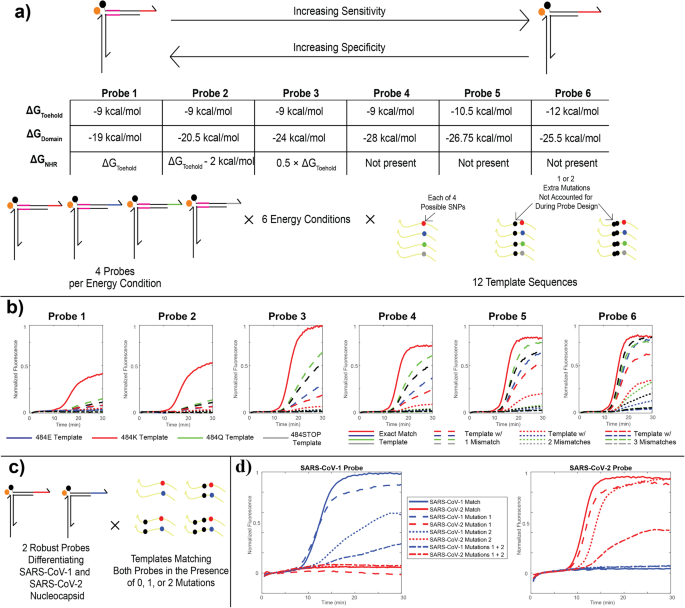
Het verfijnen van de energieën tussen robuuste en SNV-onderscheidende sondes
Om de diversiteit van de doelen die in onze RT-caPCR-test worden gedetecteerd te vergroten, hebben we de energie van de binding van de teen en de verplaatsing van de strengen geoptimaliseerd om de afweging tussen gevoeligheid en specificiteit te moduleren. Als casestudy hebben we het SARS-CoV-2 '484E'-genotype gebruikt als sjabloon voor onze energetische beoordeling. Mutaties op deze plaats zijn geassocieerd met verminderde neutraliserende activiteit van gastheerantilichamen12, en 3 van de 4 mogelijke nucleotidebasen (adenine, guanine en cytosine) worden gevonden op de eerste positie van het codon in natuurlijk ontwikkelde SARS-CoV-2-varianten9. Uracil-substitutie op deze plaats zou een stopcodon produceren en is dus niet in de natuur waargenomen, maar vanwege de nauwkeurigheid hebben we een thymine-aandoening in onze beoordeling opgenomen. We ontwierpen een probe voor elk van de 4 mogelijke nucleotiden op deze locus en stelden 6 verschillende thermodynamische omstandigheden vast op basis van de bindingsenergie (ΔGo) van de 3 regio's van de teensteunsonde (NHR, domein en teensteun); Fig. 3azie Materialen en methoden). Deze energetische omstandigheden variëren van de meest specifieke (Probe 1), waarin de energie van de NHR en de voetsteun ongeveer gelijk zijn (ongeveer -9 kcal/mol), tot de meest gevoelige (Probe 6), waarin er geen NHR is en de steun is iets sterker (−12 kcal / mol). In alle gevallen wordt de domeinbindingsenergie aangepast om de totale probebindingsenergie constant op ongeveer -40 kcal/mol te houden.
a Ontwerpparameters voor energetische karakterisering van sondes. Er werden zes verschillende energetische schema's ontworpen door de bindingsenergieën van de teenhold-, domein- en NHR-regio's te variëren, om een afweging mogelijk te maken tussen specificiteit van één nucleotide en gevoeligheid in de vorm van robuustheid voor mutaties. Voor elk van de zes energetische ontwerpen werden vier verschillende probes ontworpen, die alle vier mogelijke codons op de 484-mutatieplaats in het SARS-CoV-2-spike-eiwit bestrijken, waarvan er drie in varianten zijn waargenomen. Elke probe werd getest met 12 verschillende plasmide-templates, waaronder de 4 unieke single-nucleotide-mutaties alleen en in de aanwezigheid van een of twee andere mutaties die verder stroomopwaarts in de Omicron-variant werden gevonden. b caPCR is het resultaat van de 484 K-serie probes. Rode, ononderbroken curven geven de prestaties op het doel aan, zonder mutaties. Stippellijnen, stippellijnen en stippellijnen geven sjablonen aan met respectievelijk 1, 2 of 3 mismatches ten opzichte van de probesequentie. De kleur specificeert het codon op de 484-site (E, K, Q of STOP). c Schema voor het testen van robuuste SARS-CoV-1/2-discriminatie. Er zijn twee sondes ontworpen volgens het energetische schema “Probe 5” gedeeltelijk a voor een regio van het nucleocapsidegen in SARS-CoV-1 en SARS-CoV-2. Beide probes werden getest in de aanwezigheid van de exact passende matrijs en varianten die één of beide van twee stroomopwaartse misparingen omvatten, evenals in de aanwezigheid van de exact passende matrijs en niet-overeenkomende varianten van de andere probe. d Resultaten van de tests beschreven in (c). Ononderbroken lijnen, stippellijnen, stippellijnen en stippellijnen geven exacte match-sjablonen aan, sjablonen met een van de twee SNV's, of sjablonen met beide SNV's ten opzichte van het SARS-CoV-1 (blauw) of SARS-CoV-2 (rood) genoom.
We hebben deze probes getest met 12 verschillende DNA-sjablonen, waarvan er 4 de exacte match-sjabloon vertegenwoordigen voor elk van de 4 mogelijke basen in het 484-codon. Om de afstembaarheid van onze probe-energetica aan te tonen, hebben we mutaties genomen die ontstonden in latere lijnen van de SARS-CoV-2-evolutie en deze in de sjablonen geïntroduceerd: de T478K-mutatie gevonden in de Delta AY- en Omicron-lijnen, en de S477N-mutatie gevonden in Omicron. geslachten (schematisch Fig. 3a)10. Alle 24 probes werden geëvalueerd als reactie op de amplificatie van elk van de 12 templates. Figuur 3b toont een subset van deze resultaten, specifiek voor de probes die zijn ontworpen om zich te richten op de 484 K-mutatie die wordt aangetroffen in bèta- en gamma-VOC's10. Naarmate de sterkte van de NHR afneemt en de steun toeneemt, blijft de detectie voor het exact overeenkomende sjabloon (ononderbroken rode lijn) sterk en wordt de sonde gevoeliger voor sjablonen met 1 (stippellijnen) en 2 (stippellijnen) mismatches. Dit demonstreert ons vermogen om energieën te onderzoeken voor zeer specifieke doelbindende resultaten. Op basis van deze resultaten hebben we energetisch schema 2 geselecteerd voor SNV-onderscheidende sondes en schema 5 voor robuuste sondes.
We hebben twee robuuste probes ontworpen die zich richten op de nucleocapside-gensequentie van SARS-CoV-2 en SARS-CoV-1. Om ervoor te zorgen dat deze pathogeensequenties konden worden gedifferentieerd met behulp van robuuste probes terwijl ze tolerant bleven voor mutaties, hebben we 2 sjablonen bedacht die ofwel geen mismatches, 8 van de 1 mismatches, of beide mismatches samen bevatten (Fig. 3czie Materialen en methoden). De regio die is geselecteerd voor SARS-CoV-1- en SARS-CoV-2-detectie verschilt tussen de twee doelsequenties met 2 mutaties: als zodanig werden mismatches in de template geselecteerd op basis van deze verschillen. Als zodanig heeft de SARS-CoV-8-sjabloon met twee mismatches slechts zes verschillen ten opzichte van de SARS-CoV-2-sequentie, wat een real-life toepassing benadrukt van het belang van nauwkeurige afstemming van probes op gevoeligheid versus specificiteit. Figuur 3d toont de resultaten van deze tests. In alle gevallen hebben we geen kruisreactiviteit waargenomen tussen de SARS-CoV-1- en SARS-CoV-2-sondes en -sjablonen: SARS-CoV-1-RNA werd alleen gedetecteerd door de SARS-CoV-1-sonde, en SARS-CoV-2 RNA werd alleen gedetecteerd door de SARS-CoV-2-sonde. De templates die een van de twee mutaties bevatten, kunnen ook ondubbelzinnig worden gedetecteerd door elke geschikte probe, waarbij één van de twee mutaties de prestaties duidelijker beïnvloedt dan de andere. Op dezelfde manier worden sjablonen met beide mutaties samen nog steeds gedetecteerd, hoewel de prestaties enigszins in het gedrang komen als gevolg van de minder energetische voorkeur bij de verplaatsing van de quencherarm.
Door middel van onze gerichte onderzoeken naar probe-energetica hebben we ons vermogen gedemonstreerd om probes zo nauwkeurig te ontwerpen dat ze de gewenste afweging tussen gevoeligheid en specificiteit hebben. SNV-discriminerende probes hebben een energie die een exacte match vereist voor de verplaatsing van de quencher-arm, terwijl robuuste probes meer promiscue zijn in het binden van doelen met het vermogen om een amplicon te detecteren dat maximaal twee mutaties bevat. Het energetische bereik dat we hebben onderzocht, kan worden toegepast om sondes indien nodig aan te passen van energieschema 1 naar energieschema 6 om detectie van doelen met het gewenste aantal mismatches uit de sondesequentie mogelijk te maken.
Prestaties van robuuste en SNV-discriminerende sondes
Om het proof-of-concept voor ons zeer afstembare sondeontwerp te demonstreren, hebben we een volledig panel van zowel robuuste als SNV-discriminerende sondes gecreëerd. Ons panel benadrukt het nut van robuuste sondes door te screenen op alle 7 coronavirussen waarvan bekend is dat ze mensen infecteren (HCoVs 229E, HKU1, NL63 en OC43; MERS-CoV, SARS-CoV-1 en SARS-CoV-2) en griep A en B8. Bovendien demonstreert ons panel de functionaliteit van SNV-discriminerende probes door gebruik te maken van 14 probes en 3 amplicons die 5 algemeen gemuteerde locaties in het SARS-CoV-2-spike-gen bestrijken om differentiatie tussen zorgwekkende varianten mogelijk te maken (417, 452, 484, 501 en 614; afb. S2)9,10,11.
Om de prestaties van de robuuste probes in onze RT-caPCR-test voor gevoelige doeldetectie te beoordelen, hebben we 65 RT-caPCR-tests uitgevoerd met behulp van coronavirus-negatieve virale transportmedia (VTM)-monsters die een reeks demografische patiëntgegevens bestrijken (tabellen S1-S4) verrijkt met synthetisch RNA van elk van de 7 menselijke coronavirussen, ofwel gekocht bij Twist Bioscience of intern gesynthetiseerd met behulp van in vitro transcriptie (IVT) van genfragmenten (Tabel S5). RNA werd geïsoleerd en gezuiverd uit deze kunstmatige klinische monsters met behulp van een op kralen gebaseerde extractiemethode13 vanwege de prioriteit voor het gebruik van een dergelijke methodologie voor RNA-extractie in diagnostische workflows voor SARS-CoV-214 en werd gebruikt als input voor ons systeem. Deze test omvatte ook zowel een extractiecontrole (RNA van MS2-bacteriofaag dat voorafgaand aan de zuivering in het gekunstelde monster werd toegevoegd) als een amplificatiecontrole (interne positieve controle (IPC) RNA dat in het RT-caPCR-reactiemengsel werd toegevoegd). Tenzij anders aangegeven, ontvingen de experimenten hier ongeveer 106 kopieën van MS2-RNA en 107 kopieën van viraal RNA. De RT-caPCR-reactie werd in 40 minuten uitgevoerd met behulp van 5,000 moleculen IPC-controle-RNA, het geëxtraheerde monster-RNA en 11 primerparen die alle doelen en controles bedekten.
RT-caPCR-gegevens werden geanalyseerd om de virale doelwitten te identificeren die in elk monster aanwezig waren, en, in het geval van SARS-CoV-2, welke specifieke variant aanwezig was (Fig. 4). Figuur 4a toont een representatieve run met de SARS-CoV-2 Omicron-variant als RNA-doelwit. Alle robuuste probes voor virale discriminatie worden in rood weergegeven, terwijl SNV-onderscheidende probes in blauw worden weergegeven. De extractie- en amplificatieregelaars worden groen weergegeven. Positieve en negatieve probespots komen overeen met volledig niet-uitgedoofde of volledig uitgedoofde probes met behulp van sequenties waarvoor geen primers of matrijs waren opgenomen en worden in grijs weergegeven. Dit type gegevens kan worden samengevat in een gecomprimeerde grafiek, zoals weergegeven in figuur XNUMX. 4b: alle verwachte positieve curven worden weergegeven in de linkergrafiek, en alle verwachte negatieve curven worden weergegeven in de rechtergrafiek. Hoewel er een klein niveau van off-target-activiteit is op sommige SNV-specifieke probes, kan het vergelijken van de on-target SNV-specifieke probe-curven (484 A, 452 L, 501Y-Om, 417 N en 614 G) met andere SNV-specifieke probes worden vergeleken. specifieke sondes bevestigen dat de on-target-sondes in alle gevallen het sterkst reageren, zoals beoordeeld met het oog en via ons op fluorescentiederivaat gebaseerde SNV-discriminatie-algoritme (zie Materialen en methoden en aanvullende methoden). We merken dit op vanwege de hoge mutatielast van de Omicron-variant in vergelijking met andere varianten11hebben we de sequentie van de 484A-sonde zo ontworpen dat deze ook S477N- en T478K-mutaties omvat die in deze variant worden gevonden om de detectie te verbeteren10. Op dezelfde manier omvat een unieke 501Y-Om-probe Q493R-, G496S- en Q498R-mutaties die specifiek worden aangetroffen in de Omicron-variant. Een probe specifiek voor de BA.2-subvariant van de Omicron-lijn (501Y-Om2) werd later geïntroduceerd. Deze probe is identiek aan de 501Y-Om-sequentie, maar mist de G496S-mutatie10.
a Voorbeeldresultaten van een RT-caPCR-run waarbij de zorgwekkende SARS-CoV-2 Omicron-variant wordt gedetecteerd. Robuuste virale detectiesondes zijn rood, SNV-onderscheidende sondes zijn blauw, extractie- en amplificatiecontroles zijn groen en positieve en negatieve controles zijn grijs. Elke curve vertegenwoordigt de intensiteit van de corresponderende probe-array-vlek op elk tijdstip over de 60 verkregen beelden, genormaliseerd naar de helderste plek in de array. b Samenvattende resultaten die overeenkomen met de inloop a. Alle verwachte positieve resultaten (extractie- en amplificatiecontroles, virale identificatie op het doel en alle SNV-discriminerende probes op het doel) worden weergegeven in de linkergrafiek, terwijl alle verwachte negatieve probes (alle andere virale en SNV-discriminerende probes, zoals evenals de negatieve controles) worden weergegeven in de rechtergrafiek. c Virale detectieresultaten voor het menselijke coronavirus 229E, het Middle East Respiratory Syndrome coronavirus (MERS-CoV) en SARS-CoV-1 in het formaat beschreven in (b). d. SNV-discriminatie is het resultaat van een SARS-CoV-2 Delta-variantsjabloon in het hierboven gespecificeerde formaat.
Figuur 4c toont gecomprimeerde representatieve resultaten van 3 runs voor 3 verschillende virale pathogeensjablonen: menselijk coronavirus 229E, MERS-CoV en SARS-CoV-1. Alle drie de monsters tonen een duidelijke detectie van het virale RNA-doelwit met de robuuste pathogeen-identificerende probes, geen off-target virale identificatieprobe-activiteit en de aanwezigheid van alle controles.
tafel 1 vat de virale identificatieresultaten van alle geldige onderzoeken samen. Competitie om PCR-reactiebronnen (bijv. polymerase, dNTP's) kan de amplificatie van doelen met een lager aantal kopieën onderdrukken, dus een testrun werd als geldig beschouwd als een viraal pathogeen werd gedetecteerd en ten minste 1 van de 2 controles (MS2 en IPC) werd gedetecteerd , of er werd geen viraal pathogeen gedetecteerd, maar beide controles werden gedetecteerd. 11 van de monsters waren negatieve controles, waaronder alleen MS2- en IPC-controle-RNA's. In alle elf van deze monsters werden zowel IPC als MS11 gedetecteerd (2%). Er werden 100 van de 36 SARS-CoV-36-monsters geïdentificeerd (2%), zoals beoordeeld door detectie van de SARS-CoV-100-nucleocapsidesonde. De overige menselijke coronavirussen (2E, NL229, OC63 en HKU43) en SARS-CoV-1- en MERS-CoV-doelen vertonen ook een hoge gevoeligheid, met 1% detectie voor alle doelen. Zowel het proces van primerbinding als de door amplicon gemedieerde quencher-verplaatsing van de probes dragen bij aan de specificiteit van het panel, die voor alle doelen 100% was. Hier wordt de specificiteit berekend als het aandeel echt-negatieven dat correct is gedetecteerd. Kortom, geen enkele template induceerde detectie bij een andere robuuste probe dan degene waarvoor deze specifiek was ontworpen.
Figuur 4d toont een representatieve run die het vermogen van onze RT-caPCR-test benadrukt om zowel het RNA-doelwit te identificeren als een resolutie van één nucleotide te bieden door de detectie van virale varianten gelijktijdig met pathogeenidentificatie voor de SARS-CoV-2 Delta-variant te tonen. Hoewel de 452 L-sonde aanzienlijke activiteit buiten het doel vertoont, is de detectie ervan duidelijk minder efficiënt dan de 452 R-sonde op het doel (de blauwe curve met de hoogste intensiteit in de linkergrafiek). SNV-onderscheidende probes maken dus een duidelijk onderscheid mogelijk tussen SARS-CoV-2-varianten die slechts door een paar enkele mutaties verschillen.
tafel 2 vat de prestaties samen van de SNV-specifieke sondes voor SARS-CoV-2-varianten in 36 onderzoeken. Het aantal verwachte detecties is gelijk aan het aantal runs waarvoor een sjabloon met die mutatie is opgenomen, terwijl het aantal verwachte negatieven het aantal runs is waarvoor het overeenkomstige sjabloon niet is opgenomen. Echte positieven zijn runs waarvoor de probe van belang de dominante SNV-specifieke probe voor die mutatieplaats was (zoals algoritmisch beoordeeld), en echte negatieven zijn die runs waarvoor dit niet het geval was. In één run merken we op dat de 614G-sonde werd verduisterd door een vreemd deeltje (stof), dus het totaal voor deze doelen is 35 in plaats van 36. Afgezien van 484E vertonen alle sondes een gevoeligheid van ten minste 90%. Slechte prestaties van de 484E-probe kunnen gedeeltelijk worden verklaard door de aanwezigheid van een SNV (T478K-mutatie) in het amplicon voor de Delta-varianten. Alle andere probes vertonen een specificiteit van 100%, omdat een amplicon altijd bij voorkeur zijn overeenkomende quencher zal verdringen boven een probe met een mutatie, ervan uitgaande dat de energieën op het doel gelijk zijn.
Samen benadrukken deze gegevens het vermogen van robuuste probes en SNV-discriminerende probes om samen te werken om snel het aanwezige RNA-doelwit te identificeren (met de robuuste probe) en resolutie van één nucleotide te bieden (met de SNV-discriminerende probe).
Evaluatie van de detectielimiet van RT-caPCR
Vervolgens voerden we een reeks experimenten uit om de detectielimiet van onze RT-caPCR-test te bepalen. Bij alle voorgaande tests in Fig. 4, een virale input van 107 moleculen werden voorafgaand aan de extractie in de VTM toegevoegd, vergelijkbaar met schattingen van de piekvirale belasting voor Omicron- en Delta-patiënten15,16. Hier hebben we de LOD van onze test gekwantificeerd door het aantal invoerkopieën van viraal RNA te variëren en de controlekopieën hetzelfde te houden. Met behulp van gezuiverd RNA, verkregen van Twist Bioscience of intern gesynthetiseerd uit genfragmenten via IVT, bereikten we een LOD van 1,000 moleculen voor alle 7 coronavirussen. Representatieve resultaten worden getoond in Fig. S3. In zowel afb. S3a en S3b, ontdekken we dat we met beide controles met succes tot 1,000 moleculen per reactie kunnen detecteren voor een enkel doelwit (HCoV 229E; Fig. S3a) en voor een SARS-CoV-2-variant met beide bedieningselementen (Omicron; Fig. S3b), een hoeveelheid die drie ordes van grootte lager is dan die gebruikt voor VTM-testen bij gebruik van gezuiverd RNA. Wanneer alleen de aanwezigheid van een enkel doelwit wordt gevarieerd, zoals in Fig. S3a, vinden we alleen voor dit doel een duidelijke vertraging in de drempeltijd voor elke orde van grootte, terwijl de twee controles consistent zijn over de runs heen. Deze trend is minder duidelijk voor SARS-CoV-2, wat te verwachten is aangezien dit doel gelijktijdige amplificatie van 2 amplicons en 4 controles vereist. Concurrentie om enzymatische hulpbronnen en dNTP's lijkt de duidelijke trend te verstoren die wordt waargenomen voor doelwitten met één amplicon. In afb. S3cwerd een verdunningsreeks uitgevoerd door de hoeveelheid input-RNA naar de extractiepijplijn voor de Omicron-variant te variëren. Ook hier zien we detectie tot aan de laagste geteste waarde (invoer van 10,000 moleculen in de RNA-extractiestap, terwijl ongeveer 1,000 moleculen in de RT-stap terechtkomen als gevolg van verliezen tijdens extractie en het niet gebruiken van het volledige elutievolume van 25 µL in de reactie) en een verwachte vertraging in amplificatie als functie van de inputconcentratie.
Detectie van meerdere doelen in één monster
Nadat we het succes hadden aangetoond van onze RT-caPCR-test met afstembare probes bij het identificeren van RNA-doelen met robuuste probes (elk virus) met specificiteit voor één nucleotide met SNV-discriminerende probes (SARS-CoV-2-varianten), hebben we vervolgens onderzocht hoe onze test presteerde toen het beoordelen van complexere inputs door co-infectiemonsters te simuleren die meerdere RNA-doelen bevatten. Hier een invoerbedrag van 105 moleculen van elke template werden rechtstreeks in de RT-caPCR-reactie toegevoegd zonder VTM-extractiestappen. We hebben ook onze RT-caPCR-test uitgebreid om te testen op meer RNA-doelen door 3 extra primersets en 4 probes toe te voegen om te testen op influenza A en B (twee gericht op het M2-matrixeiwitgen van influenza A17 en 2 gericht op niet-structureel proteïne-1 (nsp1) in de Yamagata- en Victoria-lijnen van influenza B18). Deze 14-plex-test werd getest op alle vier de nieuwe doelen om de werkzaamheid ervan te bevestigen (Fig. S4).
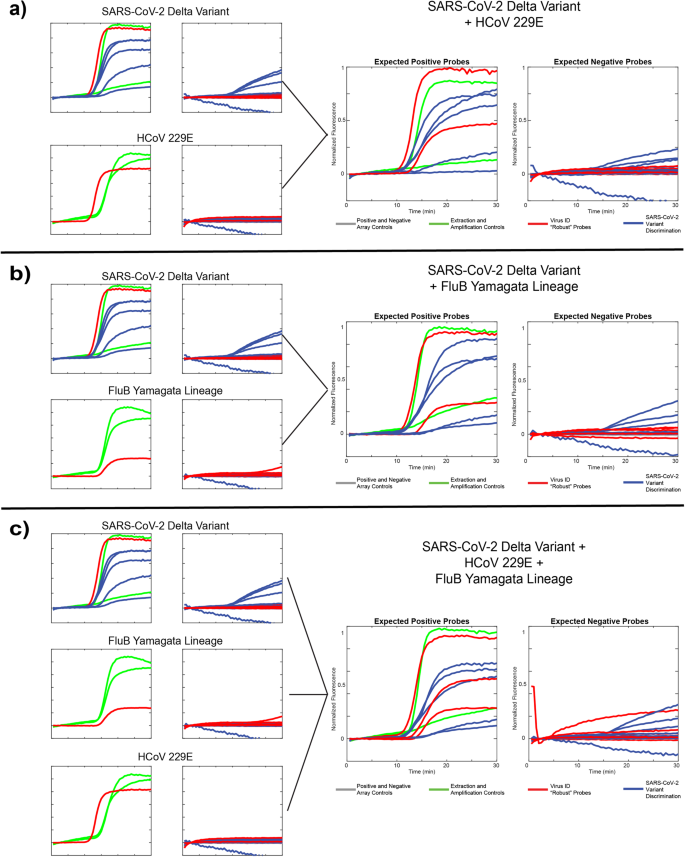
We hebben elk van de 3 verschillende virale templates afzonderlijk beoordeeld, evenals in 3 verschillende combinaties: SARS-CoV-2 Delta-variant + HCoV229E (Fig. 5a), SARS-CoV-2 Delta-variant + FluB Yamagata-afstamming (Fig. 5b), en SARS-CoV-2 + HCoV229E + FluB Yamagata-afstamming (Fig. 5c). Gegevens worden gepresenteerd als gecomprimeerde fluorescerende sporenbeelden zoals hierboven beschreven met verwachte positieve probes in het linkerpaneel en verwachte negatieve probes in het rechterpaneel. Bij alle runs hebben we het verwachte gedrag waargenomen: robuuste (virusidentificatie) probes rapporteren de aanwezigheid van alle aanwezige pathogeensjablonen, en SNV-discriminerende probes (variantidentificatie) identificeren de Delta-variant correct met lage niveaus van off-target probe-activiteit in alle runs. 5 SNV-sites. De 484E-sonde presteert het zwakst, waarschijnlijk als gevolg van de bovengenoemde mismatch in het domeingebied van deze sonde. Bovendien presteerde de MS2-extractiecontrole in sommige experimenten slecht als gevolg van de concurrentie om hulpbronnen tussen 8 verschillende amplicons. We merken ook op dat deze runs slechts 1 replica van zowel de MS2- als de IPC-besturingselementen bevatten, in plaats van 2 zoals in Fig. 4, aangezien dit een bijgewerkt paneel was inclusief de 501Y-Om2-sonde. Deze gegevens demonstreren het vermogen van onze technologie om nauwkeuriger complexere monsters te beoordelen die meerdere interessante RNA-doelen bevatten, wat de veelzijdigheid van de test aantoont.
a Resultaten met één sjabloon van de SARS-CoV-2 Delta-variant en het menselijke coronavirus 229E, samen met een ‘co-infectie’-monster dat beide sjablonen bevat. b Resultaten met één sjabloon van de SARS-CoV-2 Delta-variant en de Yamagata-lijn van influenza B, samen met een co-infectiemonster dat beide sjablonen bevat. c. Resultaten met één doel van dezelfde doelen als in a en b, samen met een co-infectiemonster dat alle drie de sjablonen bevat.
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- PlatoData.Network Verticale generatieve AI. Versterk jezelf. Toegang hier.
- PlatoAiStream. Web3-intelligentie. Kennis versterkt. Toegang hier.
- PlatoESG. carbon, CleanTech, Energie, Milieu, Zonne, Afvalbeheer. Toegang hier.
- Plato Gezondheid. Intelligentie op het gebied van biotech en klinische proeven. Toegang hier.
- Bron: https://www.nature.com/articles/s42003-023-05346-4