
Afbeelding door rawpixel on Freepik
Unsupervised learning is een tak van machine learning waarbij de modellen patronen leren van de beschikbare gegevens in plaats van voorzien van het daadwerkelijke label. We laten het algoritme de antwoorden bedenken.
Bij leren zonder toezicht zijn er twee hoofdtechnieken; clustering en dimensionaliteitsreductie. De clustertechniek gebruikt een algoritme om het patroon te leren om de gegevens te segmenteren. De dimensionaliteitsreductietechniek daarentegen probeert het aantal kenmerken te verminderen door de feitelijke informatie zoveel mogelijk intact te houden.
Een voorbeeldalgoritme voor clustering is K-Means en voor dimensionaliteitsreductie is PCA. Dit waren de meest gebruikte algoritmen voor leren zonder toezicht. We praten echter zelden over de maatstaven om leren zonder toezicht te evalueren. Hoe nuttig het ook is, we moeten het resultaat nog steeds evalueren om te weten of de uitvoer nauwkeurig is.
Dit artikel bespreekt de statistieken die worden gebruikt om algoritmen voor machinaal leren zonder toezicht te evalueren en zal in twee secties worden verdeeld; Clustering van algoritmestatistieken en dimensionaliteitsreductiestatistieken. Laten we erop ingaan.
We zullen niet in detail ingaan op het clusteralgoritme, aangezien dit niet het belangrijkste punt van dit artikel is. In plaats daarvan zouden we ons concentreren op voorbeelden van de maatstaven die voor de evaluatie zijn gebruikt en hoe het resultaat kan worden beoordeeld.
Dit artikel maakt gebruik van de Wijndataset van Kaggle als voorbeeld van onze dataset. Laten we eerst de gegevens lezen en het K-Means-algoritme gebruiken om de gegevens te segmenteren.
import pandas as pd
from sklearn.cluster import KMeans
df = pd.read_csv('wine-clustering.csv') kmeans = KMeans(n_clusters=4, random_state=0)
kmeans.fit(df)Ik initieer het cluster als 4, wat betekent dat we de gegevens in 4 clusters segmenteren. Is het het juiste aantal clusters? Of is er een geschikter clusternummer? Gewoonlijk kunnen we de techniek gebruiken die de elleboog methode: om de juiste cluster te vinden. Laat me de onderstaande code tonen.
wcss = []
for k in range(1, 11): kmeans = KMeans(n_clusters=k, random_state=0) kmeans.fit(df) wcss.append(kmeans.inertia_) # Plot the elbow method
plt.plot(range(1, 11), wcss, marker='o')
plt.xlabel('Number of Clusters (k)')
plt.ylabel('WCSS')
plt.title('Elbow Method')
plt.show() 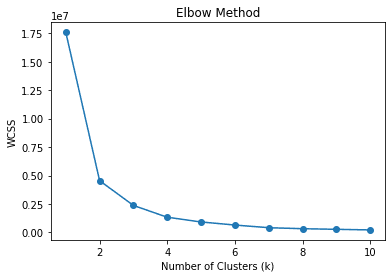
Bij de elleboogmethode gebruiken we WCSS of Within-Cluster Sum of Squares om de som van de gekwadrateerde afstanden tussen datapunten en de respectievelijke clusterzwaartepunten voor verschillende k (clusters) te berekenen. De beste k-waarde is naar verwachting degene met de meeste afname van WCSS of de elleboog in de bovenstaande afbeelding, namelijk 2.
We kunnen de elleboogmethode echter uitbreiden om andere statistieken te gebruiken om de beste k te vinden. Hoe zit het met het algoritme dat automatisch het clusternummer vindt zonder te vertrouwen op het zwaartepunt? Ja, we kunnen ze ook evalueren met vergelijkbare statistieken.
Merk op dat we een zwaartepunt kunnen aannemen als het gegevensgemiddelde voor elk cluster, ook al gebruiken we het K-Means-algoritme niet. Dus elk algoritme dat niet afhankelijk was van het zwaartepunt tijdens het segmenteren van de gegevens, zou nog steeds elke metrische evaluatie kunnen gebruiken die afhankelijk is van het zwaartepunt.
Silhouetcoëfficiënt
Silhouette is een techniek bij het clusteren om de overeenkomst van gegevens binnen het cluster te meten in vergelijking met het andere cluster. De Silhouette-coëfficiënt is een numerieke weergave van -1 tot 1. Waarde 1 betekent dat elk cluster volledig verschilde van de andere, en waarde -1 betekent dat alle gegevens aan het verkeerde cluster zijn toegewezen. 0 betekent dat er geen zinvolle clusters uit de gegevens zijn.
We kunnen de volgende code gebruiken om de Silhouette-coëfficiënt te berekenen.
# Calculate Silhouette Coefficient
from sklearn.metrics import silhouette_score sil_coeff = silhouette_score(df.drop("labels", axis=1), df["labels"])
print("Silhouette Coefficient:", round(sil_coeff, 3))Silhouetcoëfficiënt: 0.562
We kunnen zien dat onze segmentatie hierboven een positieve silhouetcoëfficiënt heeft, wat betekent dat er een mate van scheiding is tussen de clusters, hoewel er nog steeds enige overlapping is.
Calinski-Harabasz-index
De Calinski-Harabasz Index of Variance Ratio Criterion is een index die wordt gebruikt om de clusterkwaliteit te evalueren door de verhouding tussen spreiding tussen clusters en spreiding binnen clusters te meten. In feite hebben we de verschillen gemeten tussen de som in het kwadraat van de afstand van de gegevens tussen het cluster en gegevens binnen het interne cluster.
Hoe hoger de Calinski-Harabasz Index-score, hoe beter, wat betekent dat de clusters goed gescheiden waren. Er zijn echter geen bovengrenzen voor de score, wat betekent dat deze statistiek beter is voor het evalueren van verschillende k-getallen dan voor het interpreteren van het resultaat zoals het is.
Laten we de Python-code gebruiken om de Calinski-Harabasz Index-score te berekenen.
# Calculate Calinski-Harabasz Index
from sklearn.metrics import calinski_harabasz_score ch_index = calinski_harabasz_score(df.drop('labels', axis=1), df['labels'])
print("Calinski-Harabasz Index:", round(ch_index, 3))Calinski-Harabasz-index: 708.087
Een andere overweging voor de Calinski-Harabasz Index-score is dat de score gevoelig is voor het aantal clusters. Een hoger aantal clusters kan ook leiden tot een hogere score. Het is dus een goed idee om naast de Calinski-Harabasz-index andere statistieken te gebruiken om het resultaat te valideren.
Davies-Bouldin-index
De Davies-Bouldin-index is een clusteringsevaluatiemetriek die wordt gemeten door de gemiddelde overeenkomst tussen elk cluster en zijn meest vergelijkbare cluster te berekenen. De verhouding tussen afstanden binnen clusters en afstanden tussen clusters berekent de overeenkomst. Dit betekent dat hoe verder de clusters uit elkaar liggen en hoe minder verspreid, hoe beter de scores zullen zijn.
In tegenstelling tot onze eerdere statistieken, streeft de Davies-Bouldin-index naar een zo laag mogelijke score. Hoe lager de score was, hoe meer gescheiden elk cluster was. Laten we een Python-voorbeeld gebruiken om de score te berekenen.
# Calculate Davies-Bouldin Index
from sklearn.metrics import davies_bouldin_score dbi = davies_bouldin_score(df.drop('labels', axis=1), df['labels'])
print("Davies-Bouldin Index:", round(dbi, 3))Davies-Bouldin-index: 0.544
We kunnen niet zeggen dat de bovenstaande score goed of slecht is, omdat we, net als bij de vorige statistieken, het resultaat nog steeds moeten evalueren door verschillende statistieken als ondersteuning te gebruiken.
In tegenstelling tot clustering, heeft dimensionaliteitsreductie tot doel het aantal kenmerken te verminderen terwijl de originele informatie zoveel mogelijk behouden blijft. Daarom gingen veel van de evaluatiestatistieken bij dimensionaliteitsreductie allemaal over het behoud van informatie. Laten we de dimensionaliteit verminderen met PCA en kijken hoe de statistiek werkt.
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler #Scaled the data
scaler = StandardScaler()
df_scaled = scaler.fit_transform(df) pca = PCA()
pca.fit(df_scaled)In het bovenstaande voorbeeld passen we de PCA aan de gegevens aan, maar hebben we het nummer van de functie nog niet verlaagd. In plaats daarvan willen we de dimensionaliteitsreductie en variantie-afweging evalueren met de Cumulatief verklaarde variantie. Het is de algemene maatstaf voor dimensionaliteitsreductie om te zien hoe informatie behouden blijft bij elke kenmerkreductie.
#Calculate Cumulative Explained Variance
cev = np.cumsum(pca.explained_variance_ratio_) plt.plot(range(1, len(cev) + 1), cev, marker='o')
plt.xlabel('Number of PC')
plt.ylabel('CEV')
plt.title('CEV vs. Number of PC')
plt.grid() 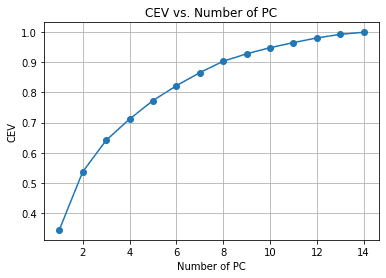
We kunnen in de bovenstaande grafiek zien hoeveel pc's zijn behouden in vergelijking met de verklaarde variantie. Als vuistregel kiezen we er vaak voor dat ongeveer 90-95% behouden blijft wanneer we dimensionaliteitsreductie proberen te maken, dus ongeveer 14 kenmerken worden teruggebracht tot 8 als we de bovenstaande tabel volgen.
Laten we naar de andere statistieken kijken om onze dimensionaliteitsreductie te valideren.
Betrouwbaarheid
Betrouwbaarheid is een meting van de kwaliteit van de dimensionaliteitsreductietechniek. Deze statistiek meet hoe goed de verkleinde dimensie de oorspronkelijke naaste buur van de gegevens behield.
Kortom, de metriek probeert te zien hoe goed de techniek voor dimensiereductie de gegevens heeft behouden bij het handhaven van de lokale structuur van de oorspronkelijke gegevens.
De metriek Betrouwbaarheid ligt tussen 0 en 1, waarbij waarden die dichter bij 1 liggen, betekenen dat de buur die zich dicht bij gegevenspunten met gereduceerde dimensie bevindt, meestal ook dichtbij is in de oorspronkelijke dimensie.
Laten we de Python-code gebruiken om de betrouwbaarheidsmetriek te berekenen.
from sklearn.manifold import trustworthiness # Calculate Trustworthiness. Tweak the number of neighbors depends on the dataset size.
tw = trustworthiness(df_scaled, df_pca, n_neighbors=5)
print("Trustworthiness:", round(tw, 3))Betrouwbaarheid: 0.87
Het in kaart brengen van Sammon
Sammon's mapping is een niet-lineaire dimensionaliteitsreductietechniek om de hoge-dimensionaliteit paarsgewijze afstand te behouden wanneer deze wordt verkleind. Het doel is om de functie Stress van Sammon te gebruiken om de paarsgewijze afstand tussen de oorspronkelijke gegevens en de reductieruimte te berekenen.
Hoe lager de stressfunctiescore van Sammon, hoe beter, omdat dit wijst op een betere paarsgewijze bewaring. Laten we proberen het Python-codevoorbeeld te gebruiken.
Eerst zouden we een extra pakket voor Sammon's Mapping installeren.
pip install sammon-mappingDan zouden we de volgende code gebruiken om de Sammon's stress te berekenen.
# Calculate Sammon's Stress
from sammon import sammon pca_res, sammon_st = sammon.sammon(np.array(df)) print("Sammon's Stress:", round(sammon_st, 5))Sammon's Stress: 1e-05
Het resultaat toonde een lage Sammon's Score, wat betekent dat de gegevensbehoud aanwezig was.
Unsupervised learning is een machine learning-tak die probeert het patroon uit de gegevens te leren. Vergeleken met begeleid leren, zal de outputevaluatie misschien niet veel zeggen. In dit artikel proberen we enkele leerstatistieken zonder toezicht te leren, waaronder:
- Binnen-Cluster Som Vierkant
- Silhouetcoëfficiënt
- Calinski-Harabasz-index
- Davies-Bouldin-index
- Cumulatief verklaarde variantie
- Betrouwbaarheid
- Het in kaart brengen van Sammon
Cornellius Yudha Wijaya is een data science assistent-manager en dataschrijver. Terwijl hij fulltime bij Allianz Indonesia werkt, deelt hij graag Python- en Data-tips via sociale media en schrijvende media.
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- Platoblockchain. Web3 Metaverse Intelligentie. Kennis versterkt. Toegang hier.
- De toekomst slaan met Adryenn Ashley. Toegang hier.
- Bron: https://www.kdnuggets.com/2023/04/exploring-unsupervised-learning-metrics.html?utm_source=rss&utm_medium=rss&utm_campaign=exploring-unsupervised-learning-metrics



