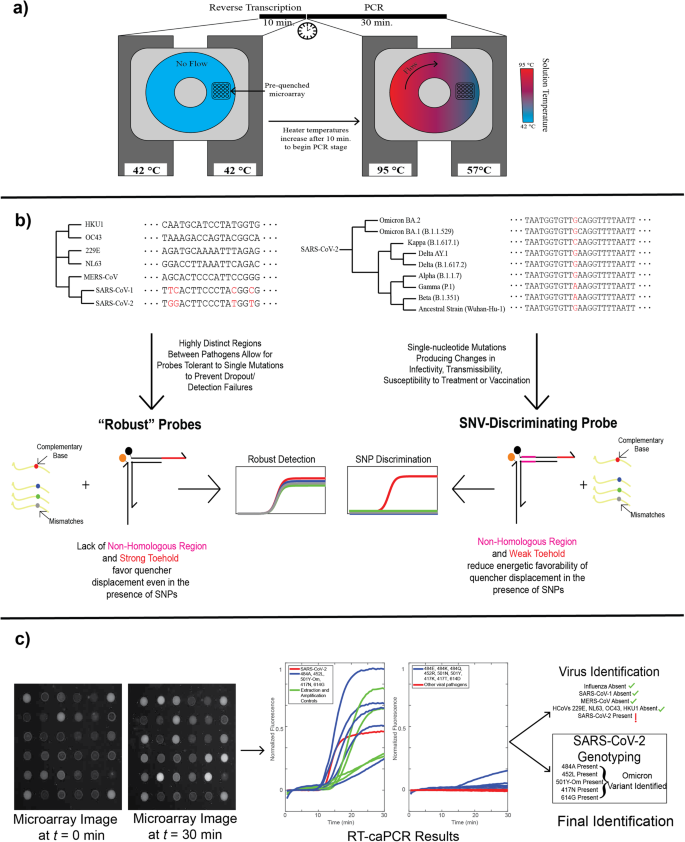
caPCR에 역전사 통합
우리는 먼저 표준 caPCR 분석에 RT 단계를 도입했습니다. caPCR은 PCR 반응 혼합물로 채워진 환상형 챔버가 포함된 카트리지의 양쪽 면을 고정하는 4개의 히터 세트를 사용하여 수행됩니다.4. 챔버 한쪽의 히터를 PCR 어닐링 온도로 설정하고 다른 쪽을 변성 온도로 설정하여 기울기가 설정되어 PCR 증폭을 유도하는 수동 대류 흐름이 생성됩니다. 사전 담금질된 발판 프로브 어레이는 카트리지 표면에 고정되어 있으며 특정 PCR 산물이 합성될 때 TMSD를 통해 반응합니다. 초기 역전사 단계를 수용하기 위해 우리는 cDNA 합성을 위해 42분 동안 10°C의 일정한 온도를 유지하도록 히터를 프로그래밍한 후 대류 흐름을 시작하기 위해 히터를 각각 57°C와 95°C로 조정했습니다(그림 XNUMX). 2a). 워크플로에서 수동 처리 단계 수를 줄이기 위해 원포트 RT-PCR 접근 방식이 사용됩니다. caPCR 플랫폼의 Toehold 프로브 매개 검출은 비대칭 PCR 조건에서 최적으로 작동합니다. 소광제를 대체하는 가닥을 생성하는 프라이머의 과잉은 1:1 정방향-역방향 프라이머 비율보다 형광 프로브 어레이에서 더 강한 신호를 생성합니다. (무화과. S1). 우리는 역전사 및 PCR 단계에 대해 별도의 프라이머를 도입할 필요가 없도록 동일한 프라이머가 RNA 표적으로부터 cDNA 합성도 매개한다는 것을 확인했습니다.
a caPCR 워크플로우는 역전사 효소를 추가하여 역전사 단계(RT-caPCR)를 포함하도록 확장할 수 있으며 모든 장치 히터가 RT 온도(42°C)로 설정된 전용 RT 단계를 포함할 수 있습니다. 그런 다음 히터 온도를 변성(95°C) 및 어닐링(57°C)을 위해 다른 온도로 높여 cDNA 템플릿의 대류 흐름과 PCR 증폭을 유도합니다. b DNA 결합 열역학을 기반으로 한 프로브의 에너지 설계 계획은 감염성 질환 진단의 두 가지 요구 사항을 해결하는 데 사용될 수 있습니다. I) 바이러스성 병원체 탐지는 새로운 변종이 발생할 경우 탈락을 방지하기 위해 뉴클레오티드 서열의 작은 변화에 강력해야 합니다. II) 변형 식별에는 단일 뉴클레오티드 분해가 필요합니다. 발판, 도메인 및 비동종 영역(NHR) 에너지의 변화를 사용하여 이러한 요구 사항을 모두 충족할 수 있습니다. c 도넛형 RT-caPCR 실행의 데이터 출력 예. 프로브 어레이 이미지는 30분의 caPCR 동안 30초마다 획득됩니다. 형광 흔적을 추출하고 분석하여 어떤 호흡기 병원체가 존재하는지, 그리고 해당되는 경우 SARS-CoV-2의 어떤 변종이 있는지 확인합니다.
돌연변이 민감성에서 돌연변이 내성에 이르기까지 프로그래밍 가능한 서열 선택성을 갖춘 조정 가능한 프로브를 개발하기 위해 우리는 발판 프로브에 대한 2세트의 에너지 기준을 활용했습니다(그림 XNUMX). 2b). 견고한 프로브는 더 강한 발판 영역과 이에 상응하는 더 약한 도메인을 특징으로 하여 결합 반응의 시작과 후속 소광제 팔 변위에 대한 단일 돌연변이의 전반적인 영향을 줄입니다. SNV 차별의 경우 발판의 길이가 줄어들고 표적에 대한 상보성이 부족한 NHR(비상동 영역)이 도입됩니다. 이 조합은 발가락 결합 및 소광제 팔 변위에 대한 전반적인 에너지 선호도를 감소시켜 표적 서열의 단일 불일치에 추가 가중치를 제공하는 역할을 합니다.
우리는 호흡기 바이러스 검출에 강력하고 SNV 식별 프로브를 모두 갖춘 RT-caPCR을 적용했습니다. RT-caPCR 시스템의 프로브 배열 이미지는 맞춤형 MATLAB 스크립트를 사용하여 처리되어 형광 흔적을 생성한 다음 해석되어 존재하는 바이러스 표적과 해당하는 경우 SARS-CoV-2 변종을 식별합니다. 그림 2에는 SARS-CoV-XNUMX Omicron 변종의 예가 나와 있습니다. 2c. 따라서 우리의 원팟 RT-caPCR 분석은 강력한 프로브를 사용한 민감한 검출과 SNV 식별 프로브를 사용한 단일 뉴클레오티드 분해능을 동시에 수행하여 단일 반응에서 관심 있는 RNA 표적을 정확하게 식별하는 능력을 보여줍니다.
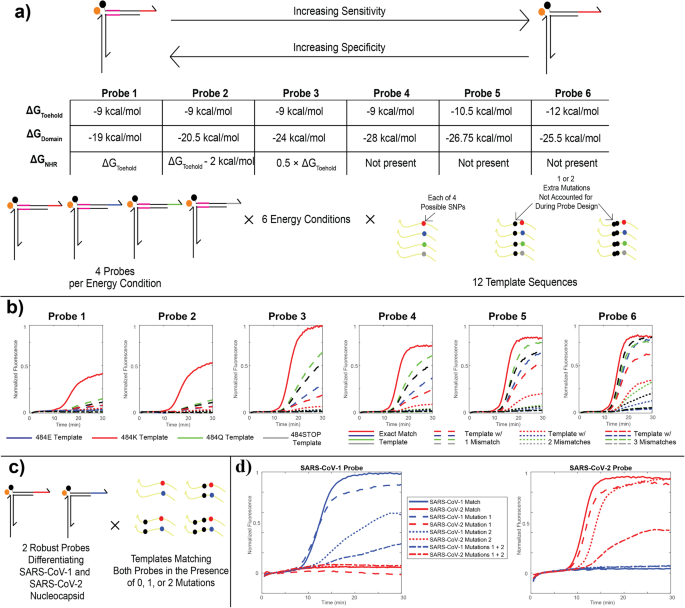
견고한 프로브와 SNV 식별 프로브 간의 에너지 미세 조정
RT-caPCR 분석에서 검출된 표적의 다양성을 높이기 위해 우리는 민감도-특이성 트레이드오프를 조절하기 위해 발가락 결합 및 가닥 변위의 에너지를 최적화했습니다. 사례 연구로서 우리는 에너지 평가를 위한 템플릿으로 SARS-CoV-2 '484E' 유전자형을 사용했습니다. 이 부위의 돌연변이는 숙주 항체의 중화 활성 감소와 관련이 있습니다.12, 그리고 3개의 가능한 뉴클레오티드 염기(아데닌, 구아닌 및 시토신) 중 4개가 자연적으로 진화된 SARS-CoV-2 변종의 코돈의 첫 번째 위치에서 발견됩니다.9. 이 부위의 우라실 대체는 정지 코돈을 생성하므로 자연에서는 관찰되지 않았지만 엄격함을 위해 우리는 평가에 티민 조건을 포함했습니다. 우리는 이 유전자좌에서 가능한 4개의 뉴클레오티드 각각에 대한 프로브를 설계하고 결합 에너지(ΔG)를 기반으로 6개의 서로 다른 열역학적 조건을 설정했습니다.o) 발끝 탐침의 3개 영역(NHR, 도메인, 발끝); 그림 XNUMX. 3a, 재료 및 방법 참조). 이러한 에너지 조건은 NHR과 발판의 에너지가 거의 동일한(약 -1 kcal/mol) 가장 구체적인(탐침 9)부터 NHR이 없고 가장 민감한(탐침 6)까지 다양합니다. 발판이 약간 더 강합니다(−12 kcal/mol). 모든 경우에 도메인 결합 에너지는 전체 프로브 결합 에너지가 약 -40 kcal/mol로 일정하게 유지되도록 조정됩니다.
a 프로브 에너지 특성화를 위한 설계 매개변수입니다. 돌연변이에 대한 견고성 형태로 단일 뉴클레오티드 특이성과 민감도 사이의 균형을 허용하기 위해 발판, 도메인 및 NHR 영역의 결합 에너지를 변화시켜 484가지 서로 다른 에너지 체계를 설계했습니다. SARS-CoV-2 스파이크 단백질의 12 돌연변이 부위에 있는 4개의 가능한 코돈을 모두 포괄하는 XNUMX개의 에너지 설계 각각에 대해 XNUMX개의 서로 다른 프로브가 설계되었으며, 그 중 XNUMX개는 변종에서 관찰되었습니다. 각 프로브는 XNUMX개의 고유한 단일 뉴클레오티드 돌연변이만 포함하고 Omicron 변이체에서 추가 상류에서 발견되는 XNUMX개 또는 XNUMX개의 다른 돌연변이가 있는 경우를 포함하여 XNUMX개의 서로 다른 플라스미드 주형으로 테스트되었습니다. b caPCR은 484K 시리즈 프로브의 결과입니다. 빨간색 실선은 돌연변이가 없는 표적 성능을 나타냅니다. 점선, 점선 및 점선은 각각 프로브 서열에 비해 1, 2 또는 3개의 불일치가 있는 템플릿을 나타냅니다. 색상은 484 부위(E, K, Q 또는 STOP)의 코돈을 지정합니다. c 강력한 SARS-CoV-1/2 차별 테스트를 위한 계획입니다. 에너지 계획에 따라 두 개의 프로브가 설계되었습니다. “탐사 5” 일부 a SARS-CoV-1 및 SARS-CoV-2의 뉴클레오캡시드 유전자 영역에 대한 것입니다. 두 프로브 모두 정확히 일치하는 템플릿과 XNUMX개의 업스트림 불일치 중 하나 또는 둘 다를 포함하는 변형이 있는 환경에서 테스트되었으며, 정확히 일치하는 템플릿과 다른 프로브의 불일치 변형이 있는 경우에도 테스트되었습니다. d (에 설명된 테스트 결과)c). 실선, 점선, 점선 및 점선은 정확히 일치하는 템플릿, SARS-CoV-1(파란색) 또는 SARS-CoV-2(빨간색) 게놈과 관련하여 두 SNV 중 하나가 포함된 템플릿 또는 두 SNV가 모두 포함된 템플릿을 나타냅니다.
우리는 12개의 서로 다른 DNA 템플릿을 사용하여 이러한 프로브를 테스트했으며, 그 중 4개는 4 코돈의 가능한 염기 484개 각각에 대해 정확히 일치하는 템플릿을 나타냅니다. 프로브 에너지의 조정 가능성을 입증하기 위해 우리는 SARS-CoV-2 진화의 후기 계통에서 발생한 돌연변이를 가져와 이를 템플릿에 도입했습니다. 즉, Delta AY 및 Omicron 계통에서 발견된 T478K 돌연변이와 Omicron에서 발견된 S477N 돌연변이입니다. 계통(도식적 그림. 3a)10. 24개 프로브 모두는 12개 주형 각각의 증폭에 반응하여 평가되었습니다. 수치 3b 특히 베타 및 감마 VOC에서 발견된 484 K 돌연변이를 표적으로 삼도록 설계된 프로브에 대한 이러한 결과의 하위 집합을 보여줍니다.10. NHR의 강도가 감소하고 발판이 증가함에 따라 정확히 일치하는 템플릿(빨간색 실선)에 대한 감지는 여전히 강력하며 프로브는 1(파선) 및 2(점선) 불일치가 있는 템플릿에 더 민감해집니다. 이는 매우 구체적인 표적 결합 결과를 위해 프로브 에너지를 설계하는 우리의 능력을 보여줍니다. 이러한 결과를 바탕으로 우리는 SNV 식별 프로브에 대해 에너지 체계 2를 선택하고 강력한 프로브에 대해 체계 5를 선택했습니다.
우리는 SARS-CoV-2 및 SARS-CoV-1의 뉴클레오캡시드 유전자 서열을 표적으로 하는 2개의 강력한 프로브를 설계했습니다. 돌연변이에 대한 내성을 유지하면서 강력한 프로브를 사용하여 이러한 병원체 서열을 차별화할 수 있도록 하기 위해 우리는 불일치 없음, 8개 중 1개 불일치 또는 두 가지 불일치가 모두 포함된 2개의 템플릿을 고안했습니다(그림 XNUMX). 3c, 재료 및 방법 참조). SARS-CoV-1 및 SARS-CoV-2 감지를 위해 선택된 영역은 2개의 표적 서열에서 8개의 돌연변이에 의해 다릅니다. 따라서 주형의 불일치는 이러한 차이를 기반으로 선택되었습니다. 따라서 2개의 불일치가 있는 SARS-CoV-2 템플릿은 SARS-CoV-6 서열과 비교하여 1개의 차이점만 갖고 있으므로 민감도 대 특이성에 대한 프로브의 정확한 조정 가능성의 중요성에 대한 실제 적용이 강조됩니다. 수치 3d 이러한 테스트에 대한 결과를 보여줍니다. 모든 경우에서 우리는 SARS-CoV-1과 SARS-CoV-2 프로브 및 템플릿 사이에 교차 반응이 없음을 관찰했습니다. SARS-CoV-1 RNA는 SARS-CoV-1 프로브에 의해서만 감지되었으며 SARS-CoV-2는 RNA는 SARS-CoV-2 프로브에 의해서만 검출되었습니다. 2개의 돌연변이 중 하나를 포함하는 템플릿은 각각의 적절한 프로브에 의해 명확하게 감지될 수 있으며, 1개의 돌연변이 중 하나가 다른 것보다 성능에 더 명확하게 영향을 미칩니다. 마찬가지로, 두 가지 돌연변이가 함께 있는 템플릿은 여전히 감지되지만 소광제 암 변위의 에너지 선호도가 낮아 성능이 약간 손상됩니다.
프로브 에너지학에 대한 목표 연구를 통해 우리는 민감도와 특이성의 원하는 균형을 유지하도록 프로브를 정밀하게 설계할 수 있는 능력을 입증했습니다. SNV 차별적 프로브는 소광제 팔의 변위와 정확히 일치해야 하는 에너지학을 갖고 있는 반면, 견고한 프로브는 최대 1개의 돌연변이를 포함하는 앰플리콘을 검출할 수 있는 능력으로 표적 결합에 있어 더 난잡합니다. 우리가 탐구한 에너지 범위는 에너지 체계 6에서 에너지 체계 XNUMX까지 필요에 따라 프로브를 조정하여 프로브 시퀀스에서 원하는 수의 불일치가 있는 표적을 감지할 수 있도록 적용할 수 있습니다.
강력하고 SNV 차별적 프로브의 성능
고도로 조정 가능한 프로브 설계에 대한 개념 증명을 입증하기 위해 강력하고 SNV 차별적인 프로브로 구성된 전체 패널을 만들었습니다. 우리 패널은 인플루엔자뿐 아니라 인간을 감염시키는 것으로 알려진 7가지 코로나바이러스(HCoV 229E, HKU1, NL63, OC43, MERS-CoV, SARS-CoV-1, SARS-CoV-2)를 모두 검사하여 강력한 프로브의 유용성을 강조합니다. A와 B8. 또한 우리 패널은 SARS-CoV-14 스파이크 유전자에서 일반적으로 돌연변이가 발생하는 3개 부위를 포괄하는 5개의 프로브와 2개의 앰플리콘을 사용하여 SNV 식별 프로브의 기능을 보여줍니다. 이를 통해 우려되는 변종(417, 452, 484, 501 및 614; 그림. S2)9,10,11.
민감한 표적 검출을 위한 RT-caPCR 분석에서 강력한 프로브의 성능을 평가하기 위해 우리는 다양한 환자 인구통계를 포괄하는 코로나바이러스 음성 바이러스 수송 매체(VTM) 샘플을 사용하여 65개의 RT-caPCR 테스트를 수행했습니다(표 S1-S4) Twist Bioscience에서 구입했거나 유전자 단편의 시험관 전사(IVT)를 사용하여 사내에서 합성한 7가지 인간 코로나바이러스 각각의 합성 RNA를 첨가했습니다(표 S5). 이러한 인위적인 임상 샘플로부터 비드 기반 추출 방법을 사용하여 RNA를 분리 및 정제했습니다.13 SARS-CoV-2 진단 작업흐름에서 RNA 추출을 위해 이러한 방법론을 사용하는 우선순위로 인해14 우리 시스템의 입력으로 사용되었습니다. 이 분석에는 또한 추출 대조군(정화 전에 인위적인 샘플에 첨가된 MS2 박테리오파지의 RNA)과 증폭 대조군(RT-caPCR 반응 혼합물에 첨가된 내부 양성 대조군(IPC) RNA)이 모두 포함되었습니다. 달리 명시하지 않는 한, 여기서 실험은 대략 10번의 결과를 받았습니다.6 MS2 RNA 사본 및 107 바이러스 RNA의 사본. RT-caPCR 반응은 40 분자의 IPC 대조 RNA, 추출된 샘플 RNA, 그리고 모든 표적과 대조를 포괄하는 5,000개의 프라이머 쌍을 사용하여 11분 만에 수행되었습니다.
각 샘플에 존재하는 바이러스 표적을 확인하기 위해 RT-caPCR 데이터를 분석했으며, SARS-CoV-2의 경우 어떤 특정 변종이 존재했는지 확인했습니다(그림 XNUMX). 4). 그림 4a SARS-CoV-2 Omicron 변종을 RNA 표적으로 사용한 대표적인 실행을 보여줍니다. 바이러스 식별을 위한 모든 강력한 프로브는 빨간색으로 표시되고 SNV 식별 프로브는 파란색으로 표시됩니다. 추출 및 증폭 컨트롤은 녹색으로 표시됩니다. 양성 및 음성 프로브 스팟은 프라이머나 템플릿이 포함되지 않고 회색으로 표시되는 시퀀스를 사용하여 완전히 켄칭되지 않거나 완전히 켄칭된 프로브에 해당합니다. 이러한 유형의 데이터는 그림 XNUMX과 같이 압축된 플롯으로 요약될 수 있습니다. 4b: 예상되는 모든 양의 곡선은 왼쪽 플롯에 표시되고 예상되는 모든 음의 곡선은 오른쪽 플롯에 표시됩니다. 일부 SNV 특이적 프로브에는 작은 수준의 표적 외 활성이 있는 반면, 표적 SNV 특이적 프로브 곡선(484 A, 452 L, 501Y-Om, 417 N 및 614 G)을 다른 SNV-특이적 프로브 곡선과 비교하면 특정 프로브는 눈뿐만 아니라 형광 유도체 기반 SNV 식별 알고리즘을 통해 평가할 때 표적 프로브가 모든 경우에서 가장 강력하게 반응한다는 것을 확인합니다 (재료 및 방법 및 보충 방법 참조). Omicron 변종은 다른 변종에 비해 돌연변이 부하가 높기 때문에11, 우리는 검출을 개선하기 위해 이 변종에서 발견된 S484N 및 T477K 돌연변이도 포함하도록 478 A 프로브의 서열을 설계했습니다.10. 마찬가지로 고유한 501Y-Om 프로브에는 Omicron 변종에서 특별히 발견되는 Q493R, G496S 및 Q498R 돌연변이가 포함되어 있습니다. Omicron 계통의 BA.2 하위 변종(501Y-Om2)에 특정한 프로브가 나중에 도입되었습니다. 이 프로브는 501Y-Om 서열과 동일하지만 G496S 돌연변이가 없습니다10.
a 우려되는 SARS-CoV-2 Omicron 변종을 검출하는 RT-caPCR 실행 결과의 예. 강력한 바이러스 검출 프로브는 빨간색, SNV 식별 프로브는 파란색, 추출 및 증폭 컨트롤은 녹색, 양성 및 음성 컨트롤은 회색으로 표시됩니다. 각 곡선은 60개의 획득된 이미지에 대한 각 시점에서 해당 프로브 어레이 지점의 강도를 나타내며 어레이에서 가장 밝은 지점으로 정규화됩니다. b 실행에 해당하는 요약 결과 a. 예상되는 모든 양성(추출 및 증폭 제어, 표적 바이러스 식별 및 모든 표적 SNV 식별 프로브)은 왼쪽 플롯에 표시되는 반면 예상되는 모든 음성 프로브(다른 모든 바이러스 및 SNV 식별 프로브)는 다음과 같습니다. 음성 대조군도 포함)이 오른쪽 플롯에 표시됩니다. c 인간 코로나바이러스 229E, 중동 호흡기 증후군 코로나바이러스(MERS-CoV) 및 SARS-CoV-1에 대한 바이러스 검출 결과는 (에 설명된 형식)b). d. SNV 식별은 위에 지정된 형식의 SARS-CoV-2 Delta 변종 템플릿에서 비롯됩니다.
그림 4c 인간 코로나바이러스 3E, MERS-CoV, SARS-CoV-3 등 229가지 서로 다른 바이러스 병원체 템플릿에 대한 1회 실행의 압축된 대표 결과를 보여줍니다. 3개의 샘플 모두 강력한 병원체 식별 프로브를 사용하여 바이러스 RNA 표적을 명확하게 검출하고 표적을 벗어난 바이러스 식별 프로브 활동이 없으며 모든 대조군의 존재를 보여줍니다.
작업대 1 모든 유효한 시험의 바이러스 식별 결과를 요약합니다. PCR 반응 자원(예: 중합효소, dNTP)에 대한 경쟁은 낮은 복제 수 표적의 증폭을 억제할 수 있으므로 바이러스 병원체가 검출되고 1개의 대조군(MS2 및 IPC) 중 최소 2개가 검출된 경우 분석 실행이 유효한 것으로 간주되었습니다. 또는 바이러스 병원체가 검출되지 않았지만 두 대조군 모두 검출되었습니다. 샘플 중 11개는 MS2 및 IPC 대조 RNA만 포함하는 음성 대조였습니다. 이들 샘플 11개 모두에서 IPC와 MS2가 모두 검출되었습니다(100%). SARS-CoV-36 뉴클레오캡시드 프로브 검출을 통해 평가한 결과 SARS-CoV-36 샘플 2개 중 100개가 식별되었습니다(2%). 나머지 인간 코로나바이러스(229E, NL63, OC43 및 HKU1)와 SARS-CoV-1 및 MERS-CoV 표적도 모든 표적에 대해 100% 탐지되는 높은 민감도를 나타냅니다. 프로브의 프라이머 결합 과정과 앰플리콘 매개 소광제 변위 모두 패널의 특이성에 기여하며 이는 모든 표적에 걸쳐 100%였습니다. 여기서 특이성은 정확하게 검출된 참음성의 비율로 계산됩니다. 간단히 말해서, 특별히 설계된 프로브 이외의 견고한 프로브에서는 템플릿 유도 감지가 없습니다.
그림 4d SARS-CoV-2 Delta 변이체에 대한 병원체 식별과 동시에 바이러스 변이체 검출을 보여줌으로써 RNA 표적을 식별할 뿐만 아니라 단일 뉴클레오티드 분해능을 제공하는 RT-caPCR 분석의 능력을 강조하는 대표적인 실행을 보여줍니다. 452 L 프로브는 상당한 비표적 활동을 보여주지만, 그 검출은 표적 452 R 프로브(왼쪽 플롯에서 강도가 가장 높은 파란색 곡선)보다 확실히 덜 효율적입니다. 따라서 SNV 식별 프로브를 사용하면 몇 가지 단일 돌연변이만 다른 SARS-CoV-2 변종을 명확하게 구분할 수 있습니다.
작업대 2 2개 임상시험에서 SARS-CoV-36 변종에 대한 SNV 특이적 프로브의 성능을 요약합니다. 예상되는 검출 수는 해당 돌연변이를 표시하는 템플릿이 포함된 실행 수와 동일하고, 예상되는 음성 수는 해당 템플릿이 포함되지 않은 실행 수입니다. 진양성은 관심 있는 프로브가 해당 돌연변이 부위에 대해 지배적인 SNV 특이적 프로브였던 실행이고(알고리즘으로 평가됨), 진음성은 그렇지 않은 실행입니다. 한 번의 실행에서 614 G 프로브가 이물질(먼지)에 의해 가려졌으므로 이러한 목표의 총계는 35이 아닌 36입니다. 484E를 제외하고 모든 프로브는 최소 90% 감도를 나타냅니다. 484E 프로브의 낮은 성능은 Delta 변이체의 앰플리콘에 SNV(T478K 돌연변이)가 존재하기 때문에 부분적으로 설명될 수 있습니다. 다른 모든 프로브는 100% 특이성을 보여줍니다. 표적 에너지가 동일하다고 가정할 때 앰플리콘은 항상 돌연변이가 있는 프로브보다 일치하는 소광제를 우선적으로 대체하기 때문입니다.
함께, 이러한 데이터는 강력한 프로브와 SNV 식별 프로브가 함께 작동하여 존재하는 RNA 표적(강력한 프로브 사용)을 신속하게 식별하고 단일 뉴클레오티드 분해능(SNV 식별 프로브 사용)을 제공하는 능력을 강조합니다.
RT-caPCR의 검출 한계 평가
다음으로 우리는 RT-caPCR 분석의 검출 한계를 결정하기 위해 일련의 실험을 수행했습니다. 이전의 모든 테스트에서는 Fig. 4, 바이러스 입력 107 분자는 추출 전에 VTM에 주입되었으며, 이는 Omicron 및 Delta 환자의 최대 바이러스 부하 추정치와 유사합니다.15,16. 여기서 우리는 바이러스 RNA의 입력 사본 수를 변경하고 대조 사본을 동일하게 유지하여 분석의 LOD를 정량화했습니다. Twist Bioscience에서 얻거나 IVT를 통해 유전자 단편에서 사내에서 합성한 정제된 RNA를 사용하여 우리는 1,000개 코로나바이러스 모두에 대해 7분자 LOD를 달성했습니다. 대표적인 결과는 그림 XNUMX에 나와 있습니다. S3. 두 그림 모두에서. S3a 과 S3b, 우리는 두 컨트롤(HCoV 1,000E; 그림 229)을 사용하여 단일 표적에 대해 반응당 최대 XNUMX개의 분자를 성공적으로 감지할 수 있음을 발견했습니다. S3a) 및 두 컨트롤이 모두 포함된 SARS-CoV-2 변종(Omicron, 그림 XNUMX)의 경우. S3b), 정제된 RNA를 사용할 때 VTM 테스트에 사용되는 것보다 3배 낮은 양입니다. 그림과 같이 단일 타겟의 존재 여부만 변경되는 경우입니다. S3a, 우리는 이 목표에 대해서만 각 규모에 대한 임계 시간의 명확한 지연을 발견한 반면, 2개의 컨트롤은 실행 전반에 걸쳐 일관됩니다. SARS-CoV-2의 경우 이러한 추세는 덜 명확하며, 이 목표에는 4개의 앰플리콘과 2개의 컨트롤의 동시 증폭이 필요하다는 점을 고려할 때 예상됩니다. 효소 자원과 dNTP에 대한 경쟁은 단일 앰플리콘 표적에서 관찰된 명확한 추세를 방해하는 것으로 보입니다. 그림에서. S3C, 희석 시리즈는 Omicron 변이체에 대한 추출 파이프라인에 대한 입력 RNA의 양을 변경하여 수행되었습니다. 여기에서도 가장 낮은 테스트 값까지 검출이 가능합니다(RNA 추출 단계에 10,000개 분자 입력, 추출 중 손실로 인해 대략 1,000개 분자가 RT 단계로 들어가고 반응에서 전체 25μL 용출량을 사용하지 않음). 입력 농도의 함수로서 예상되는 증폭 지연.
단일 샘플에서 여러 타겟 감지
SNV 식별 프로브(SARS-CoV-2 변종)를 사용하여 단일 뉴클레오티드 특이성을 갖춘 강력한 프로브(각 바이러스)로 RNA 표적을 식별하는 조정 가능한 프로브 RT-caPCR 분석의 성공을 입증한 후, 다음으로 우리의 분석이 다음과 같은 경우에 어떻게 수행되는지 탐구했습니다. 여러 RNA 표적이 포함된 동시 감염 샘플을 시뮬레이션하여 보다 복잡한 입력을 평가합니다. 여기서 입력량은 10입니다.5 각 템플릿의 분자는 VTM 추출 단계 없이 RT-caPCR 반응에 직접 첨가되었습니다. 또한 인플루엔자 A 및 B를 테스트하기 위한 3개의 추가 프라이머 세트와 4개의 프로브(2개는 인플루엔자 A의 M1 매트릭스 단백질 유전자를 표적으로 함)를 포함하여 더 많은 RNA 표적을 테스트하기 위해 RT-caPCR 분석을 확장했습니다.17 2개는 인플루엔자 B의 야마가타 및 빅토리아 계통의 비구조 단백질-1(nsp1)을 표적으로 합니다.18). 이 14-플렉스 분석은 그 효능을 확인하기 위해 4개의 새로운 표적 모두에 대해 테스트되었습니다(그림 XNUMX). S4).
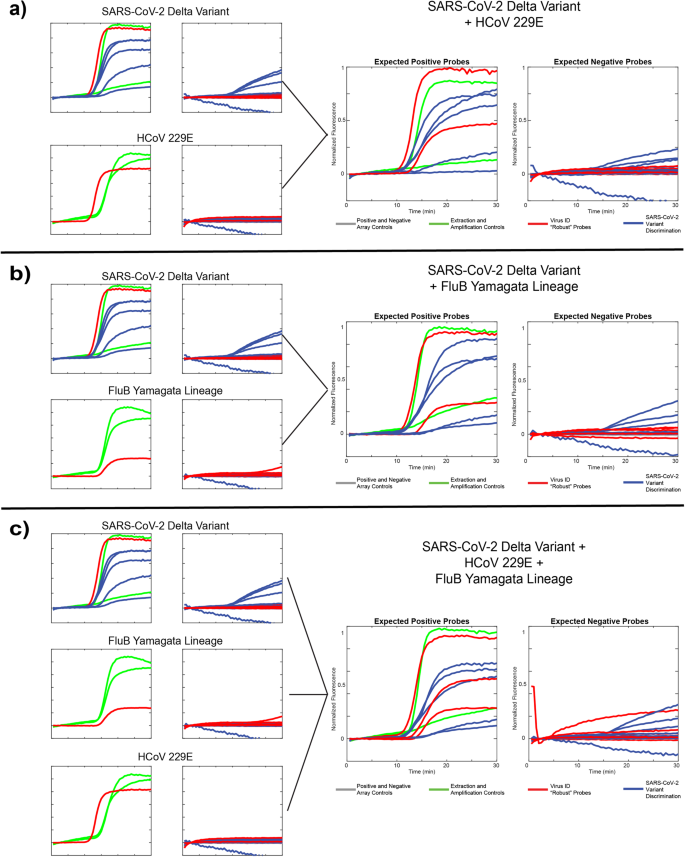
우리는 3가지 서로 다른 바이러스 템플릿 각각을 단독으로 평가했을 뿐만 아니라 SARS-CoV-3 Delta 변형 + HCoV2E의 229가지 서로 다른 조합으로 평가했습니다(그림 XNUMX). 5a), SARS-CoV-2 Delta 변종 + FluB Yamagata 계통(그림. 5b) 및 SARS-CoV-2 + HCoV229E + FluB Yamagata 계통(그림 XNUMX) 5c). 데이터는 위에서 설명한 대로 압축된 형광 추적 이미지로 표시되며 왼쪽 패널에는 예상되는 양성 프로브가 있고 오른쪽 패널에는 예상되는 음성 프로브가 있습니다. 모든 실행에서 우리는 예상되는 동작을 관찰했습니다. 강력한(바이러스 식별) 프로브는 포함된 모든 병원체 템플릿의 존재를 보고하고 SNV 식별 프로브(변종 식별)는 전체에서 낮은 수준의 표적을 벗어난 프로브 활동으로 Delta 변종을 정확하게 식별합니다. 5개의 SNV 사이트. 484E 프로브는 앞서 언급한 프로브 도메인 영역의 불일치로 인해 성능이 가장 약합니다. 또한 일부 실험에서는 2개의 서로 다른 앰플리콘 간의 리소스 경쟁으로 인해 MS8 추출 제어가 제대로 수행되지 않았습니다. 또한 이러한 실행에는 그림에서와 같이 1개가 아닌 MS2 및 IPC 컨트롤 각각의 2개 복제만 포함된다는 점에 유의하세요. 4, 이는 501Y-Om2 프로브를 포함하는 업데이트된 패널이기 때문입니다. 이러한 데이터는 관심 있는 여러 RNA 표적을 포함하는 보다 복잡한 샘플을 정확하게 평가할 수 있는 당사 기술의 능력을 입증하며 분석의 다양성을 보여줍니다.
- SEO 기반 콘텐츠 및 PR 배포. 오늘 증폭하십시오.
- PlatoData.Network 수직 생성 Ai. 자신에게 권한을 부여하십시오. 여기에서 액세스하십시오.
- PlatoAiStream. 웹3 인텔리전스. 지식 증폭. 여기에서 액세스하십시오.
- 플라톤ESG. 탄소, 클린테크, 에너지, 환경, 태양광, 폐기물 관리. 여기에서 액세스하십시오.
- PlatoHealth. 생명 공학 및 임상 시험 인텔리전스. 여기에서 액세스하십시오.
- 출처: https://www.nature.com/articles/s42003-023-05346-4