In questo post, dimostriamo come mettere a punto in modo efficiente un modello all'avanguardia del linguaggio proteico (pLM) per prevedere la localizzazione subcellulare delle proteine utilizzando Amazon Sage Maker.
Le proteine sono le macchine molecolari del corpo, responsabili di tutto, dal movimento dei muscoli alla risposta alle infezioni. Nonostante questa varietà, tutte le proteine sono costituite da catene ripetitive di molecole chiamate amminoacidi. Il genoma umano codifica 20 aminoacidi standard, ciascuno con una struttura chimica leggermente diversa. Queste possono essere rappresentate da lettere dell'alfabeto, il che ci permette quindi di analizzare ed esplorare le proteine come una stringa di testo. L’enorme numero possibile di sequenze e strutture proteiche è ciò che conferisce alle proteine la loro ampia varietà di usi.
Le proteine svolgono anche un ruolo chiave nello sviluppo di farmaci, come potenziali bersagli ma anche come agenti terapeutici. Come mostrato nella tabella seguente, molti dei farmaci più venduti nel 2022 erano proteine (soprattutto anticorpi) o altre molecole come l’mRNA tradotte in proteine nel corpo. Per questo motivo, molti ricercatori nel campo delle scienze della vita devono rispondere a domande sulle proteine in modo più rapido, economico e accurato.
| Nome | Costruttore | Vendite globali 2022 (miliardi di dollari) | indicazioni |
| comirnaty | Pfizer / Biontech | $40.8 | COVID-19 |
| spikevax | moderno | $21.8 | COVID-19 |
| Humira | Abbvie | $21.6 | Artrite, morbo di Crohn e altri |
| Chiavetruda | Merck | $21.0 | Vari tumori |
Fonte dei dati: Urquhart, L. Principali aziende e farmaci per vendite nel 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Poiché possiamo rappresentare le proteine come sequenze di caratteri, possiamo analizzarle utilizzando tecniche originariamente sviluppate per il linguaggio scritto. Ciò include modelli linguistici di grandi dimensioni (LLM) preaddestrati su enormi set di dati, che possono poi essere adattati per attività specifiche, come il riepilogo del testo o i chatbot. Allo stesso modo, i pLM vengono pre-addestrati su grandi database di sequenze proteiche utilizzando l'apprendimento senza etichetta e autosuperato. Possiamo adattarli per prevedere cose come la struttura 3D di una proteina o come potrebbe interagire con altre molecole. I ricercatori hanno persino utilizzato i pLM per progettare nuove proteine da zero. Questi strumenti non sostituiscono le competenze scientifiche umane, ma hanno il potenziale per accelerare lo sviluppo preclinico e la progettazione degli studi clinici.
Una sfida con questi modelli è la loro dimensione. Sia gli LLM che i pLM sono cresciuti di diversi ordini di grandezza negli ultimi anni, come illustrato nella figura seguente. Ciò significa che può essere necessario molto tempo per addestrarli a una precisione sufficiente. Significa anche che è necessario utilizzare hardware, in particolare GPU, con grandi quantità di memoria per archiviare i parametri del modello.
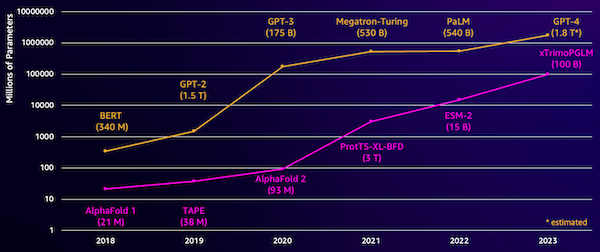
Lunghi tempi di formazione, oltre a istanze di grandi dimensioni, equivalgono a costi elevati, che possono rendere questo lavoro fuori dalla portata di molti ricercatori. Ad esempio, nel 2023, a gruppo di ricerca ha descritto l'addestramento di un pLM da 100 miliardi di parametri su 768 GPU A100 per 164 giorni! Fortunatamente, in molti casi possiamo risparmiare tempo e risorse adattando un pLM esistente al nostro compito specifico. Questa tecnica si chiama ritocchi, e ci consente anche di prendere in prestito strumenti avanzati da altri tipi di modellazione del linguaggio.
Panoramica della soluzione
Il problema specifico che affrontiamo in questo post è localizzazione subcellulare: Data una sequenza proteica, possiamo costruire un modello in grado di prevedere se vive all'esterno (membrana cellulare) o all'interno di una cellula? Questa è un'informazione importante che può aiutarci a comprenderne la funzione e se potrebbe costituire un buon bersaglio farmacologico.
Iniziamo scaricando un set di dati pubblico utilizzando Amazon Sage Maker Studio. Quindi utilizziamo SageMaker per mettere a punto il modello del linguaggio proteico ESM-2 utilizzando un metodo di formazione efficiente. Infine, implementiamo il modello come endpoint di inferenza in tempo reale e lo utilizziamo per testare alcune proteine conosciute. Il diagramma seguente illustra questo flusso di lavoro.

Nelle sezioni seguenti, esaminiamo i passaggi per preparare i dati di formazione, creare uno script di formazione ed eseguire un processo di formazione SageMaker. Tutto il codice presente in questo post è disponibile su GitHub.
Prepara i dati di allenamento
Usiamo parte del Set di dati DeepLoc-2, che contiene diverse migliaia di proteine SwissProt con posizioni determinate sperimentalmente. Filtriamo per sequenze di alta qualità tra 100 e 512 aminoacidi:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Successivamente, tokenizziamo le sequenze e le dividiamo in set di training e valutazione:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Infine, carichiamo i dati di formazione e valutazione elaborati su Servizio di archiviazione semplice Amazon (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Crea uno script di formazione
Modalità script SageMaker ti consente di eseguire il tuo codice di formazione personalizzato in contenitori framework di machine learning (ML) ottimizzati gestiti da AWS. Per questo esempio, adattiamo an script esistente per la classificazione del testo da Il viso che abbraccia. Questo ci permette di provare diversi metodi per migliorare l’efficienza del nostro lavoro di formazione.
Metodo 1: lezione di allenamento ponderata
Come molti set di dati biologici, i dati DeepLoc sono distribuiti in modo non uniforme, il che significa che non esiste un numero uguale di proteine di membrana e non di membrana. Potremmo ricampionare i nostri dati ed eliminare i record della classe maggioritaria. Tuttavia, ciò ridurrebbe i dati di allenamento totali e potrebbe compromettere la nostra precisione. Invece, calcoliamo i pesi delle classi durante il lavoro di formazione e li utilizziamo per correggere la perdita.
Nel nostro script di training sottoclassiamo the Trainer classe da transformers con una WeightedTrainer classe che tiene conto dei pesi delle classi nel calcolo della perdita di entropia incrociata. Ciò aiuta a prevenire distorsioni nel nostro modello:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMetodo 2: accumulo del gradiente
L'accumulo del gradiente è una tecnica di training che consente ai modelli di simulare l'addestramento su batch di dimensioni maggiori. In genere, la dimensione del batch (il numero di campioni utilizzati per calcolare il gradiente in una fase di training) è limitata dalla capacità di memoria della GPU. Con l'accumulo del gradiente, il modello calcola prima i gradienti su lotti più piccoli. Quindi, invece di aggiornare immediatamente i pesi del modello, i gradienti vengono accumulati in più piccoli lotti. Quando i gradienti accumulati equivalgono alla dimensione batch maggiore target, viene eseguita la fase di ottimizzazione per aggiornare il modello. Ciò consente ai modelli di addestrarsi con batch effettivamente più grandi senza superare il limite di memoria della GPU.
Tuttavia, è necessario un calcolo aggiuntivo per i passaggi avanti e indietro dei lotti più piccoli. L'aumento delle dimensioni dei batch tramite l'accumulo del gradiente può rallentare l'addestramento, soprattutto se vengono utilizzate troppe fasi di accumulo. L'obiettivo è massimizzare l'utilizzo della GPU ma evitare rallentamenti eccessivi dovuti a troppi passaggi aggiuntivi di calcolo del gradiente.
Metodo 3: checkpoint del gradiente
Il gradient checkpoint è una tecnica che riduce la memoria necessaria durante l'addestramento mantenendo un tempo di calcolo ragionevole. Le grandi reti neurali occupano molta memoria perché devono memorizzare tutti i valori intermedi del passaggio in avanti per poter calcolare i gradienti durante il passaggio all'indietro. Ciò può causare problemi di memoria. Una soluzione è non memorizzare questi valori intermedi, ma poi devono essere ricalcolati durante il passaggio all'indietro, che richiede molto tempo.
Il checkpoint del gradiente fornisce un approccio equilibrato. Salva solo alcuni dei valori intermedi, chiamati posti di bloccoe ricalcola gli altri secondo necessità. Pertanto utilizza meno memoria rispetto all'archiviazione di tutto, ma anche meno calcoli rispetto al ricalcolo di tutto. Selezionando strategicamente quali attivazioni sottoporre a checkpoint, il gradient checkpoint consente di addestrare reti neurali di grandi dimensioni con un utilizzo della memoria e un tempo di calcolo gestibili. Questa importante tecnica rende possibile l'addestramento di modelli molto grandi che altrimenti incorrerebbero in limitazioni di memoria.
Nel nostro script di training, attiviamo l'attivazione del gradiente e il checkpoint aggiungendo i parametri necessari al file TrainingArguments oggetto:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Metodo 4: adattamento di basso rango dei LLM
Modelli linguistici di grandi dimensioni come ESM-2 possono contenere miliardi di parametri costosi da addestrare ed eseguire. Ricercatori ha sviluppato un metodo di formazione chiamato Low-Rank Adaptation (LoRA) per rendere più efficiente la messa a punto di questi enormi modelli.
L'idea chiave alla base di LoRA è che quando si mette a punto un modello per un'attività specifica, non è necessario aggiornare tutti i parametri originali. LoRA aggiunge invece nuove matrici più piccole al modello che trasformano gli input e gli output. Solo queste matrici più piccole vengono aggiornate durante la messa a punto, che è molto più veloce e utilizza meno memoria. I parametri del modello originale rimangono congelati.
Dopo la messa a punto con LoRA, è possibile unire nuovamente le piccole matrici adattate nel modello originale. Oppure puoi tenerli separati se desideri mettere a punto rapidamente il modello per altre attività senza dimenticare quelle precedenti. Nel complesso, LoRA consente di adattare in modo efficiente i LLM a nuovi compiti a una frazione del costo normale.
Nel nostro script di formazione, configuriamo LoRA utilizzando il file PEFT libreria da Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Invia un lavoro di formazione SageMaker
Dopo aver definito lo script di formazione, è possibile configurare e inviare un lavoro di formazione SageMaker. Innanzitutto, specifica gli iperparametri:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Successivamente, definisci quali metriche acquisire dai registri di addestramento:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Infine, definisci uno stimatore Hugging Face e invialo per l'addestramento su un tipo di istanza ml.g5.2xlarge. Si tratta di un tipo di istanza conveniente e ampiamente disponibile in molte regioni AWS:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)La tabella seguente mette a confronto i diversi metodi di formazione di cui abbiamo discusso e il loro effetto sui requisiti di runtime, precisione e memoria GPU del nostro lavoro.
| Configurazione | Tempo fatturabile (min) | Precisione della valutazione | Utilizzo massimo della memoria della GPU (GB) |
| Modello Base | 28 | 0.91 | 22.6 |
| Base+GA | 21 | 0.90 | 17.8 |
| Base+GC | 29 | 0.91 | 10.2 |
| Base + LoRA | 23 | 0.90 | 18.6 |
Tutti i metodi hanno prodotto modelli con un'elevata precisione di valutazione. L'utilizzo di LoRA e dell'attivazione del gradiente ha ridotto il tempo di esecuzione (e i costi) rispettivamente del 18% e del 25%. L'utilizzo del checkpoint gradiente ha ridotto l'utilizzo massimo della memoria della GPU del 55%. A seconda dei vincoli (costi, tempo, hardware), uno di questi approcci potrebbe avere più senso di un altro.
Ciascuno di questi metodi funziona bene da solo, ma cosa succede quando li usiamo in combinazione? La tabella seguente riassume i risultati.
| Configurazione | Tempo fatturabile (min) | Precisione della valutazione | Utilizzo massimo della memoria della GPU (GB) |
| Tutti i metodi | 12 | 0.80 | 3.3 |
In questo caso, vediamo una riduzione della precisione del 12%. Tuttavia, abbiamo ridotto l'autonomia del 57% e l'utilizzo della memoria della GPU dell'85%! Si tratta di una riduzione massiccia che ci consente di addestrarci su un'ampia gamma di tipi di istanze convenienti.
ripulire
Se segui il tuo account AWS, elimina eventuali endpoint e dati di inferenza in tempo reale che hai creato per evitare ulteriori addebiti.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Conclusione
In questo post, abbiamo dimostrato come mettere a punto in modo efficiente modelli di linguaggio proteico come ESM-2 per un compito scientificamente rilevante. Per ulteriori informazioni sull'utilizzo delle librerie Transformers e PEFT per addestrare pLMS, consulta i post Apprendimento profondo con le proteine ed ESMBind (ESMB): adattamento di basso rango di ESM-2 per la previsione del sito di legame delle proteine sul blog Hugging Face. Puoi anche trovare altri esempi di utilizzo dell'apprendimento automatico per prevedere le proprietà delle proteine nel file Impressionante analisi delle proteine su AWS Archivio GitHub.
L'autore
 Brian leale è Senior AI/ML Solutions Architect nel team Global Healthcare and Life Sciences di Amazon Web Services. Ha più di 17 anni di esperienza nella biotecnologia e nell'apprendimento automatico ed è appassionato di aiutare i clienti a risolvere le sfide genomiche e proteomiche. Nel tempo libero ama cucinare e mangiare con i suoi amici e la sua famiglia.
Brian leale è Senior AI/ML Solutions Architect nel team Global Healthcare and Life Sciences di Amazon Web Services. Ha più di 17 anni di esperienza nella biotecnologia e nell'apprendimento automatico ed è appassionato di aiutare i clienti a risolvere le sfide genomiche e proteomiche. Nel tempo libero ama cucinare e mangiare con i suoi amici e la sua famiglia.
- Distribuzione di contenuti basati su SEO e PR. Ricevi amplificazione oggi.
- PlatoData.Network Generativo verticale Ai. Potenzia te stesso. Accedi qui.
- PlatoAiStream. Intelligenza Web3. Conoscenza amplificata. Accedi qui.
- PlatoneESG. Carbonio, Tecnologia pulita, Energia, Ambiente, Solare, Gestione dei rifiuti. Accedi qui.
- Platone Salute. Intelligence sulle biotecnologie e sulle sperimentazioni cliniche. Accedi qui.
- Fonte: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



