Detta är ett gästinlägg medförfattare av Ajay K Gupta, Jean Felipe Teotonio och Paul A Churchyard från HSR.health.
HSR.hälsa är en geospatial hälsoriskanalysfirma vars vision är att globala hälsoutmaningar är lösbara genom mänsklig uppfinningsrikedom och fokuserad och korrekt tillämpning av dataanalys. I det här inlägget presenterar vi ett tillvägagångssätt för förebyggande av zoonotiska sjukdomar som använder Amazon SageMaker geospatiala funktioner att skapa ett verktyg som ger mer exakt information om sjukdomsspridning till hälsoforskare för att hjälpa dem att rädda fler liv, snabbare.
Zoonotiska sjukdomar drabbar både djur och människor. Övergången av en sjukdom från djur till människa, känd som spillovers, är ett fenomen som ständigt förekommer på vår planet. Enligt hälsoorganisationer som Centers for Disease Control and Prevention (CDC) och Världshälsoorganisationen (VEM), en spridningshändelse på en våt marknad i Wuhan, Kina, orsakade sannolikt coronavirussjukdomen 2019 (COVID-19). Studier tyder på att ett virus som finns i fruktfladdermöss genomgick betydande mutationer, vilket gjorde att det kunde infektera människor. Den initiala patienten, eller "patient noll", för COVID-19 startade förmodligen ett efterföljande lokalt utbrott som så småningom spred sig internationellt. HSR.hälsas Zoonotic Spillover Risk Index syftar till att hjälpa till att identifiera dessa tidiga utbrott innan de korsar internationella gränser och leder till omfattande global påverkan.
Det främsta vapnet som folkhälsan har mot spridningen av regionala utbrott är sjukdomsövervakning: ett helt sammankopplat system av sjukdomsrapportering, utredning och datakommunikation mellan olika nivåer i ett folkhälsosystem. Detta system är inte bara beroende av mänskliga faktorer, utan också på teknik och resurser för att samla in sjukdomsdata, analysera mönster och skapa en konsekvent och kontinuerlig ström av dataöverföring från lokala till regionala till centrala hälsomyndigheter.
Hastigheten med vilken covid-19 gick från ett lokalt utbrott till en global sjukdom som finns på varje enskild kontinent borde vara ett nykter exempel på det trängande behovet av att utnyttja innovativ teknik för att skapa effektivare och mer exakta sjukdomsövervakningssystem.
Risken för spridning av zoonotiska sjukdomar är skarpt korrelerad med flera sociala, miljömässiga och geografiska faktorer som påverkar hur ofta människor interagerar med vilda djur. HSR.hälsan Zoonotic Disease Spillover Risk Index använder över 20 distinkta geografiska, sociala och miljömässiga faktorer som historiskt är kända för att påverka risken för interaktion mellan människor och vilda djur och därmed risken för zoonotiska sjukdomar. Många av dessa faktorer kan kartläggas genom en kombination av satellitbilder och fjärranalys.
I det här inlägget utforskar vi hur HSR.hälsa använder SageMakers geospatiala funktioner för att hämta relevanta funktioner från satellitbilder och fjärranalys för att utveckla riskindexet. SageMakers geospatiala funktioner gör det enkelt för datavetare och maskininlärningsingenjörer (ML) att bygga, träna och distribuera modeller med hjälp av geospatial data. Med SageMakers geospatiala funktioner kan du effektivt transformera eller berika storskaliga geospatiala datauppsättningar, accelerera modellbyggandet med förutbildade ML-modeller och utforska modellförutsägelser och geospatial data på en interaktiv karta med hjälp av 3D-accelererad grafik och inbyggda visualiseringsverktyg.
Använda ML och geospatial data för riskreducering
ML är mycket effektivt för avvikelsedetektering på rumslig eller tidsmässig data på grund av dess förmåga att lära av data utan att vara explicit programmerad att identifiera specifika typer av anomalier. Rumslig data, som relaterar till objekts fysiska position och form, innehåller ofta komplexa mönster och samband som kan vara svåra att analysera för traditionella algoritmer.
Att integrera ML med geospatiala data förbättrar förmågan att systematiskt upptäcka anomalier och ovanliga mönster, vilket är viktigt för system för tidig varning. Dessa system är avgörande inom områden som miljöövervakning, katastrofhantering och säkerhet. Prediktiv modellering med hjälp av historisk geospatial data gör det möjligt för organisationer att identifiera och förbereda sig för potentiella framtida händelser. Dessa händelser sträcker sig från naturkatastrofer och trafikstörningar till, som det här inlägget diskuterar, sjukdomsutbrott.
Upptäcka zoonotiska spridningsrisker
För att förutsäga zoonotiska spridningsrisker, HSR.hälsa har antagit en multimodal strategi. Genom att använda en blandning av datatyper – inklusive miljö-, biogeografisk och epidemiologisk information – möjliggör denna metod en omfattande bedömning av sjukdomsdynamiken. Ett sådant mångfacetterat perspektiv är avgörande för att utveckla proaktiva åtgärder och möjliggöra ett snabbt svar på utbrott.
Tillvägagångssättet inkluderar följande komponenter:
- Data om sjukdomar och utbrott – HSR.hälsa använder de omfattande sjukdoms- och utbrottsdata som tillhandahålls av Gideon och Världshälsoorganisationen (WHO), två pålitliga källor till global epidemiologisk information. Dessa data fungerar som en grundläggande pelare i analysramverket. För Gideon kan data nås via ett API, och för WHO, HSR.hälsa har byggt en stor språkmodell (LLM) för att bryta utbrottsdata från tidigare rapporter om sjukdomsutbrott.
- Jordobservationsdata – Miljöfaktorer, markanvändningsanalys och upptäckt av habitatförändringar är viktiga komponenter för att bedöma zoonotisk risk. Dessa insikter kan härledas från satellitbaserade jordobservationsdata. HSR.hälsa kan effektivisera användningen av jordobservationsdata genom att använda SageMakers geospatiala funktioner för att komma åt och manipulera storskaliga geospatiala datauppsättningar. SageMaker geospatial erbjuder en rik datakatalog, inklusive datauppsättningar från USGS Landsat-8, Sentinel-1, Sentinel-2 och andra. Det är också möjligt att ta in andra datauppsättningar, till exempel högupplösta bilder från Planet Labs.
- Sociala bestämningsfaktorer för risk – Utöver biologiska och miljömässiga faktorer, teamet på HSR.hälsa betraktas också som sociala bestämningsfaktorer, som omfattar olika socioekonomiska och demografiska indikatorer och spelar en avgörande roll för att forma zoonotisk spridningsdynamik.
Från dessa komponenter, HSR.hälsa utvärderade en rad olika faktorer, och följande egenskaper har identifierats som inflytelserika för att identifiera zoonotiska spridningsrisker:
- Djurens livsmiljöer och beboeliga zoner – Att förstå livsmiljöerna för potentiella zoonotiska värdar och deras beboeliga zoner är grundläggande för att bedöma överföringsrisken.
- Befolkningscentra – Närhet till tätbefolkade områden är en viktig faktor eftersom det påverkar sannolikheten för interaktioner mellan människor och djur.
- Förlust av livsmiljö – Försämringen av naturliga livsmiljöer, särskilt genom avskogning, kan påskynda zoonotiska spillover-händelser.
- Gränssnitt mellan människa och vildmark – Områden där mänskliga bosättningar skär sig med vilda livsmiljöer är potentiella hotspots för zoonotisk överföring.
- Sociala egenskaper – Socioekonomiska och kulturella faktorer kan avsevärt påverka zoonotisk risk och HSR.hälsa undersöker även dessa.
- Mänskliga hälsoegenskaper – Hälsostatusen för lokalbefolkningen är en viktig variabel eftersom den påverkar känsligheten och överföringsdynamiken.
Lösningsöversikt
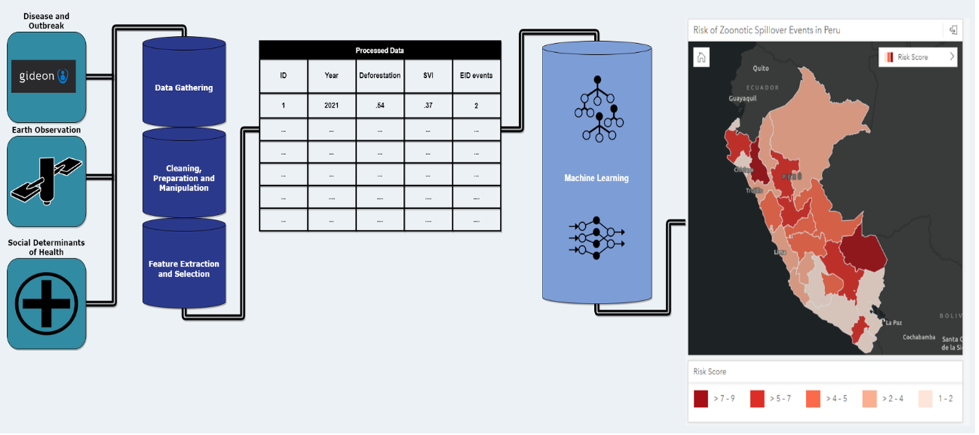
HSR.hälsaArbetsflödet omfattar dataförbearbetning, funktionsextraktion och skapandet av informativa visualiseringar med hjälp av ML-tekniker. Detta möjliggör en tydlig förståelse av datas utveckling från dess råa form till handlingsbara insikter.
Följande är en visuell representation av arbetsflödet, som börjar med indata från Gideon, jordobservationsdata och sociala bestämningsfaktorer för riskdata.
Hämta och bearbeta satellitbilder med SageMakers geospatiala funktioner
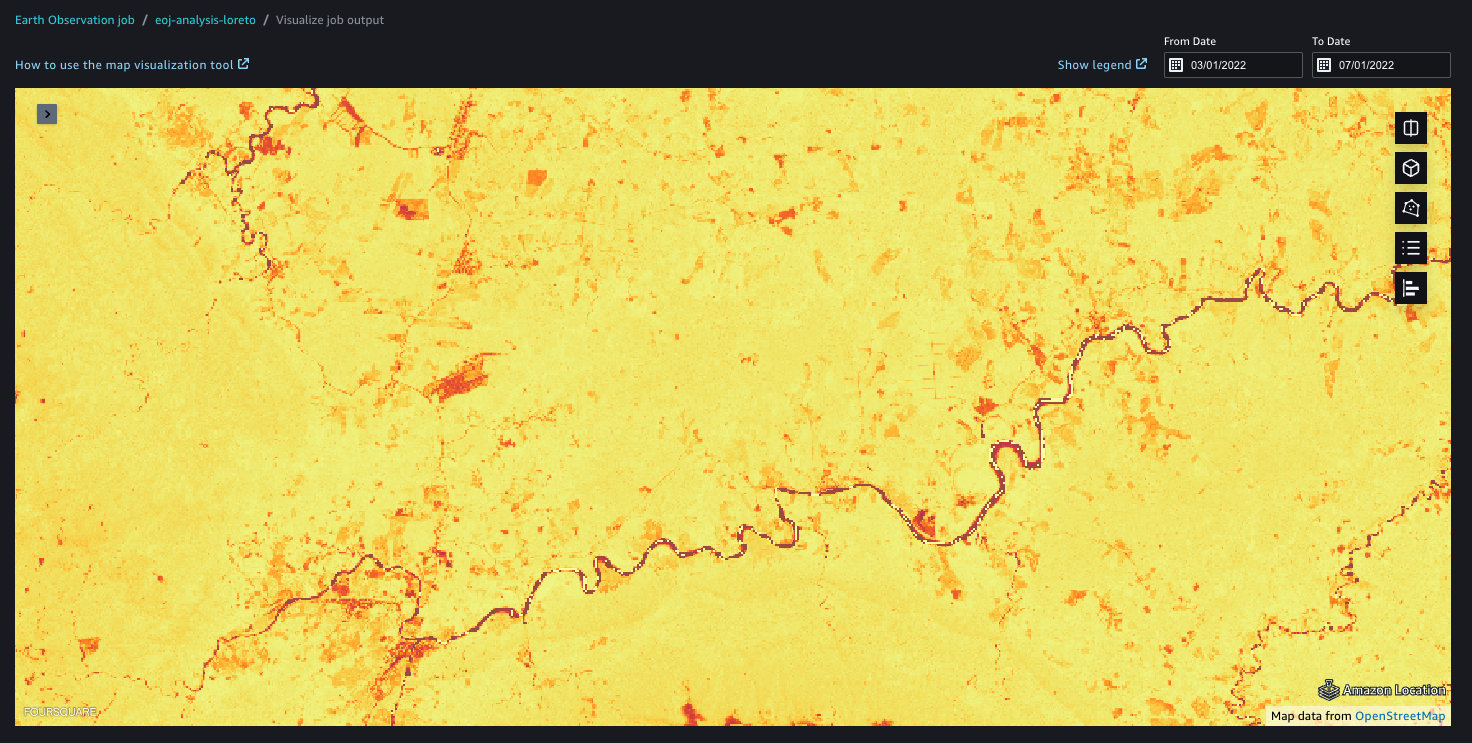
Satellitdata utgör en hörnsten i den analys som utförs för att bygga upp riskindexet, vilket ger kritisk information om miljöförändringar. För att generera insikter från satellitbilder, HSR.hälsa användningar Jordobservation jobb (EOJs). EOJs möjliggör förvärv och transformation av rasterdata som samlats in från jordens yta. En EOJ erhåller satellitbilder från en angiven datakälla – till exempel en satellitkonstellation – över ett specifikt område och tidsperiod. Den applicerar sedan en eller flera modeller på de hämtade bilderna.
Dessutom används Amazon SageMaker Studio erbjuder en geospatial anteckningsbok förinstallerad med ofta använda geospatiala bibliotek. Den här anteckningsboken möjliggör direkt visualisering och bearbetning av geospatial data i en Python-anteckningsbokmiljö. EOJ kan skapas i den geospatiala notebook-miljön.
För att konfigurera en EOJ används följande parametrar:
- InputConfig – Ingångskonfigurationen anger datakällorna och filtreringskriterierna som ska användas under datainsamlingen:
- RasterDataCollectionArn – Anger från vilken satellit data ska samlas in.
- Intresseområde – Det geografiska området av intresse (AOI) definierar polygongränserna för bildinsamling.
- TimeRangeFilter – Tidsintervallet av intresse:
{StartTime: <string>, EndTime: <string>}. - PropertyFilters – Ytterligare egenskapsfilter, såsom acceptabel andel molntäckning eller önskade solazimutvinklar.
- JobConfig – Den här konfigurationen definierar vilken typ av jobb som ska tillämpas på den hämtade satellitbildsdatan. Det stöder operationer som bandmatematik, omsampling, borttagning av geomosaik eller moln.
Följande exempelkod visar att en EOJ körs för molnborttagning, representativ för de steg som utförs av HSR.hälsa:
HSR.hälsa använde flera operationer för att förbehandla data och extrahera relevanta funktioner. Detta inkluderar operationer som klassificering av marktäcke, kartläggning av temperaturvariationer och vegetationsindex.
Ett vegetationsindex som är relevant för att indikera vegetationshälsa är Normalized Difference Vegetation Index (NDVI). NDVI kvantifierar vegetationens hälsa genom att använda nära-infrarött ljus, vilket vegetationen reflekterar, och rött ljus, som vegetationen absorberar. Övervakning av NDVI över tid kan avslöja förändringar i vegetationen, såsom effekterna av mänskliga aktiviteter som avskogning.
Följande kodavsnitt visar hur man beräknar ett vegetationsindex som NDVI baserat på data som har skickats genom molnborttagning:
Vi kan visualisera jobbresultatet med SageMakers geospatiala funktioner. SageMakers geospatiala funktioner kan hjälpa dig att lägga över modellförutsägelser på en baskarta och tillhandahålla visualisering i lager för att göra samarbetet enklare. Med den GPU-drivna interaktiva visualizern och Python-datorer är det möjligt att utforska miljontals datapunkter i en vy, vilket underlättar samarbetsutforskningen av insikter och resultat.
Stegen som beskrivs i det här inlägget visar bara en av de många rasterbaserade funktionerna som HSR.hälsa har extraherat för att skapa riskindex.
Kombinera rasterbaserade funktioner med hälso- och socialdata
Efter att ha extraherat de relevanta funktionerna i rasterformat, HSR.hälsa använt zonstatistik för att aggregera rasterdata inom de administrativa gränspolygoner som social- och hälsodata är tilldelade. Analysen innefattar en kombination av geospatiala raster- och vektordata. Denna typ av aggregering möjliggör hantering av rasterdata i en geodataram, vilket underlättar dess integration med hälso- och socialdata för att producera det slutliga riskindexet.
Följande kodavsnitt visar hur man aggregerar rasterdata till administrativa vektorgränser:
För att utvärdera de extraherade funktionerna effektivt, används ML-modeller för att förutsäga faktorer som representerar varje funktion. En av modellerna som används är en stödvektormaskin (SVM). SVM-modellen hjälper till att avslöja mönster och associationer inom data som ger underlag för riskbedömningar.
Indexet representerar en kvantitativ bedömning av risknivåer, beräknade som ett vägt genomsnitt av dessa faktorer, för att hjälpa till att förstå potentiella spridningshändelser i olika regioner.
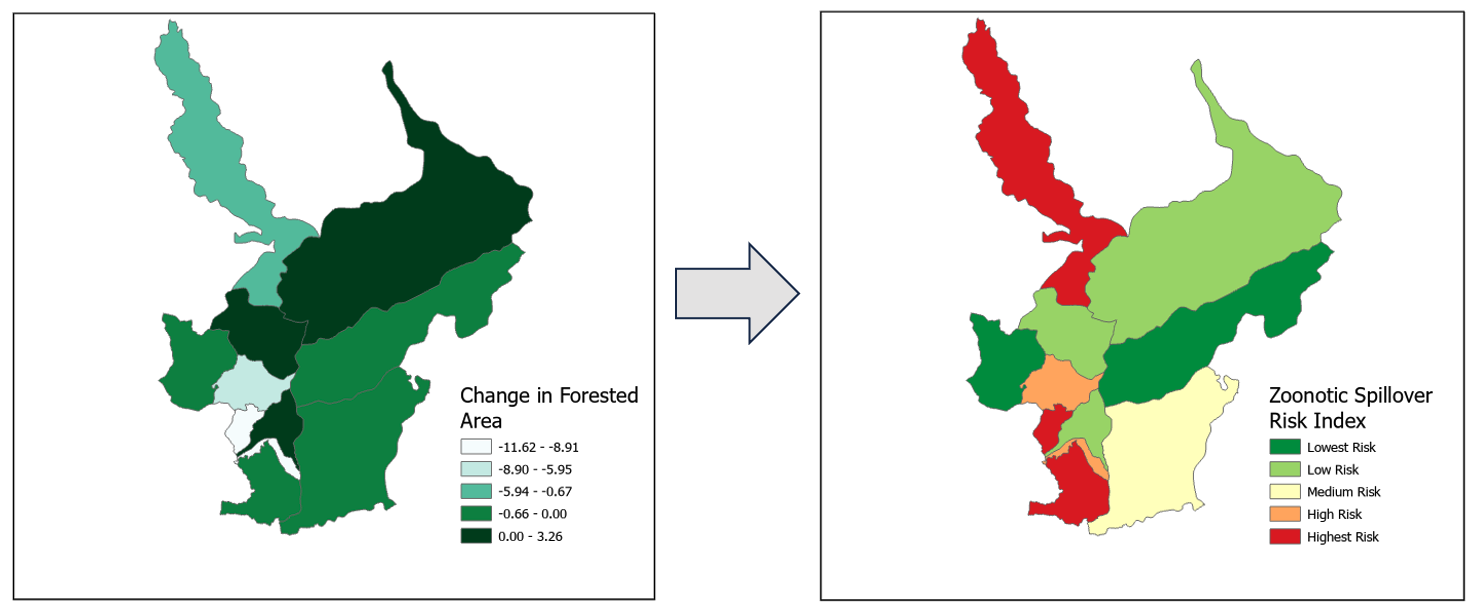
Följande figur till vänster visar aggregeringen av bildklassificeringen från testområdesscenen i norra Peru aggregerad till distriktsadministrativ nivå med den beräknade förändringen av skogsarealen mellan 2018–2023. Avskogning är en av nyckelfaktorerna som avgör risken för zoonotisk spillover. Figuren till höger belyser risken för zoonotisk spridningsrisk inom de täckta regionerna, från högsta (röd) till lägsta (mörkgrön) risk. Området valdes som ett av träningsområdena för bildklassificeringen på grund av mångfalden av marktäcke som fångats på scenen, inklusive: stad, skog, sand, vatten, gräsmark och jordbruk, bland annat. Dessutom är detta ett av många områden av intresse för potentiella zoonotiska spillover-händelser på grund av avskogning och interaktion mellan människor och djur.
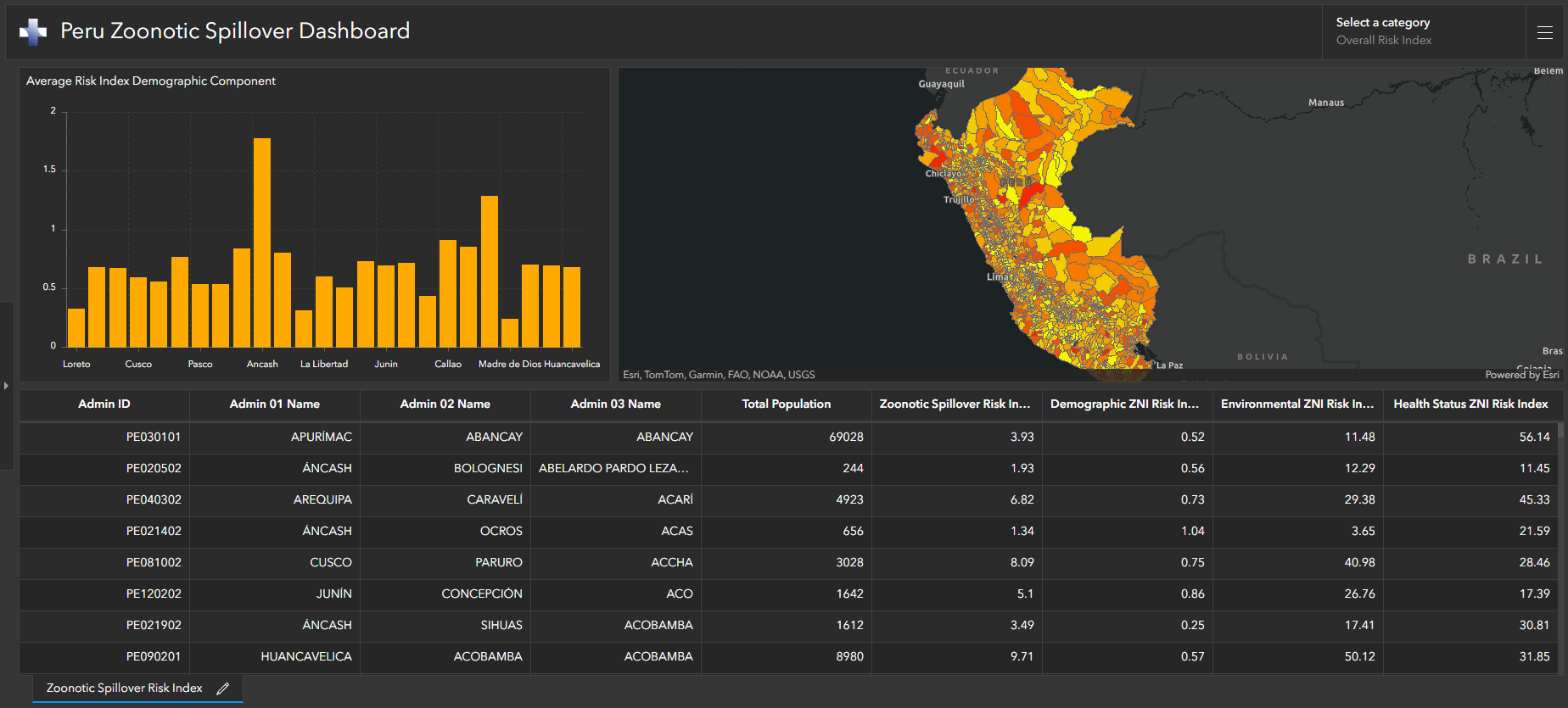
Genom att använda detta multimodala tillvägagångssätt, som omfattar historiska data om sjukdomsutbrott, jordobservationsdata, sociala bestämningsfaktorer och ML-tekniker, kan vi bättre förstå och förutsäga zoonotisk spridningsrisk, och i slutändan rikta sjukdomsövervakning och förebyggande strategier till områden med störst utbrottsrisk. Följande skärmdump visar en instrumentpanel med resultatet från en zoonotisk riskanalys. Denna riskanalys belyser var resurser och övervakning för nya potentiella zoonotiska utbrott kan inträffa så att nästa sjukdom kan begränsas innan den blir en endemisk eller en ny pandemi.
En ny metod för att förebygga pandemi
År 1998, längs Nipah-floden i Malaysia, mellan hösten 1998 och våren 1999, infekterades 265 personer med ett då okänt virus som orsakade akut hjärninflammation och svår andningsbesvär. 105 av dem dog, en dödlighet på 39.6 %. Covid-19s obehandlade dödlighet är däremot 6.3 %. Sedan dess har Nipah-viruset, som det nu kallas, övergått från sin skogsmiljö och orsakat över 20 dödliga utbrott, mestadels i Indien och Bangladesh.
Virus som Nipah dyker upp varje år och utgör utmaningar för vårt dagliga liv, särskilt i länder där det är svårare att etablera starka, varaktiga och robusta system för sjukdomsövervakning och upptäckt. Dessa detektionssystem är avgörande för att minska riskerna förknippade med sådana virus.
Lösningar som använder ML och geospatial data, såsom Zoonotic Spillover Risk Index, kan hjälpa lokala folkhälsomyndigheter att prioritera resursallokering till områden med högst risk. Genom att göra det kan de upprätta riktade och lokaliserade övervakningsåtgärder för att upptäcka och stoppa regionala utbrott innan de sträcker sig utanför gränserna. Detta tillvägagångssätt kan avsevärt begränsa effekterna av ett sjukdomsutbrott och rädda liv.
Slutsats
Det här inlägget visade hur HSR.hälsa framgångsrikt utvecklat Zoonotic Spillover Risk Index genom att integrera geospatiala data, hälsa, sociala bestämningsfaktorer och ML. Genom att använda SageMaker skapade teamet ett skalbart arbetsflöde som kan identifiera de mest betydande hoten från en potentiell framtida pandemi. Effektiv hantering av dessa risker kan leda till en minskning av den globala sjukdomsbördan. De betydande ekonomiska och sociala fördelarna med att minska pandemirisken kan inte överskattas, med fördelarna som sträcker sig regionalt och globalt.
HSR.hälsa använde SageMakers geospatiala möjligheter för en första implementering av Zoonotic Spillover Risk Index och söker nu partnerskap, såväl som stöd från värdländer och finansieringskällor, för att utveckla indexet ytterligare och utöka dess tillämpning till ytterligare regioner runt om i världen. För mer information om HSR.hälsa och Zoonotic Spillover Risk Index, besök www.hsr.health.
Upptäck potentialen med att integrera jordobservationsdata i dina vårdinitiativ genom att utforska SageMakers geospatiala funktioner. För mer information, se Amazon SageMaker geospatiala funktioner, eller samarbeta med ytterligare exempel för att få praktisk erfarenhet.
Om författarna
 Ajay K Gupta är medgrundare och VD för HSR.health, ett företag som stör och förnyar hälsoriskanalyser genom geospatial teknik och AI-tekniker för att förutsäga sjukdomens spridning och svårighetsgrad. Och ger dessa insikter till industri, regeringar och hälsosektorn så att de kan förutse, mildra och dra nytta av framtida risker. Utanför jobbet kan du hitta Ajay bakom mikrofonens sprängande trumhinnor samtidigt som han tar fram sina favoritpoplåtar från U2, Sting, George Michael eller Imagine Dragons.
Ajay K Gupta är medgrundare och VD för HSR.health, ett företag som stör och förnyar hälsoriskanalyser genom geospatial teknik och AI-tekniker för att förutsäga sjukdomens spridning och svårighetsgrad. Och ger dessa insikter till industri, regeringar och hälsosektorn så att de kan förutse, mildra och dra nytta av framtida risker. Utanför jobbet kan du hitta Ajay bakom mikrofonens sprängande trumhinnor samtidigt som han tar fram sina favoritpoplåtar från U2, Sting, George Michael eller Imagine Dragons.
 Jean Felipe Teotonio är en driven läkare och passionerad expert inom hälso- och sjukvårdens kvalitet och epidemiologi av infektionssjukdomar, Jean Felipe leder HSR.health folkhälsoteamet. Han arbetar mot det gemensamma målet att förbättra folkhälsan genom att minska den globala sjukdomsbördan genom att utnyttja GeoAI-metoder för att utveckla lösningar för vår tids största hälsoutmaningar. Utanför jobbet inkluderar hans hobbyer att läsa sci-fi-böcker, vandring, engelska Premier League och spela bas.
Jean Felipe Teotonio är en driven läkare och passionerad expert inom hälso- och sjukvårdens kvalitet och epidemiologi av infektionssjukdomar, Jean Felipe leder HSR.health folkhälsoteamet. Han arbetar mot det gemensamma målet att förbättra folkhälsan genom att minska den globala sjukdomsbördan genom att utnyttja GeoAI-metoder för att utveckla lösningar för vår tids största hälsoutmaningar. Utanför jobbet inkluderar hans hobbyer att läsa sci-fi-böcker, vandring, engelska Premier League och spela bas.
 Paul A Kyrkogård, CTO och Chief Geospatial Engineer för HSR.health, använder sin breda tekniska kompetens och expertis för att bygga kärninfrastrukturen för företaget såväl som dess patenterade och proprietära GeoMD-plattform. Dessutom införlivar han och datavetenskapsteamet geospatial analys och AI/ML-tekniker i alla hälsoriskindex som HSR.health producerar. Utanför jobbet är Paul en självlärd DJ och älskar snö.
Paul A Kyrkogård, CTO och Chief Geospatial Engineer för HSR.health, använder sin breda tekniska kompetens och expertis för att bygga kärninfrastrukturen för företaget såväl som dess patenterade och proprietära GeoMD-plattform. Dessutom införlivar han och datavetenskapsteamet geospatial analys och AI/ML-tekniker i alla hälsoriskindex som HSR.health producerar. Utanför jobbet är Paul en självlärd DJ och älskar snö.
 Janosch Woschitz är Senior Solutions Architect på AWS, specialiserad på geospatial AI/ML. Med över 15 års erfarenhet stödjer han kunder globalt med att utnyttja AI och ML för innovativa lösningar som drar nytta av geospatial data. Hans expertis spänner över maskininlärning, datateknik och skalbara distribuerade system, förstärkt av en stark bakgrund inom mjukvaruteknik och branschexpertis inom komplexa domäner som autonom körning.
Janosch Woschitz är Senior Solutions Architect på AWS, specialiserad på geospatial AI/ML. Med över 15 års erfarenhet stödjer han kunder globalt med att utnyttja AI och ML för innovativa lösningar som drar nytta av geospatial data. Hans expertis spänner över maskininlärning, datateknik och skalbara distribuerade system, förstärkt av en stark bakgrund inom mjukvaruteknik och branschexpertis inom komplexa domäner som autonom körning.
 Emmett Nelson är en Account Executive på AWS som stödjer Nonprofit Research-kunder inom sektorerna Healthcare & Life Sciences, Earth/ Environmental Sciences och Education. Hans primära fokus är att möjliggöra användningsfall inom analys, AI/ML, högpresterande beräkningar (HPC), genomik och medicinsk bildbehandling. Emmett gick med i AWS 2020 och är baserad i Austin, TX.
Emmett Nelson är en Account Executive på AWS som stödjer Nonprofit Research-kunder inom sektorerna Healthcare & Life Sciences, Earth/ Environmental Sciences och Education. Hans primära fokus är att möjliggöra användningsfall inom analys, AI/ML, högpresterande beräkningar (HPC), genomik och medicinsk bildbehandling. Emmett gick med i AWS 2020 och är baserad i Austin, TX.
- SEO-drivet innehåll och PR-distribution. Bli förstärkt idag.
- PlatoData.Network Vertical Generative Ai. Styrka dig själv. Tillgång här.
- PlatoAiStream. Web3 Intelligence. Kunskap förstärkt. Tillgång här.
- Platoesg. Kol, CleanTech, Energi, Miljö, Sol, Avfallshantering. Tillgång här.
- PlatoHealth. Biotech och kliniska prövningar Intelligence. Tillgång här.
- Källa: https://aws.amazon.com/blogs/machine-learning/how-hsr-health-is-limiting-risks-of-disease-spillover-from-animals-to-humans-using-amazon-sagemaker-geospatial-capabilities/