I dette innlegget demonstrerer vi hvordan du effektivt finjusterer en toppmoderne proteinspråkmodell (pLM) for å forutsi protein subcellulær lokalisering ved å bruke Amazon SageMaker.
Proteiner er kroppens molekylære maskiner, ansvarlige for alt fra å bevege musklene til å reagere på infeksjoner. Til tross for denne variasjonen, er alle proteiner laget av repeterende kjeder av molekyler kalt aminosyrer. Det menneskelige genomet koder for 20 standard aminosyrer, hver med en litt forskjellig kjemisk struktur. Disse kan representeres av bokstaver i alfabetet, som deretter lar oss analysere og utforske proteiner som en tekststreng. Det enorme mulige antallet proteinsekvenser og strukturer er det som gir proteiner deres mange forskjellige bruksområder.
Proteiner spiller også en nøkkelrolle i legemiddelutvikling, som potensielle mål, men også som terapeutiske midler. Som vist i tabellen nedenfor, var mange av de mest solgte legemidlene i 2022 enten proteiner (spesielt antistoffer) eller andre molekyler som mRNA oversatt til proteiner i kroppen. På grunn av dette må mange livsvitenskapsforskere svare på spørsmål om proteiner raskere, billigere og mer nøyaktig.
| Navn | Produsent | Globalt salg i 2022 (milliarder USD) | Indikasjoner |
| Comirnaty | Pfizer / BioNTech | $40.8 | Covid-19 |
| Spikevax | moderne | $21.8 | Covid-19 |
| Humira | AbbVie | $21.6 | Leddgikt, Crohns sykdom og andre |
| keytruda | Merck | $21.0 | Ulike kreftformer |
Datakilde: Urquhart, L. Toppbedrifter og legemidler etter salg i 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Fordi vi kan representere proteiner som sekvenser av tegn, kan vi analysere dem ved å bruke teknikker som opprinnelig ble utviklet for skriftspråk. Dette inkluderer store språkmodeller (LLM) som er forhåndsopplært på enorme datasett, som deretter kan tilpasses for spesifikke oppgaver, som tekstoppsummering eller chatbots. På samme måte er pLM-er forhåndstrenet på store proteinsekvensdatabaser ved bruk av umerket, selvovervåket læring. Vi kan tilpasse dem til å forutsi ting som 3D-strukturen til et protein eller hvordan det kan samhandle med andre molekyler. Forskere har til og med brukt pLM-er for å designe nye proteiner fra bunnen av. Disse verktøyene erstatter ikke menneskelig vitenskapelig ekspertise, men de har potensiale til å fremskynde preklinisk utvikling og utforming av forsøk.
En utfordring med disse modellene er størrelsen. Både LLM-er og pLM-er har vokst i størrelsesordener de siste årene, som illustrert i følgende figur. Dette betyr at det kan ta lang tid å lære dem opp til tilstrekkelig nøyaktighet. Det betyr også at du må bruke maskinvare, spesielt GPUer, med store mengder minne for å lagre modellparametrene.
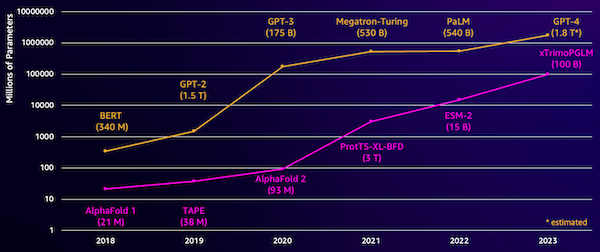
Lang opplæringstid, pluss store forekomster, tilsvarer høye kostnader, noe som kan sette dette arbeidet utenfor rekkevidde for mange forskere. For eksempel, i 2023, en Forskerteamet teamet~~POS=HEADCOMP beskrev å trene en pLM med 100 milliarder parametere på 768 A100 GPUer i 164 dager! Heldigvis kan vi i mange tilfeller spare tid og ressurser ved å tilpasse en eksisterende pLM til vår spesifikke oppgave. Denne teknikken kalles finjustering, og lar oss også låne avanserte verktøy fra andre typer språkmodellering.
Løsningsoversikt
Det spesifikke problemet vi tar opp i dette innlegget er subcellulær lokalisering: Gitt en proteinsekvens, kan vi bygge en modell som kan forutsi om den lever på utsiden (cellemembranen) eller innsiden av en celle? Dette er en viktig informasjon som kan hjelpe oss å forstå funksjonen og om den vil være et godt stoffmål.
Vi starter med å laste ned et offentlig datasett vha Amazon SageMaker Studio. Deretter bruker vi SageMaker til å finjustere ESM-2-proteinspråkmodellen ved hjelp av en effektiv treningsmetode. Til slutt distribuerer vi modellen som et endepunkt for sanntidsslutning og bruker den til å teste noen kjente proteiner. Følgende diagram illustrerer denne arbeidsflyten.

I de følgende delene går vi gjennom trinnene for å forberede treningsdataene dine, lage et treningsskript og kjøre en SageMaker-treningsjobb. All koden i dette innlegget er tilgjengelig på GitHub.
Forbered treningsdataene
Vi bruker en del av DeepLoc-2 datasett, som inneholder flere tusen SwissProt-proteiner med eksperimentelt bestemte lokasjoner. Vi filtrerer etter sekvenser av høy kvalitet mellom 100–512 aminosyrer:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Deretter tokeniserer vi sekvensene og deler dem inn i trenings- og evalueringssett:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Til slutt laster vi opp de behandlede opplærings- og evalueringsdataene til Amazon enkel lagringstjeneste (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Lag et treningsskript
SageMaker skriptmodus lar deg kjøre din egendefinerte opplæringskode i rammeverkbeholdere for optimalisert maskinlæring (ML) administrert av AWS. For dette eksemplet tilpasser vi en eksisterende skript for tekstklassifisering fra Hugging Face. Dette lar oss prøve flere metoder for å forbedre effektiviteten i treningsjobben vår.
Metode 1: Vektet treningstime
Som mange biologiske datasett er DeepLoc-dataene ujevnt fordelt, noe som betyr at det ikke er like mange membran- og ikke-membranproteiner. Vi kan prøve dataene våre på nytt og forkaste poster fra majoritetsklassen. Dette vil imidlertid redusere de totale treningsdataene og potensielt skade nøyaktigheten vår. I stedet beregner vi klassevektene under treningsjobben og bruker dem til å justere tapet.
I treningsskriptet vårt underklasser vi Trainer klasse fra transformers med en WeightedTrainer klasse som tar hensyn til klassevekter ved beregning av kryssentropitap. Dette bidrar til å forhindre skjevhet i modellen vår:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMetode 2: Gradientakkumulering
Gradientakkumulering er en treningsteknikk som lar modeller simulere trening på større batchstørrelser. Vanligvis er batchstørrelsen (antall prøver som brukes til å beregne gradienten i ett treningstrinn) begrenset av GPU-minnekapasiteten. Med gradientakkumulering beregner modellen gradienter på mindre partier først. Så, i stedet for å oppdatere modellvektene med en gang, blir gradientene akkumulert over flere små batcher. Når de akkumulerte gradientene tilsvarer målet for større batchstørrelse, utføres optimaliseringstrinnet for å oppdatere modellen. Dette lar modeller trene med effektivt større batcher uten å overskride GPU-minnegrensen.
Imidlertid er det nødvendig med ekstra beregning for de mindre partiene fremover og bakover. Økte batchstørrelser via gradientakkumulering kan bremse treningen, spesielt hvis det brukes for mange akkumuleringstrinn. Målet er å maksimere GPU-bruken, men unngå overdreven nedgang fra for mange ekstra gradientberegningstrinn.
Metode 3: Gradientkontroll
Gradient checkpointing er en teknikk som reduserer minnet som trengs under trening, samtidig som beregningstiden holdes rimelig. Store nevrale nettverk tar opp mye minne fordi de må lagre alle mellomverdiene fra foroverpasseringen for å beregne gradientene under bakoverpasseringen. Dette kan forårsake minneproblemer. En løsning er å ikke lagre disse mellomverdiene, men da må de beregnes på nytt under bakoverpasseringen, noe som tar mye tid.
Gradientkontroll gir en balansert tilnærming. Den lagrer bare noen av de mellomliggende verdiene, kalt sjekkpunkter, og beregner de andre på nytt etter behov. Derfor bruker den mindre minne enn å lagre alt, men også mindre beregning enn å beregne alt på nytt. Ved å strategisk velge hvilke aktiveringer som skal kontrolleres, gjør gradientkontrollpunkt det mulig å trene store nevrale nettverk med håndterbar minnebruk og beregningstid. Denne viktige teknikken gjør det mulig å trene veldig store modeller som ellers ville ha hukommelsesbegrensninger.
I treningsskriptet vårt slår vi på gradientaktivering og sjekkpunkt ved å legge til de nødvendige parameterne til TrainingArguments gjenstand:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Metode 4: Lavrangstilpasning av LLM-er
Store språkmodeller som ESM-2 kan inneholde milliarder av parametere som er dyre å trene og kjøre. Forskere utviklet en treningsmetode kalt Low-Rank Adaptation (LoRA) for å gjøre finjusteringen av disse enorme modellene mer effektiv.
Nøkkelideen bak LoRA er at når du finjusterer en modell for en spesifikk oppgave, trenger du ikke å oppdatere alle de originale parameterne. I stedet legger LoRA til nye mindre matriser til modellen som transformerer inngangene og utgangene. Bare disse mindre matrisene oppdateres under finjustering, som er mye raskere og bruker mindre minne. De originale modellparametrene forblir frosne.
Etter finjustering med LoRA kan du slå sammen de små tilpassede matrisene tilbake til den opprinnelige modellen. Eller du kan holde dem adskilt hvis du raskt vil finjustere modellen for andre oppgaver uten å glemme tidligere. Samlet sett lar LoRA LLM-er effektivt tilpasses nye oppgaver til en brøkdel av den vanlige kostnaden.
I opplæringsskriptet vårt konfigurerer vi LoRA ved å bruke PEFT bibliotek fra Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Send inn en SageMaker-treningsjobb
Etter at du har definert treningsskriptet ditt, kan du konfigurere og sende inn en SageMaker-treningsjobb. Først spesifiser hyperparametrene:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Deretter definerer du hvilke beregninger som skal hentes fra treningsloggene:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Definer til slutt en Hugging Face-estimator og send den inn for trening på en ml.g5.2xlarge forekomsttype. Dette er en kostnadseffektiv forekomsttype som er allment tilgjengelig i mange AWS-regioner:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)Tabellen nedenfor sammenligner de forskjellige treningsmetodene vi diskuterte og deres effekt på kjøretid, nøyaktighet og GPU-minnekrav for jobben vår.
| Konfigurasjon | Fakturerbar tid (min) | Evalueringsnøyaktighet | Maks GPU-minnebruk (GB) |
| Basismodell | 28 | 0.91 | 22.6 |
| Base + GA | 21 | 0.90 | 17.8 |
| Base + GC | 29 | 0.91 | 10.2 |
| Base + LoRA | 23 | 0.90 | 18.6 |
Alle metodene produserte modeller med høy evalueringsnøyaktighet. Bruk av LoRA og gradientaktivering reduserte kjøretiden (og kostnadene) med henholdsvis 18 % og 25 %. Bruk av gradientkontrollpunkt reduserte maksimal GPU-minnebruk med 55 %. Avhengig av begrensningene dine (kostnad, tid, maskinvare), kan en av disse tilnærmingene være mer fornuftige enn en annen.
Hver av disse metodene fungerer godt i seg selv, men hva skjer når vi bruker dem i kombinasjon? Følgende tabell oppsummerer resultatene.
| Konfigurasjon | Fakturerbar tid (min) | Evalueringsnøyaktighet | Maks GPU-minnebruk (GB) |
| Alle metoder | 12 | 0.80 | 3.3 |
I dette tilfellet ser vi en reduksjon på 12 % i nøyaktigheten. Vi har imidlertid redusert kjøretiden med 57 % og GPU-minnebruken med 85 %! Dette er en massiv nedgang som lar oss trene på et bredt spekter av kostnadseffektive instanstyper.
Rydd opp
Hvis du følger med på din egen AWS-konto, slett eventuelle sanntidsslutningsendepunkter og data du opprettet for å unngå ytterligere kostnader.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()konklusjonen
I dette innlegget demonstrerte vi hvordan man effektivt finjusterer proteinspråkmodeller som ESM-2 for en vitenskapelig relevant oppgave. For mer informasjon om bruk av Transformers og PEFT bibliotekene til å trene pLMS, sjekk ut innleggene Dyplæring med proteiner og ESMBind (ESMB): Lav rangering av ESM-2 for prediksjon av proteinbindingssted på Hugging Face-bloggen. Du kan også finne flere eksempler på bruk av maskinlæring for å forutsi proteinegenskaper i Fantastisk proteinanalyse på AWS GitHub-depot.
om forfatteren
 Brian lojal er senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han har mer enn 17 års erfaring innen bioteknologi og maskinlæring, og brenner for å hjelpe kunder med å løse genomiske og proteomiske utfordringer. På fritiden liker han å lage mat og spise sammen med venner og familie.
Brian lojal er senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han har mer enn 17 års erfaring innen bioteknologi og maskinlæring, og brenner for å hjelpe kunder med å løse genomiske og proteomiske utfordringer. På fritiden liker han å lage mat og spise sammen med venner og familie.
- SEO-drevet innhold og PR-distribusjon. Bli forsterket i dag.
- PlatoData.Network Vertical Generative Ai. Styrk deg selv. Tilgang her.
- PlatoAiStream. Web3 Intelligence. Kunnskap forsterket. Tilgang her.
- PlatoESG. Karbon, CleanTech, Energi, Miljø, Solenergi, Avfallshåndtering. Tilgang her.
- PlatoHelse. Bioteknologisk og klinisk etterretning. Tilgang her.
- kilde: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



