Genemining van NLR-genen in Arachis soorten
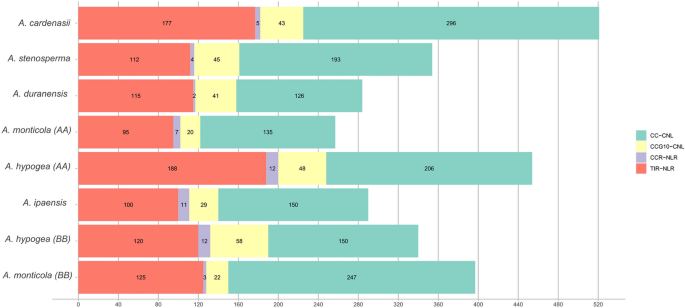
Hier hebben we gebruik gemaakt van de NLR-trackerpijplijn15 voor NLR-genenontginning en opeenvolgende annotaties. In het geval van wilde diploïde voorouderlijke soorten voor A-genoom werden in totaal 521, 354, 284 NLR-genen geïdentificeerd uit A. cardenasii, A. stenosperma en A. duranensis. In totaal werden 257 en 454 geïdentificeerd uit A-subgenoom van wild (A. monticola) en gedomesticeerd (A. hypogea) tetraploïden (AABB) respectievelijk (Fig. 1). Terwijl B-genomen soorten inclusief A. ipaensis, A. monticola, A. hypogaea bevatten 290, 397 en 340 NLR-genen in hun diverse repertoire. Interessant is dat A-subgenoom van gedomesticeerde tetraploïde soorten (A. hypogea) onthulde de forse uitbreiding in het NLR in tegenstelling tot A. monticola waar een verminderd aantal NLR-genen werd geïdentificeerd. Integendeel B-subgenoom van gedomesticeerde tetraploïde A. hypogea geopenbaarde samentrekking in vergelijking met van A. monticola. Bovendien onder de wilde soorten A. cardenasi toont onder meer het uitgebreide NLRome-repertoire Arachis soorten (afb. 1). Alle vier klassen van NLR-genen waren aanwezig in alle leden van Arachis geslacht. Over het algemeen hebben CC-NLR de hoogste bijdrage van andere klassen laten zien, gemiddeld 51.35% CC-NLR, 35% TIR, 12.1% CCG10 en 1-2% CCR -NLR-genen werden geïdentificeerd in leden van het geslacht Arachis. Interessant is dat helper NLR in relatief grote aantallen aanwezig was in tetraploïde soorten, vooral in A. hypogea waarbij zowel het AA- als het BB-genoom 12 CC delenR-NLR en alle diploïde leden bezitten 3-5 CCR behalve A. ipeansis waar 11 ccR helpergenen werden gemeld. Het is consistent met de eerdere waarneming dat polyploïdisatie het aantal bepaalde genenfamilies kan verhogen of verlagen32, hier CCR toont symmetrische expansie in A. hypogea in beide genomen. Verdeling van NLR-lengte, lengte van geconserveerde NB-ARC en soortgewijze domeinorganisatie wordt ook gegeven (Figuur S1, S2, S3).
Landschap van NLR-genen tussen genus Arachis
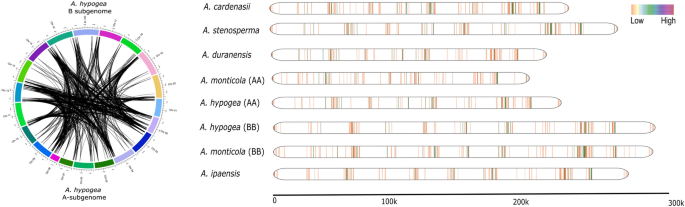
We vergeleken ook de syntenische relatie tussen A. hypogea subgenomen en hun voorloper. Sterk geconserveerde homeologenclusters werden geïdentificeerd in de syntenische vergelijking tussen A-subgenoom en B-subgenoom van A. hypogea (Figuur S4, A). Opgemerkt moet worden dat er minder syntenische relaties werden waargenomen voor vergelijking van elk subgenoom met zijn voorlopers (Figuur S4 B, C). We bestudeerden ook het landschap van NLR-genen in alle acht genomen van Arachis geslacht door de gendichtheid van NLR-genen uit te zetten op gelineariseerde chromosomen (Fig. 2). Interessant, A. cardeansii onthulde de hoogste genendichtheid met betrekking tot zijn grootte. We hebben ook het effect van allopolypoid waargenomen bij zowel wilde als gedomesticeerde tetraploïde soorten. Interessant is dat A-subgenoom samentrekking heeft laten zien in wilde tetraploïde en later een significante expansie vertoont bij domesticatie in A. hypogea. Integendeel, B-subgenoom van A. monticola aanzienlijk uitgebreid na allopolyploïdie met de op één na hoogste gendichtheid daarna A. cardenasi (Vijg 2). Over het algemeen suggereren syntenie- en gendichtheidskaarten sterk dat allopolyploïdisatie de uitbreiding van de NLR-gendichtheid bevordert Arachis soorten met uitzondering van A-subgenoom van A. monticola.
Daarnaast vergeleken we de architectuur van resistentiegenclusters (RGC's) in elke soort van het geslacht Arachis (Figuur S10). De meeste NLR-genen werden toegewezen in de vorm van RGC's. De meeste RGC's waren toegewezen aan Chr02, Chr04, Chr05, Chr08 en Chr09 (Figuur S10). Hoogste aantal van in totaal 29 RGC's gevonden in A. cardenasii en A. stenosperma en de minste aantallen werden waargenomen A. monticola (A-subgenoom). Interessant is dat het individuele aantal genen in elk cluster werd geamplificeerd A. cardenasii, A. hypogea (A-subgenoom) en A. monticola (B-subgenoom) suggereert een actieve rol van tandemduplicatie bij de uitbreiding van hun NLRome. Bovendien waren de aan- en afwezigheid van RGC's variabel in elk genomen.
Fylogenetische analyse en classificatie van NB-coderende genen
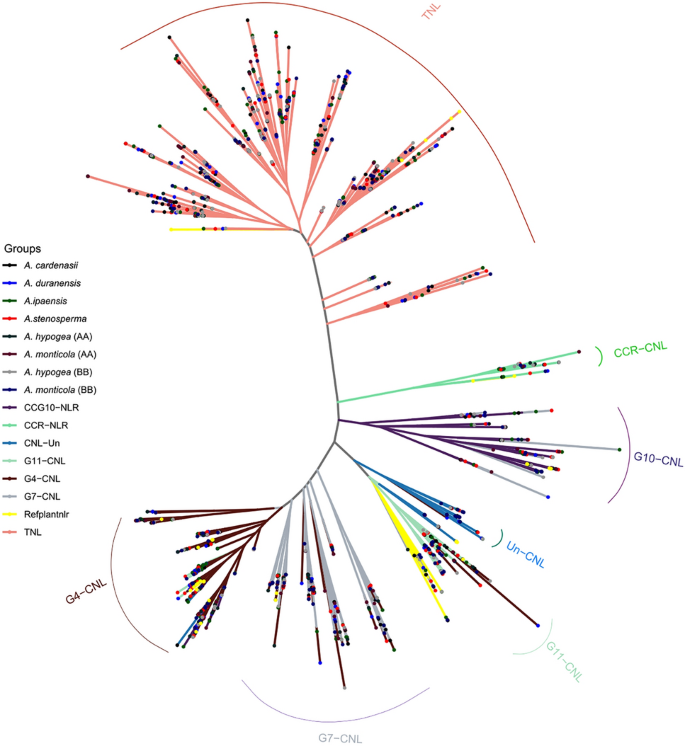
Van elk werd het geconserveerde NBARC-domein geëxtraheerd Arachis soorten en geclusterd op 75 procent sequentie-identiteit met behulp van CD-HIT33. Representatieve leden van elk cluster (totaal = 380) werden gebruikt voor de reconstructie van de fylogenetische relatie tussen A. stenosperma, A. duranensis, A. cardenasii, A. monticola (AA), A. Monticola (BB), A. hypogea (AA), A. hypogea (BB) en A. ipensis (Fig. 3). De TNL-clade was zoals verwacht vertakt, maar TNL bleef polyfyletisch en er werden drie grote stralingen waargenomen. Aan de andere kant was de CNL-clade verdeeld in drie monofyletische grote sub-clades CC-NLR, CCR-NLR en CCG10-NLR. CC-NLR werd verder onderverdeeld in vier grote subgroepen CNL-Un, CNL-G11, CNL-G7 en G4. Aanzienlijke uitbreiding en diversiteit werd waargenomen in G4 en vooral in G7, waar vier sterk ondersteunde polyfyletische sub-clades werden waargenomen. Interessant is dat de CNL-groepen G1, G2, G3, G4, G6, G8 eerder zijn geïdentificeerd uit Solanaceae familie waren afwezig in genus Arachis. Dat komt overeen met de onderzoeken van cicer en dalbergioïden, wat dat sterk suggereert Fabaceae leden missen G1-G8-groepen34,35.
Classificatie van subgroepen van NLR-genen met behulp van fylogenetische reconstructie. Fylogenetische boomconstructie is gebaseerd op de Maximum Likelihood-methode op het VT + F + R9-model. De boom is verdeeld in 7 CNL- en 1 TNL-subgroepen. Alle takken zijn gemarkeerd met hun subgroepspecifieke kleuren.
Fylogenetische analyse suggereert verder dat voorloper van AA-subgenoom, A. duranensis hadden minder TIR- en CC-NLR-genen. Na allopolyploïdie kan een significante uitbreiding van TIR- en CC-NLR-genen worden waargenomen. Het grootste aantal van deze groepen kan worden geïdentificeerd in het AA-subgenoom van gedomesticeerde tetraploïde A. hypogea. Interessant is dat tussen alle soorten A. cardenasi heeft het hoogste aantal TIR- en CC-NLR-genen gezien zijn diploïde aard. Deze onevenwichtige genduplicatie komt overal voor Arachis soorten suggereren mogelijke rol van terminale duplicatie na de afwijking van gemeenschappelijke voorouders.
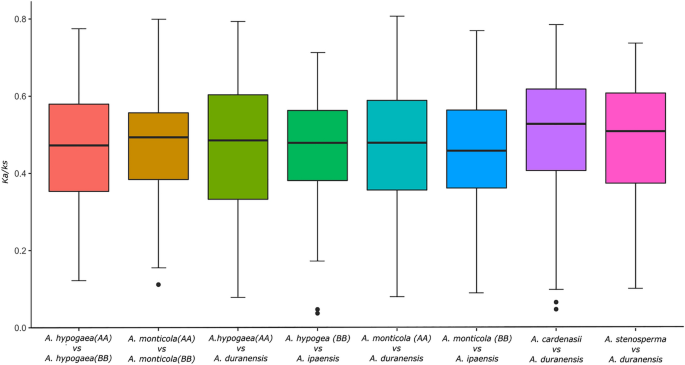
We vergeleken ook de selectiedruk binnen de paren paralogen van vier grote subgroepen (G4, G7, CCG10-NLR, TIR-NLR). G4 (mediaan = 0.502) en G7 (mediaan = 0.534) hebben de hoogste waarden van Ka/K's in vergelijking met de andere twee hoofdgroepen TIR-NLR (mediaan = 0.457) en G10 (mediaan = 0.427), die evolueerden onder zuiverende selectie. Deze waarneming komt overeen met het feit dat preferentiële uitbreiding van G4 en G7 ook in andere wordt waargenomen Fabacae geslacht cicer en dalbergia (niet-gepubliceerde resultaten). In het geval van A. monticola er werden hogere Ka/Ks-waarden van 1.002 waargenomen voor de CCG10-subgroep, wat suggereert dat deze evolueert onder neutrale selectie (Figuur S5, S6, Tafel S3).
Duplicatie-test
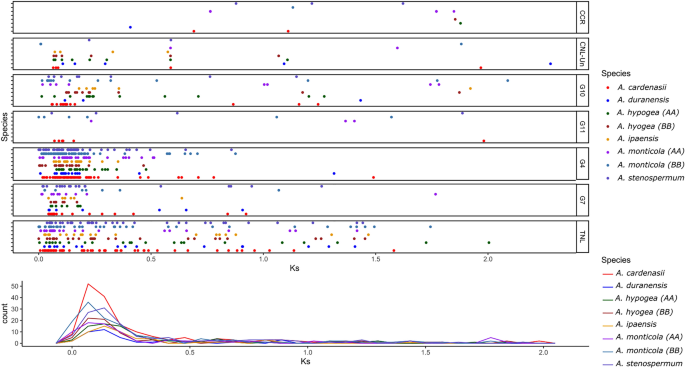
Uitgebreide NLRome van A. cardenasi kan het gevolg zijn van meerdere evolutionaire mechanismen, waaronder duplicatie, recombinatie en transpositie. Hier hebben we de duplicatiegeschiedenis van verkend Arachis NLR's door de Ks waarden tussen paralogen van elke subgroep. Met name de Arachis afstamming hebben zich snel geaccumuleerde stille veranderingen (~ 1.4 keer sneller) sinds de divergentie van de Dalbergioïde clade1. De beste schattingen voor divergentie tussen twee voorlopers van elke AA (A. duranensis) en BB (A. ipensis) subgenoom is onlangs berekend als 2.12 Mya32. De precieze schatting van de divergentie van andere soorten van de gemeenschappelijke voorouder wordt echter nog steeds niet gerapporteerd. Collectief Ks waarden verkregen uit alle groepen suggereren één gemeenschappelijke duplicatiecurve tussen 0.04 en 0.1 Ks (2.1-6 Mya) (Fig. 4, Tafel S3). Dat suggereert sterk dat NLR-genduplicatie heeft plaatsgevonden vóór de soortvorming. De hoogste frequentie voor genduplicatie werd waargenomen in A. cardenasi, waar piekwaarde van Ks komt overeen met 0.08 (~ 4.92 Mya). TNL en subgroepen G4-CNL, G7-CNL-gen waren dramatisch geamplificeerd door genduplicatiegebeurtenissen vóór speciatie. Zo ook andere soorten A. monticola (B-subgenoom) en A. stenosperma onthulde ook relatief hogere frequenties van genduplicatie. Interessant is dat de voorlopersoort A. duranensis en A. ipensis had de minste frequentie van Ks waarde voor genenparen. Verder hebben we ook genduplicatie getest met behulp van orthofinder (v 2.5.4: Fig. 5B, D). Overeenkomend met Ks schattingen, suggereert het dat in zowel A- als B-genoomsoorten de hoogste duplicatie werd waargenomen in de gemeenschappelijke voorouder van Arachis. Bovendien levert Orthofinder bewijs voor relatief hogere terminale duplicatie in A. cardenasi (95) en A. monticola (83: B-subgenoom) (Fig. 5B, D). Kortom, alle soorten vertegenwoordigen een gemeenschappelijke golf van duplicatie die leidde tot een grote uitbreiding in NLRome die plaatsvond in de gemeenschappelijke voorouder van het geslacht Arachis. Daarnaast werd terminale duplicatie ook waargenomen na speciatie in specifieke soorten die het repertoire van NLR-genen uitbreidden A. cardenasi en A. monticola (B-subgenoom).
Duplicatiegeschiedenis van NLR-genen in genus Arachis. Ks-waarden tussen paralogen van elke onderverdeling worden weergegeven voor alle zes soorten, waarbij tetraploïde soorten zijn onderverdeeld in hun samenstellende subgenomen. (A) X en Y vertegenwoordigen respectievelijk de Ks-waarden en frequenties (B). Algeheel duplicatiepatroon van NLR-genen in genus Arachis.
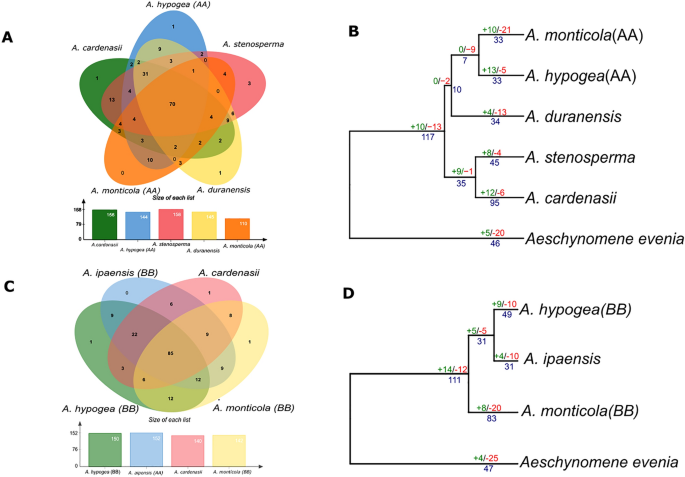
Ortholoog en analyse van winst en verlies van genen. (A, C) Venn-diagram vertegenwoordigt de gedeelde en gemeenschappelijke genen (orthologe clusters) verdeling gevonden tussen respectievelijk A-genoomgerelateerde soorten en B-genoomgerelateerde soorten. (B, D) Genwinst en -verlies worden op elk knooppunt aangegeven met het aantal genwinst (groen), verlies (rood) en duplicatie (blauw) voor respectievelijk A- en B-genoomgerelateerde soorten.
Gene winst en verlies
Er werden in totaal 70 gemeenschappelijke orthogroepen gevonden die geconserveerd waren in A-genoom-gerelateerde soorten, terwijl B-subgenoom 85 gemeenschappelijke orthogroepen heeft (Fig. 5A, C). We construeerden de fylogenetische boom voor elk subgenoom met geboorte en dood van genetische gebeurtenissen onder leden van het geslacht Arachis. Aeschynomeme evenia werd beschouwd als de meest verwante outgroup voor de gemeenschappelijke voorouder van Arachis. Geboorte- en doodsmodel van A-subgenoom onthult dat samentrekking van NLR-genfamilies plaatsvond Aesechynomen evenia wat consistent is met de algehele NLR-contractie na volledige genoomduplicatie na diploïdisatie. Bovendien gemeenschappelijke voorouder van het geslacht Arachis suggereren een groter aantal genduplicaties en winsten van 10 extra NLR-genfamilies (Fig. 5B, D). Voorloper van A-subgenoom A. duranensis en wilde tetraploïde A. monticola heeft de dood van genfamilies aangetoond, behalve A. hypogea waar een uitbreiding van het aantal NLR-gezinnen werd waargenomen. Zo ook A. cardenasi onthulde de hoogste uitbreiding van het aantal verschillende genfamilies, waarschijnlijk als gevolg van terminale duplicatie en kreeg 12 genfamilies (Fig. 5B, D). Een vergelijkbare trend werd gevonden in het geval van evolutie van het B-subgenoom, samentrekking in de outgroup-soort en uitbreiding van NLRome in de gemeenschappelijke voorouder Arachis Vooral in het geval van A. monticola (B-subgenoom) waar uitbreiding van NLR-genen plaatsvond die consistent is met uitbreiding van andere genfamilies, waaronder zetmeel- en suikermetabolisme, linolzuurmetabolisme en cutinesynthese32. Kortom, asymmetrische evolutie van NLR-genen in subgenoom A en B werd waargenomen bij wilde en gedomesticeerde tetraploïde soorten.
Impact van natuurlijke en kunstmatige selectiedruk op NLR-genen
We bestudeerden verder de impact van natuurlijke en kunstmatige selectie op NLR-genevolutie in respectievelijk wilde en gedomesticeerde tetraploïde soorten. Hiervoor vergeleken we de ka/ks verhouding van aanwezige orthologen tussen subgenomen en hun voorlopersoort (Fig. 6). Ka/K's waarden voor orthologen tussen A-subgenoom van A. monticola en A. duranensis waren significant hoger in A (mediaan = 0.479) in vergelijking met B-subgenoom (mediaan = 0.455). Evenzo, ka/ks waarden waren hoger in A (mediaan = 0.488) in vergelijking met subgenoom B (mediaan = 0.479) van A. hypogea (Fig. 6). Er werd een vertekening waargenomen in de selectiedruk voor A-subgenoom NLR-genen in zowel wilde als gedomesticeerde tetraploïde. We bestudeerden ook de aard van selectiedruk op twee vroege divergerende soorten van A-subgenoom, voor dit doel hebben we vergeleken ka/ks waarden orthologen van A. cardenasi en A. stenosperma rekeninghoudend met A. duranensis. Vooral deze soort vertoont de hoogste mate van natuurlijke selectie in vergelijking met andere wilde soorten A. cardenasi met de selectiedruk van M = 0.528. dat is mogelijk de reden voor een uitgebreid repertoire van NLR-genen.
Expressie-analyse van NLR-genen in Arachis soorten
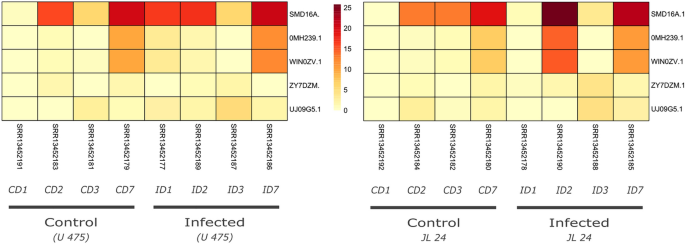
We vergeleken verder het basale expressieniveau van geïdentificeerde genen in A. hypogea in drie weefselpeul, zaad en schaal. In totaal werden 37 NLR-genen constitutief tot expressie gebracht in alle drie de weefseltypen, met name twee genen HV9GRN.1 en 256JRY.1 die behoort tot subgroep CCR-NLR en CCG10-NLR vertoont respectievelijk de hoogste expressieniveaus in alle weefseltypes (Figuur S7). In een andere studie evalueerden we de expressie van NLR-genen in susceptibe (JL 24) en resistente cultivar (U-475) van A. hypogea op Aspergillus flavius infectie (afb. 7). In beide cultivars werden 12 NLR-genen differentieel tot expressie gebracht en vertoonden een sterke correlatie met ziekteprogressie. Vooral drie genen (SMD16A.1, OMH239.1 en WIN0WV.1) onthulden hogere opwaartse regulatie tijdens 3 en 7 dpi in beide cultivars (Fig. 8). Alle drie de genen behoren tot de subgroep G4-CNL, de belangrijkste receptor die het coiled coil-domein bevat voor de herkenning van pathogenen. Interessant is dat er geen significante verschillen werden waargenomen in het expressieprofiel van NLR-genen in vatbaar en resistent genotype. Vermoedelijk kunnen andere resistentiegenen, waaronder receptorachtige kinasen (RLK) en receptorachtige eiwitten (RLP), verantwoordelijk zijn voor het verschil in hun genotype.
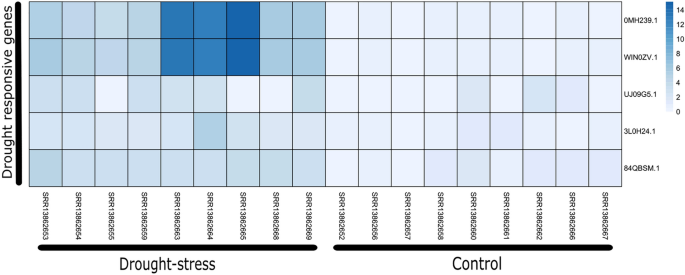
Onlangs werd gemeld dat NLR-genen ook een belangrijke rol spelen onder de omstandigheden van droogtestress36. Hier hebben we deze hypothese voor getest A. hypogea door de expressie van NLR-genen te vergelijken onder goed bewaterde versus droge omstandigheden. In deze dataset hebben we vijf op droogte reagerende genen geïdentificeerd (OMH239.1, WIN0ZV.1, UJJ09G5.1, 3L0H24.1 en 84QBSM.1) die tot overexpressie kwamen tijdens droogte. Aangezien deze dataset biologische replicatie bevat gedurende 5 dagen, 7 dagen en 9 dagen na de droogtesituatie (pds), hebben we interessant genoeg de hoogste expressie waargenomen tot 14-voudig in 9 dagen (pds), met name voor twee genen (0MH239.1, WIN0ZV.1) ( Afb. 8).
We evalueerden ook de expressie van NLR-genen in zowel tetraploïden en hun voorlopers als synthetische ontluikende interspecifieke hybriden en neopolyploïden. Bertoili et al.37 reconstrueerde de hybriden van A. duranensis x A. ipaensis en induceerde vervolgens polyploïdie door behandeling met colchicine. RNA-seq-analyse werd uitgevoerd op initiële diploïden, neopolyploïden (1e en 9e generatie). Geconserveerde NLR-genen die gemeenschappelijk zijn voor major Arachis soort heeft expressiebias getoond voor interspecifieke diploïde en neo-allopolyploïden. Hogere individuele en cumulatieve expressieniveaus werden waargenomen in synthetische interspecifieke diploïde (AB, A. duranensis x A. ipensis), neopolyploïde (4x) en A. monticola in vergelijking met Arachis hypogea (Figuur S8, S9). Kortom, NLR-genen vertonen hogere kwantitatieve expressieniveaus in A. monticola en neopolyploïden (figuur S8).
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- EVM Financiën. Uniforme interface voor gedecentraliseerde financiën. Toegang hier.
- Quantum Media Groep. IR/PR versterkt. Toegang hier.
- PlatoAiStream. Web3 gegevensintelligentie. Kennis versterkt. Toegang hier.
- Bron: https://www.nature.com/articles/s41598-023-36302-1