Generatie en karakterisering van patiëntspecifieke hiPSC's en hiPSC-CM's
hiPSC-lijnen werden vastgesteld op basis van twee TOF-DG-patiënten, twee TOF-ND-patiënten en twee gezonde controles met geverifieerde pluripotentiemarkers en kiemlaagmarkers (aanvullende figuren XNUMX). 1 en 2). Sequencing van het hele genoom bevestigde respectievelijk de aanwezigheid en de afwezigheid van 22q11.2-deletie in de hiPSC-lijnen van TOF-DG- en TOF-ND-patiënten (aanvullende figuur XNUMX). 3). Er werden geen missense- of nonsense-mutaties van eerder gerapporteerde CHD-gerelateerde genen gevonden in een van de hiPSC-lijnen. Cardiale differentiatie werd bereikt via de APLNR+ sorteerprotocol ontwikkeld door ons laboratorium32. Alle hiPSC-lijnen vertoonden een bevredigende differentiatie-efficiëntie met meer dan 70% TNNT2+-cellen. Op dag 12 na differentiatie werden hiPSC-CM's in hCAS gefabriceerd en nog eens 10 dagen laten rijpen vóór transcriptomische en functionele beoordelingen.
TOF-DG patiëntspecifieke hiPSC-CM's vertonen een defecte ventriculaire specificatie
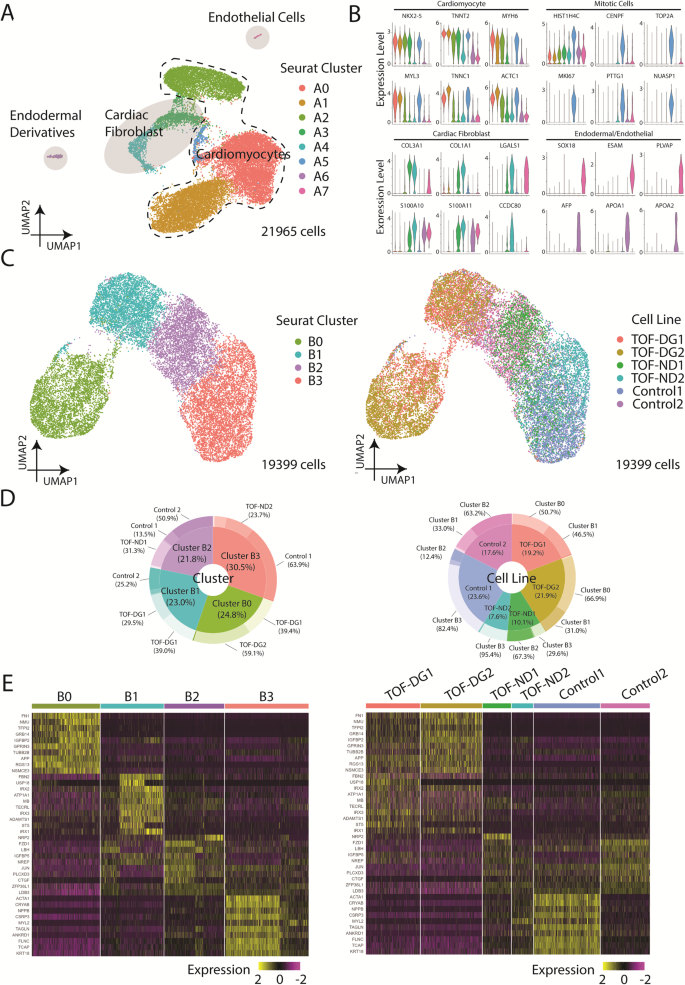
We bepaalden eerst het transcriptomische profiel van gedifferentieerde hiPSC-CM's van patiënten en controles door scRNA-seq met behulp van het 10X Genomics-platform. Clustering van 21,965 cellen met behulp van Uniform Manifold Approximation and Projection (UMAP) identificeerde 7 verschillende Seurat-clusters (Fig. 1A). Seurat-clusters A0, A1, A2, A5 brachten relatief hogere niveaus van hartgenen tot expressie, waaronder NKX2-5, myosine lichte/zware keten en troponine (Fig. 1B), die deze clusters identificeerde als hartspiercellen. Cluster A5 bracht ook hogere niveaus van mitotische genen tot expressie, wat de proliferatieve aard van de hartspiercellen suggereert. Clusters A3 en A4 brachten relatief lagere niveaus van hartgenen tot expressie (NKX2-5, TNNT2) maar verrijkt in de expressie van genen gerelateerd aan remodellering van de extracellulaire matrix (ECM) (Fig. 1B), waarvan het profiel compatibel is met hartfibroblasten. Clusters A6 en A7 werden gedefinieerd door endodermale afstamming (AFP, APOA1, APOA2) en endotheel (FLT1, ESAM, PLVAP) markeringen, respectievelijk (Fig. 1B). Daarom bestond het merendeel van de cellen waarvan de sequentie werd bepaald uit hartspiercellen (86%), sommige uit hartfibroblasten (10%) en zeer weinig uit endodermale (<1%) en endotheliale derivaten (<1%).
A UMAP-presentatie van alle celtypen geïdentificeerd in hCAS (TOF-DG, TOF-ND en controle). Kleuring: Seuratclusters; stippellijn: hartspiercellen; lichtgrijs: niet-myocyten. B Vioolgrafieken van genexpressies van hartspiercellen, mitotische cellen en niet-myocyten: cardiale fibroblasten, endodermale derivaten en endotheelcellen. Kleuring: Seurat-celclusters zoals in (A). C UMAP-presentaties van alle hiPSC-CM's geïdentificeerd in hCAS (TOF-DG, TOF-ND en controle). Gegevens werden gekleurd door respectievelijk Seurat-clusters (linkerpaneel) en cellijn (rechterpaneel). D Donutplots van de composities in respectievelijk elk Seurat-cluster (linkerpaneel) en cellijn (rechterpaneel). Gegevens werden alleen berekend op basis van hiPSC-CM's. E Heatmap-presentaties van de top 10 van opgereguleerde genen die tot expressie worden gebracht in elk hiPSC-CMs-Seurat-cluster. Gegevens werden gegroepeerd per Seurat-cluster (linkerpaneel) en cellijn (rechterpaneel).
Met een focus op het onderzoeken van het cardiale transcriptoom, isoleerden we hiPSC-CM's (clusters A0, A1, A2, A5) en voerden we clustering uit met behulp van UMAP (Fig. 1C). Nieuwe Seurat-clusters (B0, B1, B2, B3) werden geïdentificeerd (Fig. 1C). hiPSC-CM's afgeleid van patiënten en controles waren ongelijk verdeeld over clusters (Fig. 1D). Cluster B0 bestond bijna uitsluitend uit hiPSC-CM's (98%) afkomstig van TOF-DG-patiënten, cluster B1 bestond voornamelijk uit cellen van een controlepersoon en de overige cellen van TOF-DG-patiënten, terwijl clusters B2 en B3 uit hiPSC-CM's bestonden. afgeleid van TOF-ND-patiënten en controles.
Cluster B0 bleek een verhoogde expressie van niet-myocytgenen te hebben, waarbij 4 van de 10 top-opgereguleerde genen zelden (<1%) in andere clusters werden aangetroffen (Fig. 1E). Deze niet-myocytgenen waren niet-endodermaal of endotheelgerelateerd. De afwezigheid van verrijking in genen gerelateerd aan de hermodellering van de extracellulaire matrix maakte ook onderscheid tussen cluster B0-cellen en hartfibroblasten. Cluster B1 is verrijkt met de IRX genfamilie (IRX1, IRX2, IRX3), terwijl cluster B2 werd gemarkeerd door LBH uitdrukking (afb. 1E). De orthologen van IRX genen en LBH Er is gerapporteerd dat ze tot expressie worden gebracht in vroege in vivo gedifferentieerde hartspiercellen van muizen33,34. Cluster B2 vertoonde, vergeleken met cluster B1, een verhoogde expressie van ventriculaire cardiomyocytmarkers (MYH7, NPPB). Cluster B3 vertoonde de hoogste expressie van de ventriculaire cardiomyocytmarker (MYL2) (Fig. 1E). Het bestaan van clusters B1, B2 en B3 suggereerde een progressieve specificatie van hiPSC-CM's in de richting van een ventriculair transcriptomisch profiel.
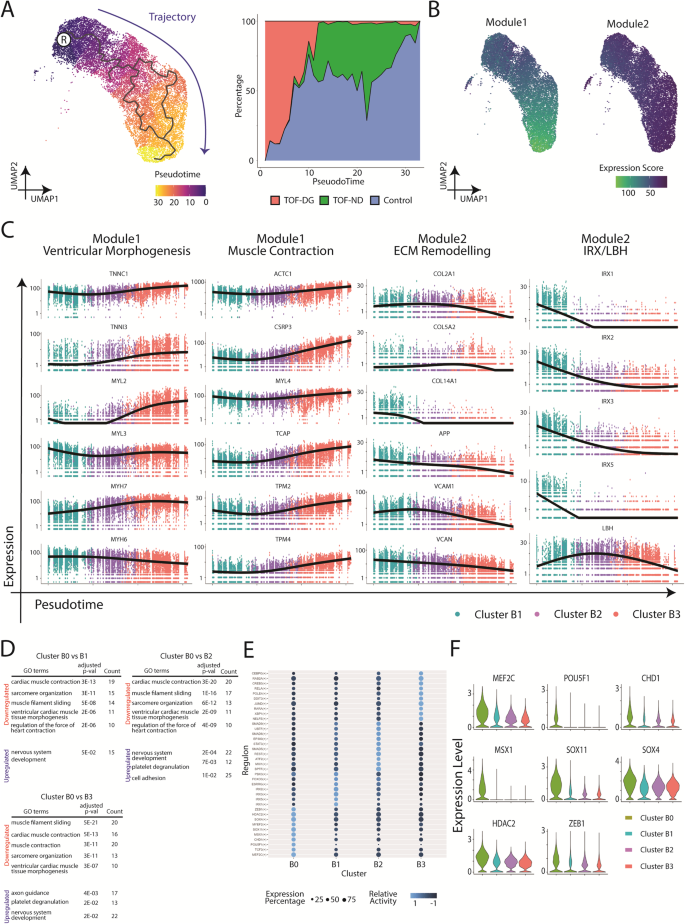
Om de transcriptomische veranderingen tijdens ventriculaire specificatie verder te onderzoeken, hebben we pseudo-tijdanalyse uitgevoerd op clusters B1, B2 en B3. Gebaseerd op de verrijking in de IRX genenfamilie, werd cluster B1 handmatig geselecteerd als het hoofdknooppunt voor de pseudo-tijdanalyse (Fig. 2A). Langs het traject werden TOF-DG-hiPSC-CM's voornamelijk in het begin gedistribueerd, terwijl TOF-ND-hiPSC-CM's voornamelijk in het midden tot het einde werden gedistribueerd. Controle-hiPSC-CM's konden gedurende het hele traject worden gevonden (Fig. 2A).
A Pseudotime-analyse van hCAS-hiPSC-CM's. UMAP-coördinatie en genexpressies van cluster B1, B2 en B3 (Fig. 1C) werden geëxtrapoleerd voor Monocle3-berekening. Het wortelknooppunt werd aangegeven met ®. Traject was blauw gekleurd. B Genmodules dienen als de functie van de pseudo-tijdanalyse en trajectafleiding van hCAS-hiPSC-CM's. C Genexpressiedynamiek langs het traject. Ventriculaire morfogenese, spiercontractie en ECM-remodellering waren de GO-termen verrijkt in de genmodules. X-as: Pseudotijd; Y-as: expressieniveau. ECM extracellulaire matrix. D GO-verrijkingsanalyse van cluster B0 van hCAS-hiPSC-CM's. Er werden GO-termen getoond waarvan werd vastgesteld dat ze verrijkt waren in de gedownreguleerde/opreguleerde genen van cluster B0 (vergeleken met cluster B1/B2/B3). P-waarden werden aangepast voor meerdere Bonferroni-vergelijkingen. E Regulon-analyse van hCAS-hiPSC-CM's. De bovenste regulons (maximaal 10) met hogere relatieve activiteit in elke Seurat-cluster werden weergegeven in de dot-heatmap-plot. F Genexpressie van de bovenste regulons (geïdentificeerd als DEG's) uit cluster B0. Genexpressies werden getoond in de vioolplot.
Genen met vergelijkbare expressiepatronen werden gegroepeerd in modules (Fig. 2B) en onderworpen aan Gene Ontology (GO)-verrijkingsanalyse. Significante GO-termen werden gevonden in zowel Module 1 als 2 (Fig. 2C). Module 1, die hoge expressiescores langs het traject laat zien, was verrijkt met genen gerelateerd aan ventriculaire morfogenese (TNNI3, MYL2, MYH7) en spiercontractie (ACTC1, CSRP3, TCAP). Module 2, die afnemende expressiescores langs het traject laat zien, was verrijkt met genen die verband houden met de hermodellering van extracellulaire matrix (COL2A1, COL5A2, COL14A1). De IRX genenfamilie en LBH werden ook toegewezen aan module 2, waarbij afnemende expressiescores langs het traject werden getoond.
Kortom, pseudo-tijdanalyse op hiPSC-CM's afgeleid van het hCAS-platform identificeerde progressieve ventriculaire specificatie met verhoogde cardiale genexpressie. Controle- en TOF-ND-hiPSC-CM's waren vergelijkbaar in dergelijke progressieve specificaties. De meeste TOF-DG-hiPSC-CM's behielden echter het meer primitieve expressieprofiel.
Een subset van TOF-DG-patiëntspecifieke hiPSC-CM's vertoonde downregulatie van cardiale genexpressie, maar upregulatie van neurale genexpressie
Om de niet-myocytgenexpressie in cluster B0 verder te analyseren, werden differentieel tot expressie gebrachte genen (DEG's) geïdentificeerd door cluster B0 te vergelijken met elk van de andere clusters. Er werden meer dan 200 DEG's, opgereguleerd of gedownreguleerd, geïdentificeerd uit cluster B0 (aanvullende gegevens 1). GO-verrijkingsanalyse identificeerde consistent significante GO-termen die verband houden met hartspiercontractie onder gedownreguleerde genen en die gerelateerd aan neurale ontwikkeling onder opgereguleerde genen in cluster B0 (Fig. 2D). Daarom verschilde cluster B0 van de rest van de clusters door zowel de gedownreguleerde cardiale genexpressie als de opgereguleerde neurale genexpressie. Toch kon geen van de bovengenoemde DEG's, inclusief gedownreguleerde hartgenen en opgereguleerde neurale genen, in kaart worden gebracht in het haploinsufficiënte gebied in beide TOF-DG hiPSC-lijnen.
We hebben verder geprobeerd de overeenkomstige transcriptiefactoren (TF's) en genregulerende netwerken van elk van de clusters te identificeren via Python-implementatie van de Single-Cell regulatoire Network Inference and Clustering (pySCENIC). Van de top 10 TF’s met hogere activiteit in cluster B0 waren er 4 betrokken bij neurale ontwikkeling (TCF3, SOX11, SOX4 en ZEB1) en 2 in hart- en neurale ontwikkeling (MEF2C en MIJNEF2) (Fig. 2E). De expressieniveaus van vijf neurale TF's (SOX11, SOX4, ZEB1, MEF2C en MIJNEF2) werden opgereguleerd in cluster B0 vergeleken met die in andere clusters (Fig. 2F). De bevinding van opgereguleerde expressie van deze neurale TF's was consistent met verhoogde expressie van neurale genen in cluster B0. Net als bij de DEG's werden de bovengenoemde neurale TF's niet in kaart gebracht in het haplo-onvoldoende gebied in beide TOF-DG-cellijnen.
Daarom vertoonde een aanzienlijk deel van de hiPSC-CM's van TOF-DG (cluster B0) downregulatie van cardiale genen en upregulatie van ectopische neurale genen. Het niet in kaart brengen van de DEG's en TF's in cluster B0 naar de 22q11.2-regio suggereerde dat deze transcriptomische signatuur waarschijnlijk een indirect gevolg is van de haplo-insufficiëntie. In tegenstelling tot de hiPSC-CM's van de controle- en de TOF-ND-groepen, die een continuüm in de ventriculaire specificatie vertoonden, vormde cluster B0 een onderscheidend cluster van de rest van de hiPSC-CM's van TOF-DG (cluster B1). De oorsprong van cluster B0 werd verder onderzocht, zoals hieronder beschreven.
Vertakte cardiale differentiatie van TOF-DG cardiale voorlopercellen
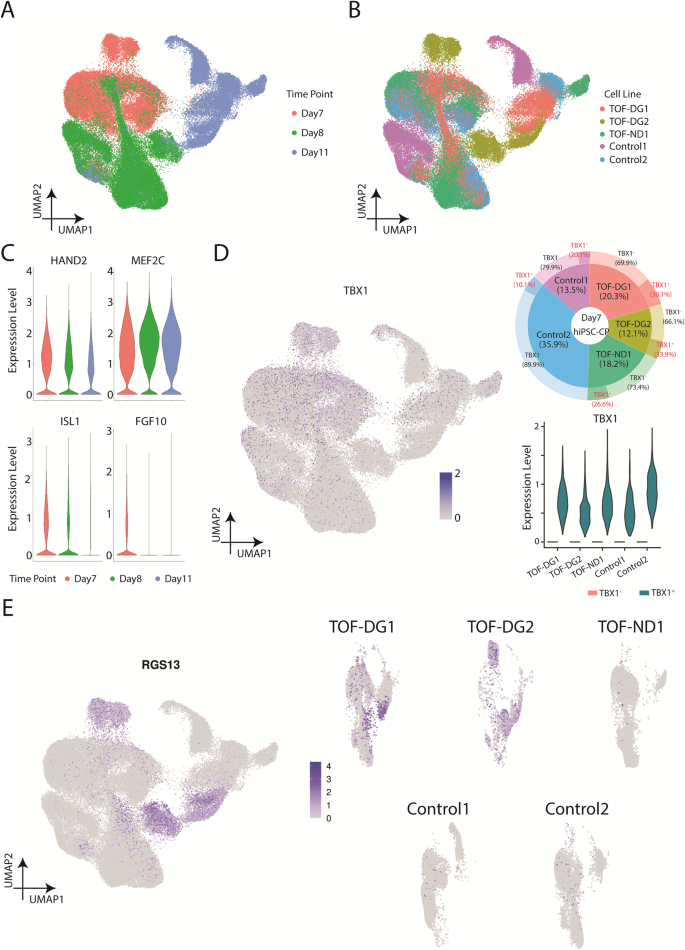
Om de opkomst van TOF-DG-cluster B0 te begrijpen, onderzochten we het transcriptomische profiel van in vitro cardiale voorlopercellen. Gebaseerd op ons eerdere onderzoek32, piekte de genexpressie van het tweede hartveld (SHF) op dag (D) 7 en 8 na differentiatie. Daarom voerden we scRNA-seq uit op hiPSC-cardiale voorlopercellen (hiPSC-CPs) op D7 en D8 en onrijpe hiPSC-CMs op D11 van 2 controles (controle 1 en 2) en 2 TOF-DG (TOF-DG1 en 2) en 1 TOF-ND (TOF-ND1) patiënten (Fig. 3A, B). In totaal werden 87834 cellen behouden na uitsluiting van minder dan 1% van de endodermale derivaten en endotheelcellen. Pseudotime- en GO-verrijkingsanalyse identificeerden upregulatie in cardiale genexpressie en downregulatie in transcriptie en translatie en celcyclusactiviteit langs het traject (aanvullende figuur XNUMX). 4A–D). Vergelijkbaar met ons vorige onderzoek32, SHF-genexpressies, inclusief ISL1, MEF2C, HAND2 en FGF10, werden gevonden in de opeenvolgende tijdstippen (Fig. 3C). TBX1, een van de genen binnen het 22q11.2-microdeletiegebied, is ook een marker van SHF. Onder de opeenvolgende tijdstippen, TBX1 expressie werd gevonden op dag 7, met een variabel aandeel van TBX1+ hiPSC-CP's (10-30%) gevonden uit verschillende lijnen (Fig. 3D). echter, de TBX1 expressieniveau was vergelijkbaar tussen de D7-TBX1+ hiPSC-CP's van verschillende lijnen (Fig. 3D).
A UMAP-presentaties van alle hiPSC-CP's en hiPSC-CM's (TOF-DG, TOF-ND en controle). Gegevens werden gekleurd op basis van een bepaald tijdstip. B UMAP-presentaties van alle hiPSC-CP's en hiPSC-CM's (TOF-DG, TOF-ND en controle). Gegevens werden gekleurd door cellijn. C Vioolgrafieken van SHF-genexpressie, inclusief ISL1, MEF2C, HAND2 en FGF10. D TBX1 expressie in hiPSC-CP's. TBX1 expressie op Dag 7/8/11 in vitro cardiale differentiatie werd getoond in de UMAP-grafiek. Hoogste TBX1 expressie werd gevonden in D7-hiPSC-CP's, en het aandeel van de TBX1+ werd weergegeven in een donutplot en gemarkeerd met een rood label. TBX1 uitdrukking in TBX1+ D7-hiPSC-CP's werden getoond in een vioolplot. E UMAP-percelen van RGS13 expressie op dag 7/8/11 in vitro cardiale differentiatie. RGS13 expressie werden gepresenteerd met alle cellijnen samen (linkerpaneel) en opgesplitst in elke individuele cellijn (rechterpaneel).
Tijdgerelateerde vergelijking met controles identificeerde alleen 20, 10 en 22 DEG's van D7-, D8- en D11-TOF-DG-hiPSC-CP/CM's zonder dat er significante GO-termen werden gevonden. Geen van deze DEG's werd toegewezen aan de 22q11.2 microdeletiegebieden in de twee TOF-DG-hiPSC-lijnen. Ectopische genexpressies van TOF-DG-cluster B0 werden vervolgens onderzocht in de hiPSC-CP's en onvolwassen hiPSC-CM's. In tegenstelling tot TOF-DG-cluster B0 (gedifferentieerde hiPSC-CM's) werden neurale-gerelateerde genen niet gevonden in TOF-DG-hiPSC-CP's (D7 en D8) en D11-onrijpe hiPSC-CM's. Aan de andere kant is een subset van cellen gemarkeerd met RGS13 expressie, 1 van de top 10 opgereguleerde genen in TOF-DG-cluster B0 (Fig. 1E), werd consistent gevonden in TOF-DG-afgeleide cellen op alle gesequentieerde tijdstippen (D7, D8 en D11) (Fig. 3E), maar niet in TOF-ND1 en de controlegroepen.
Ectopische genexpressie is onlangs gerapporteerd in de voorlopercellen van Tbx1 voorwaardelijke nul (Tbx1-cKO) muis35. Om onze hiPSC-CP en onvolwassen hiPSC-CM-gegevens te vergelijken met de Tbx1-cKO muis, hebben we een soortoverschrijdende vergelijking gemaakt met R-pakket SingleCellNet36. Er werden veertien clusters geïdentificeerd uit de Tbx1-cKO-muisgegevensset (aanvullende figuur XNUMX). 5A) met genexpressie gerapporteerd in de publicatie35 (Aanvullende afb. 5B), waaronder vier verschillende cardiale voorlopercellen (multilineage voorlopercellen, MLP; anterieure SHF, aSHF; posterieure SHF, pSHF en proepicardium; PEO) en hartspiercellen. Onze D7-hiPSC-CP's waren grotendeels vergelijkbaar met aSHF, terwijl D11-hiPSC-onrijpe hartspiercellen grotendeels vergelijkbaar waren met hartspiercellen (aanvullende figuur XNUMX). 5C). In de Tbx1-cKO-muisgegevensset, Pax 8, het belangrijkste ectopische gen gerapporteerd door Nomaru et al.35, werd uitsluitend gevonden in MLP en longvoorlopercellen, maar niet in aSHF en hartspiercellen (aanvullende figuur XNUMX). 5D). Wij hebben ons niet geïdentificeerd PAX8 expressie in onze D7-D11-dataset. Ook, RGS13, het ectopische gen gevonden in onze D7-D11-dataset, werd niet geïdentificeerd in de Tbx1-cKO-muis. Met andere woorden, terwijl in beide gevallen ectopische genexpressie werd gevonden Tbx1-cKO-muis en onze DG-hiPSC-CP's/CM's, de identiteit van en de populatie die het ectopische gen tot expressie brengt, zijn verschillend.
Veranderde elektrofysiologische parameters en verhoogde aritmogeniciteit in TOF-DG-hiPSC-CM's
Naast transcriptomische profilering hebben we de elektrofysiologie en aritmogeniciteit van de gedifferentieerde hiPSC-CM's onderzocht met behulp van ons hCAS-platform om een uitgebreidere beoordeling van de hiPSC-CM's te bieden. Elektrofysiologische parameters, waaronder actiepotentiaal (AP), calciumverwerking en effectieve refractaire periode (ERP), werden gemeten onder elektrische stimulatie bij 1 Hz.
Vergeleken met beide controles vertoonde TOF-DG2-hCAS een significante verkorting van de AP-duur (50 en 90% tot repolarisatie, APD50 en APD90) (Fig. 4A). Op vergelijkbare wijze vertoonde TOF-DG2-hCAS, vergeleken met beide controles, een significante verkorting van de AP-opwaartse slagtijd en de tijd tot verval (50 en 90% ten opzichte van de pieken) (Fig. 4A). Daarom verschilden de AP-kenmerken aanzienlijk tussen TOF-DG2 en controles.
A Dotplots van actiepotentiaal (AP)-karakteristiek. APD-actiepotentiaalduur. B Dotplots van calcium-transient (CaT)-karakteristiek. C Dotplot van de effectieve refractaire periode. Gegevens werden gepresenteerd in gemiddelde ± SD voor Fig. 1A-C. Statistische test: gewone eenrichtings-ANOVA gevolgd door de meervoudige vergelijkingstest van Tukey (APD50, AP90%Decay, CaT 50%Decay, CaT Upstroke en effectieve refractaire periode); Kruskal-Wallis-test gevolgd door Dunn's meervoudige vergelijkingstest (APD90, AP opwaartse slag, AP50% Decay en CaT 90% Decay). Significante bevindingen tussen TOF(DG/ND) en controle werden gemarkeerd door sterretjes. * P <0.05; ** P <0.01; *** P < 0.001; **** P <0.0001. D Percentage aritmische herintreding van TOF (DG/ND) en controle-hCAS tijdens steady-state stimulatie (SteadyState) en geprogrammeerde elektrische stimulatie (PES). Statistische test: Fisher's exacte test. *** P <0.001. E Representatieve tracering van actiepotentialen (AP) en isochrone kaart van re-entry-gebeurtenis tijdens PES van TOF-DG2. Het bovenste paneel toonde de representatieve AP-tracering tijdens PES (S1S2), gevolgd door de incidentie van aritmische re-entry-gebeurtenissen. Het onderste paneel toonde twee isochrone kaarten die overeenkomen met een normaal AP (links) en een aritmisch AP (rechts). F Downregulatie van cardiale genen in cluster B0-TOF-DG2. GO-verrijkingsanalyse werd uitgevoerd op de vergelijking tussen cluster B0-TOF-DG2 en cluster B0-TOF-DG1. GO-termen verrijkt met de gedownreguleerde genen worden weergegeven in de tabel (links) en de overeenkomstige genen worden weergegeven in de vioolgrafiek (rechts).
De behandeling van calcium werd beoordeeld met de opwaartse slagtijd (CaT) van calcium en de tijd tot verval (50 en 90% vanaf de piek). Een significante verkorting van de CaT-opwaartse slagtijd werd gevonden in TOF-DG2-hCAS, terwijl een significante verkorting van de tijd tot verval (50% vanaf de piek) werd gevonden in zowel TOF-DG2-hCAS als TOF-ND1-hCAS in vergelijking met beide controles ( Afb. 4B).
We hebben verder de ERP van de hiPSC-CM's beoordeeld, waarvan de verkorting predisponeert voor de ontwikkeling van hartritmestoornissen. Een significante verkorting van EPR werd gevonden in TOF-DG2-hCAS, terwijl verlenging van ERP werd gevonden in TOF-ND2-hCAS (Fig. 4C). Belangrijk is dat TOF-DG2-hCAS een significant hogere incidentie van re-entry-aritmie vertoonde tijdens geprogrammeerde elektrische stimulatie (PES) (Fig. 4D). Een representatieve AP-tracering met de isochrone kaart van herintredende aritmie van TOF-DG2-hCAS wordt getoond in Fig. 4E.
Terwijl TOF-DG2-hCAS een significante verkorting van de AP-duur, CaT-opwaartse slagtijd en tijd tot verval en ERP vertoonde, was TOF-DG1-hCAS vergelijkbaar met controles in termen van deze beoordeelde elektrofysiologische parameters (Fig. 4A–D). We hebben daarom de transcriptomische verschillen tussen TOF-DG1 en TOF-DG2 in cluster B0 verder onderzocht. Vergeleken met cluster B0-TOF-DG1 vertoonde cluster B0-TOF-DG2 downregulatie van hartgenen gerelateerd aan spiercontractie (Fig. 4F). Hoewel deze hartgenen in zowel cluster B0-TOF-DG1 als cluster B0-TOF-DG2 werden gedownreguleerd in vergelijking met andere clusters (B1, B2 en B3) (Fig. 4F), was hun mate van downregulatie prominenter in cluster B0-TOF-DG2 (Fig. 4F). Voorts PKP2Er wordt gerapporteerd dat de mutatie ervan geassocieerd is met aritmogene RV-dysplasie37, bleek een van de gedownreguleerde hartgenen in cluster B0-TOF-DG2 te zijn (Fig. 4F). De variërende mate van downregulatie van cardiale genen kan daarom de functionele discrepantie verklaren die wordt waargenomen tussen cellijnen van verschillende patiënten in het hCAS-platform.
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- PlatoData.Network Verticale generatieve AI. Versterk jezelf. Toegang hier.
- PlatoAiStream. Web3-intelligentie. Kennis versterkt. Toegang hier.
- PlatoESG. carbon, CleanTech, Energie, Milieu, Zonne, Afvalbeheer. Toegang hier.
- Plato Gezondheid. Intelligentie op het gebied van biotech en klinische proeven. Toegang hier.
- Bron: https://www.nature.com/articles/s42003-023-05344-6