컴퓨팅 설계 전략
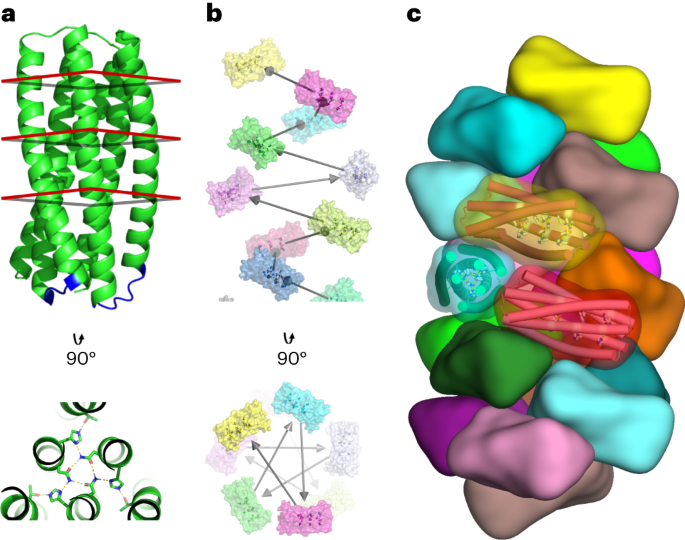
pRO-2.3 나선을 단일 사슬로 연결하는 짧은 루프는 고해상도 결정학 구조에서 DSSP에 의해 식별된 두 개의 나선 영역에 걸쳐 있는 조각으로 구성된 백본 샘플의 철저한 데이터베이스를 사용하여 설계되었습니다(이전에 설명한 대로).14). 루프는 최적화된 중첩 알고리즘을 사용하여 단편과 표적의 말단 잔기의 엄격한 정렬을 통해 이 데이터베이스에서 식별되었습니다.15. 0.35Å RMSD의 정렬 허용 오차를 충족하는 후보는 정렬된 후보 백본 중원자 좌표에 대한 비틀림 공간 좌표 및 소프트 좌표 제약 조건을 통해 대상 백본에 정렬되었습니다. 그런 다음 소스 구조 데이터베이스에 대한 루프 백본의 정렬을 통해 생성된 시퀀스 프로필 제약 조건에 따라 후보 루프 시퀀스를 설계했습니다. 가장 낮은 점수를 받은 후보가 최종 루프 설계를 위해 선택되었습니다.
나선형 도킹 및 설계 방법7 나선형 필라멘트 설계 모델을 생성하기 위해 연결된 pRO-2.3에 적용되었습니다. 다음 기준은 개별 설계 궤적을 필터링했습니다. 결합된(고분자) 상태와 결합되지 않은(단량체) 상태 사이의 로제타 에너지 단위 -15.0을 초과하는 불일치, 700Å을 초과하는 인터페이스 표면적2, 0.62를 초과하는 로제타 모양 상보성 및 5 미만의 충족되지 않은 극성 잔기 수. 이러한 기준을 충족하는 디자인은 인터페이스의 바인딩 상태를 안정화하는 데 기여하지 않는 것으로 간주되는 돌연변이에 대한 단일 지점 복귀를 포함하여 수동으로 개선되었습니다. 각 도킹 구성에 대한 최고 점수 디자인은 실험적 검증을 위해 최종 단백질 세트에 통합되었습니다.
단백질 발현 및 정제
총 18개 디자인의 합성 유전자는 발현에 최적화되었습니다. 대장균 IDT로부터 획득한 후 NdeI 및 XhoI 제한 사이트 사이의 pET29b+ 벡터의 다중 클로닝 사이트에 삽입합니다. 이러한 구성은 BL21*(DE3)에 도입되었습니다. E. 대장균 유능한 세포. 형질전환체는 50 mg l 가 보충된 200 ml Terrific Broth 배지에서 배양되었습니다.- 1 카나마이신. T7 프로모터의 제어 하에 발현은 Studier 자가유도를 사용하여 24°C에서 37시간 동안 진행되었습니다.16 배양물이 원심분리에 의해 수확될 때까지. 세포 펠릿을 트리스 완충 식염수(TBS)에 재현탁시키고 Bugbuster 세제로 용해시켰습니다. 원심분리에 의해 정화된 가용성 분획은 Ni를 통해 정제되었습니다.2+ Ni-NTA Superflow 수지를 사용한 고정화 금속 친화성 크로마토그래피. 세포 용해물이 결합된 수지를 40 컬럼 부피의 500mM 이미다졸 및 400mM NaCl로 세척한 후, 75mM 이미다졸 및 0.5mM NaCl로 용출시켰습니다. 가용성 및 불용성 분획을 SDS-폴리아크릴아미드 겔 전기영동 분석에 적용했습니다. 정확한 분자량의 단백질 밴드를 나타내는 샘플을 전자 현미경 스크리닝을 위해 선택했습니다. 선택된 디자인은 추가 특성화를 위해 24l까지 확장되었으며, Studier 자동 유도를 사용하여 37°C에서 XNUMX시간 동안 발현이 다시 진행되었습니다.16 원심분리로 수확하기 전. 세포 펠릿을 TBS에 재현탁시키고 미세유동화에 의해 용해시킨 다음, 위에서 설명한 대로 정제했습니다.
네거티브 스테인 EM
전자현미경 스크리닝을 위해 가용성 분획을 TBS(25 mM Tris 완충액, 75 mM NaCl, pH 8)에 농축했습니다. 6μl 액적(1μl의 완충액으로 즉시 희석된 5μl 샘플)을 음광 방전 탄소 코팅 200메시 구리 그리드에 적용하고 Milli-Q 물로 세척한 후 0.75% 우라닐 포메이트(pH 4.0)를 사용하여 염색했습니다. ) 또는 이전에 설명한 대로 Nanoprobes, Inc.에서 구입한 Nano-W(pH 6.8)17. 스크리닝은 100kV Morgagni M268 투과 전자 현미경(FEI) 또는 120kV Talos L120C 투과 전자 현미경(ThermoFisher)을 사용하여 수행되었습니다. 하단 마운트 Teitz CMOS 4k 카메라 시스템을 사용하여 이미지를 캡처하고 Fiji 소프트웨어(버전: 2.14.0/1.54f)를 사용하여 향상된 대비를 위해 처리했습니다.18 명확성을 위해.
cryoSPARC의 섬유 추적 알고리즘을 사용하여 섬유 길이를 정량화했습니다.8. 이 방법은 템플릿 클래스와의 상호 상관 관계와 식별된 입자에서 인접한 섬유를 추적하여 섬유를 식별합니다. DpHF19에서 생성된 템플릿 클래스가 측정된 모든 섬유에 사용되었습니다. 섬유는 평균 곡률(<0.0005Å)에 따라 필터링되었습니다.- 1) 및 각 섬유에 대한 평균 정규화 상호 상관(>0.5)입니다. DpHF18의 경우 pH 5, 2, 3, 20, 28 및 21~3에 대해 각각 3.5, 4.2, 5, 8, 3 및 8개의 이미지를 사용했습니다. DpHF19의 경우 pH 7, 8, 8, 28, 4 및 5~3에 대해 각각 3.5, 4.2, 5, 8, 3 및 8 이미지를 사용했습니다. DpHF19_9his의 경우 6, 6, 8, 14, 15, 8 및 4개의 이미지를 각각 pH 3, 3.5, 4.2, 5, 6, 8 및 3~8에 사용했습니다.
극저온-EM
Cryo-EM 샘플은 CFLAT 홀리-탄소 그리드에 단백질을 적용하고, 액체를 닦아내고 Vitrobot(ThermoFisher)을 사용하여 그리드를 액체 에탄에 넣어 준비했습니다. DpHF19의 경우, 픽셀 크기가 픽셀당 2Å, 1.16프레임 및 총 전자 선량인 카운팅 모드에서 작동하는 K-50 Summit Direct Detect 카메라(Gatan Inc.)가 장착된 Glacios 현미경(ThermoFisher)에서 비디오를 수집했습니다. 65Å 중-2. DpHF18 및 DpHF7의 경우 비디오는 초고해상도 모드에서 작동하는 K-2 Summit Direct Detect 카메라(Gatan Inc.)가 장착된 Titan Krios(ThermoFisher)에서 픽셀 크기가 픽셀당 0.525Å, 50프레임 및 총 전자량 90Å-2. Leginon을 사용하여 자동 데이터 수집을 수행했습니다.19 버전 3.4. 데이터 처리는 cryoSPARC를 사용하여 수행되었습니다.8, 워크플로는 보충 그림에 요약되어 있습니다. 10-12. 비디오는 패치 모션 보정을 통해 정렬되었으며, 초고해상도 비디오는 1.05Å의 픽셀 크기로 비닝되었습니다. 대비 전달 함수(CTF) 매개변수는 패치 CTF를 사용하여 추정되었습니다. 이미지의 하위 집합에 대해 템플릿 없는 필라멘트 추적을 수행하고 결과 입자를 2D 분류했습니다. 선택한 2D 클래스는 전체 데이터 세트에서 템플릿 기반 필라멘트 추적을 위한 템플릿으로 사용되었습니다. 여러 라운드의 2D 분류 후에 선택된 입자는 나선형 대칭이 적용되고 불균일 정제가 활성화된 3D 정제를 거쳤습니다. DpHF19의 경우 7개 시작 나선형 대칭 매개변수 대신 개별 비접촉 하위 단위와 관련된 19개 시작 나선형 대칭을 적용했습니다. DpHFXNUMX 및 DpHFXNUMX의 경우 입자별 디포커스, 빔 틸트 및 구면 수차도 개선되었습니다. 밀도 수정은 Phenix의 ResolveCryoEM을 사용하여 수행되었습니다.20,21 버전 phenix-1.20.1. DpHF18 및 DpHF19에 대한 원자 모델은 ISOLDE를 사용하여 저온 EM 맵으로 정제되었습니다.22, 로타머 및 Ramachandran 제한이 비활성화되고 입력 시작 모델에 의해 부과된 참조 제한이 있는 Phenix의 실제 공간 개선이 이어집니다. DpHF7에 대한 모델 설명은 분할된 극저온-EM 비대칭 단위 밀도에 대한 새로운 모델 구축 프로토콜을 사용했습니다.23. 후속 잔류물 통합 및 정제는 RosettaCM을 사용하여 달성되었습니다.24 버전 2019.31, 최적의 밀도 맞춤 및 필라멘트 내 인터페이스를 위해 분할되지 않은 저온 EM 맵 전체에서 대칭을 활용합니다. DpHF18 및 DpHF19에 대해 위에서 설명한 대로 실제 공간 개선의 최종 라운드가 Phenix에서 수행되었습니다. Cryo-EM 데이터 수집, 정제 및 검증 통계는 보충 표에 요약되어 있습니다. 1.
TIRFM
섬유 집합체
pH 반응성 섬유의 시드 핵생성을 이미지화하기 위해 DpHF18 섬유는 두 가지 서로 다른 말레이미드 결합 형광단인 Oregon488 및 sulfo-Cy5로 표지되었습니다. Zeba 스핀 컬럼에서 TBS(10mM Tris, 1mM NaCl, pH 4)로 완충액을 교환하고 25μM로 농축하기 전에 섬유를 실온에서 100시간 동안 PBS + 8.0mM TCEP에서 30x 몰 과량으로 표지했습니다. . 30μM의 녹색 섬유는 1M 구연산염(0.6μl의 섬유에 20μl의 구연산염)을 첨가하여 분해되어 pH를 3.0으로 감소시켰습니다. pH를 5으로 되돌리기 위해 Tris(3.6μl의 1M 스톡)를 첨가하기 전에 용액을 8.0분 동안 배양했습니다. 1μM의 조립된 DpHF18-Cy5 섬유 30μl를 용액에 추가했습니다. 이후 용액을 실온에서 배양한 후 13,000°C에서 원심분리했습니다. g 벤치탑 원심분리기에서 2분 동안. 섬유를 TBS에 재현탁하고 TIRFM으로 이미지화했습니다.
섬유분해
낮은 pH에서 분해되는 섬유의 빠른 TIRFM 이미징은 빠른 초점 시스템과 함께 완벽한 초점 시스템을 갖춘 Nikon Ti 스탠드를 기반으로 맞춤형 TIRF 시스템에서 수행되었습니다. Z 피에조 스테이지(ASI), 맞춤형 확장 시야(Cairn) 및 PLAN Apo 2 NA ×1.45 대물렌즈를 갖춘 방위각 TIRF 조명기(iLas100, Roper France). 방위각 조명과 동기화된 의사 글로벌 셔터 모드에서 실행되는 Photometrics Prime 95B 후면 조명 sCMOS 카메라를 사용하여 이미지를 획득했습니다. 시스템은 Metamorph 7.10.1.161에 의해 운영되었습니다. Sulfo-Cy5 말레이미드 표지 섬유는 630nm 레이저(Cairn 레이저 발사에 장착된 150mW Coherent OBIS)로 이미지화하고 Cairn Optospin 휠에 장착된 Chroma ET655lp 필터를 사용하여 1ms마다 16프레임의 프레임 속도로 이미지화했습니다.
섬유는 클린룸 등급 커버슬립(맞춤형, 25 × 8.0mm)에 장착된 Ibidi 플로우 셀의 이미징 버퍼(100mM Tris pH 25, 75mM NaCl)에서 이미지화되었습니다.2, Nexterion), PLL-PEG (0.1 mg ml )로 부동태화- 1 20 mM Hepes, pH 7.6; 5 분). 결합되지 않은 섬유를 이미징 버퍼로 제거하기 전에 섬유를 커버슬립에 5분 동안 침착시켰습니다. 빠른 획득 동안 낮은 pH 완충액(25mM Tris, 100mM NaCl, pH 3.0)을 유입하여 pH를 감소시켰습니다.
벌크 용액에서 섬유 분해를 측정하기 위해 1.5ml Eppendorf 튜브에 미리 형성된 섬유를 더 낮은 pH의 구연산염 완충액으로 교환하여 분해를 자극했습니다. 각 pH 반응의 일부를 다양한 시점에서 제거하고 96웰 플레이트에 10분 동안 첨가하여 섬유가 침전되어 유리 기판에 부착되도록 했습니다. 각 조건 및 시점에 대해 Nikon ×2500 PLAN Apo 60 NA 공기 대물렌즈와 0.95nm LED 여기 소스, 631ms 노출 시간, 수집된 방출을 사용하여 IN Cell Analyser 150HS 현미경(Molecular Devices)에서 684개 시야를 획득했습니다. 24 ± XNUMX nm 대역 통과 필터를 통해. Otsu 임계값 알고리즘으로 섬유를 분할하기 위해 맞춤형 CellProfiler 스크립트를 사용하여 이미지를 정량화했습니다.25. 배경 신호를 기준으로 섬유가 올바르게 식별될 때까지 임계값의 상한 및 하한과 객체 ID에 대한 적응형 창을 조정했습니다. CellProfiler 파이프라인을 사용하여 식별된 개체의 장축 길이는 각 pH 조건의 배양 시간에 대해 플롯되었습니다.
액상 AFM
샘플 준비
새로 절단된 백운모 표면(10mm, Ted Pella Inc.)에 0.01wt% 폴리 라이신 용액 12μl를 2분 동안 배양했습니다. 과잉 용액을 제거하고 표면을 물로 헹구고 NXNUMX로 건조시켰다.2 가스7. 그런 다음 이미징 버퍼(30mM Tris-HCl, 10mM NaCl, pH 25)에 들어 있는 400μl 8μM 단백질 용액을 폴리 라이신 코팅 운모에서 30분 동안 배양하고 이미지 버퍼로 세척하여 과잉 단백질을 제거했습니다. 분해 완충액(25mM Tris-HCl, 400mM NaCl, pH 4.1, 4.4, 4, 5 또는 4.7)의 pH를 10M NaOH 또는 1M 구연산으로 조정하고 0.1μm 기공 크기의 PVDF 필터로 여과한 후 사용했습니다. . 광산 실험을 위해, 10mM Tris-HCl pH 25에 용해된 8μM 단백질 용액을 운모 위에서 30분간 배양하고 25mM Tris-HCl pH 5.5로 세척했습니다. 표면의 섬유 개수 밀도가 낮은 경우 추가 증착 및 린스 단계를 수행했습니다. 또한 1mM Tris-HCl pH 2에 25mM 5.5-nitrobenzaldehyde(Sigma-Aldrich)를 새롭게 준비하여 어떤 단계에서도 빛에 노출되지 않고 즉시 사용했습니다.26. 분광학 및 pH 측정에 따르면 2-니트로벤즈알데히드는 200~405nm의 파장 사이에서 활성화되고 pH를 5.5에서 2.7로 낮추며, 레이저 강도가 높을수록 소비가 빨라지고 산성화가 빨라지는 것으로 나타났습니다.
영상
일정한 조성에서의 동역학 연구를 위해, 단백질 코팅된 폴리-라이신 운모 기질을 AFM 액체 셀(Bruker Multimode8) 아래에 배치했습니다. 이미지는 깨끗한 질화규소 캔틸레버(Bruker, SNL-10, 스프링 상수: 0.12 N·m)를 사용하여 이미징 버퍼에서 캡처되었습니다.- 1, UV 오존 처리(5분 동안))는 실온(25°C)에서 태핑 모드로 수행됩니다. 분해 버퍼를 흐르게 하기 전에 섬유를 10분 동안 연속적으로 이미지화하여 매개변수(256개 스캔 라인, 1.5Hz 스캔 속도, 높은 적분 이득(3-4) 및 50-100mV 자유 진폭)을 최적화했습니다. 캔틸레버로 인한 손상이 발생하지 않은 것을 확인한 후 분해 완충액을 25 µl min 씩 지속적으로 주입했습니다.- 1. 통과 설정은 무시할 수 있는 체류 시간과 빠른 pH 전환을 제공하도록 최적화되었습니다.10.
광산 연구를 위해 25mM Tris-HCl pH 5.5의 단백질 코팅 운모를 환기 밸브가 있는 BlueDrive 레이저(×0.3 강도 필터, 405nm 파장)가 장착된 Cypher VRS AFM(Asylum Research)의 액체 셀 아래에 배치했습니다. 탭핑 모드로 열리고 작동됩니다. 섬유의 높은 표면 적용 범위를 확인한 후 이미징 버퍼를 1mM Tris-HCl pH 2에서 25mM 5.5-니트로벤즈알데히드로 교체하고 가시적 배경광에 노출시키지 않고 작동하고 다시 이미지화했습니다. 그런 다음 캔틸레버를 집어넣고 BlueDrive를 켜고 AFM의 전동 광학 현미경을 사용하여 미리 선택된 영역에 걸쳐 반복적으로 래스터링했습니다. 스폿 및 라인 패턴에 대한 래스터/드웰 중 총 UV 노출 시간은 10분을 넘지 않았으며, 그 후 캔틸레버를 노출된 영역으로 다시 이동하여 이미지를 촬영했습니다. 전반적인 pH 변화를 위해 광산 용액과 접촉하는 AFM 액체 셀의 석영 창을 휴대용 UV 램프(364 nm 파장)에 7분 동안 노출시킨 후 이미지화했습니다.
이미지는 Gwyddion SPM v2.62 데이터 분석 소프트웨어로 처리되었으며 Fiji 소프트웨어 v1.53s로 분석되었습니다.18. 동역학에서는 전체 섬유 길이를 측정하였고, 이미 분해된 것으로 간주되는 조각은 길이 측정에서 제외하였다. 개별 섬유의 각 끝 부분에서 분해 속도를 측정합니다(보조 그림 2). 8), 섬유의 중심(초기 길이의 절반)을 길이 측정의 두 번째 끝으로 지정하고, 섬유 조각의 경우 조각의 중심을 두 번째 끝으로 측정했습니다.
- SEO 기반 콘텐츠 및 PR 배포. 오늘 증폭하십시오.
- PlatoData.Network 수직 생성 Ai. 자신에게 권한을 부여하십시오. 여기에서 액세스하십시오.
- PlatoAiStream. 웹3 인텔리전스. 지식 증폭. 여기에서 액세스하십시오.
- 플라톤ESG. 탄소, 클린테크, 에너지, 환경, 태양광, 폐기물 관리. 여기에서 액세스하십시오.
- PlatoHealth. 생명 공학 및 임상 시험 인텔리전스. 여기에서 액세스하십시오.
- 출처: https://www.nature.com/articles/s41565-024-01641-1



