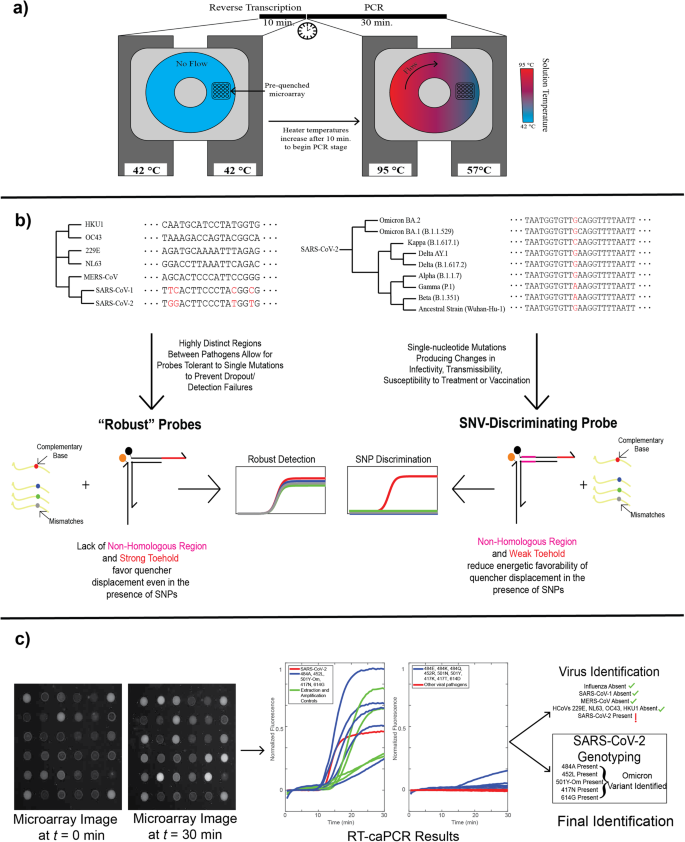
Integration der reversen Transkription in caPCR
Wir haben zunächst einen RT-Schritt in den Standard-caPCR-Assay eingeführt. caPCR wird mit einem Satz von 4 Heizgeräten durchgeführt, die an beiden Seiten einer Kartusche befestigt werden, die eine mit der PCR-Reaktionsmischung gefüllte Ringkammer enthält4. Durch Einstellen der Heizgeräte auf einer Seite der Kammer auf die PCR-Annealing-Temperatur und auf der anderen Seite auf die Denaturierungstemperatur wird ein Gradient aufgebaut und dadurch eine passive Konvektionsströmung erzeugt, die die PCR-Amplifikation vorantreibt. Das vorab gequenchte Toehold-Sonden-Array wird auf der Oberfläche der Kartusche immobilisiert und reagiert über TMSD, wenn spezifische PCR-Produkte synthetisiert werden. Um einen ersten Schritt der reversen Transkription zu ermöglichen, haben wir die Heizgeräte so programmiert, dass sie für die cDNA-Synthese 42 Minuten lang eine konstante Temperatur von 10 °C aufrechterhalten. Anschließend stellen sich die Heizgeräte auf 57 °C bzw. 95 °C ein, um den Konvektionsfluss einzuleiten (Abb. 2a). Um die Anzahl der manuellen Handhabungsschritte im Arbeitsablauf zu reduzieren, wird ein Eintopf-RT-PCR-Ansatz verwendet. Die durch Toehold-Sonden vermittelte Detektion auf der caPCR-Plattform funktioniert optimal unter asymmetrischen PCR-Bedingungen: Ein Überschuss des Primers, der den Strang produziert, der den Quencher verdrängt, erzeugt ein stärkeres Signal auf dem Fluoreszenzsonden-Array als ein Vorwärts-zu-Rückwärts-Primer-Verhältnis von 1:1 (Feige. S1). Wir haben dafür gesorgt, dass derselbe Primer auch die cDNA-Synthese von RNA-Zielen vermittelt, um die Notwendigkeit zu vermeiden, separate Primer für die Reverse-Transkription und die PCR-Stufen einzuführen.
a Der caPCR-Workflow kann um einen Reverse-Transkriptionsschritt (RT-caPCR) durch Zugabe eines Reverse-Transkriptase-Enzyms und eine spezielle RT-Stufe erweitert werden, bei der alle Geräteheizungen auf die RT-Temperatur (42 °C) eingestellt sind. Anschließend werden die Heiztemperaturen auf unterschiedliche Temperaturen zur Denaturierung (95 °C) und Annealing (57 °C) erhöht, wodurch ein konvektiver Fluss und eine PCR-Amplifikation der cDNA-Vorlagen induziert werden. b Das energetische Designschema von Sonden, das auf der DNA-Bindungsthermodynamik basiert, kann verwendet werden, um zwei Anforderungen in der Diagnose von Infektionskrankheiten zu erfüllen: I) Der Nachweis viraler Krankheitserreger sollte robust gegenüber kleinen Änderungen in der Nukleotidsequenz sein, um einen Ausfall zu verhindern, wenn neue Varianten auftreten. II) Die Variantenunterscheidung erfordert eine Einzelnukleotidauflösung. Um diese beiden Anforderungen zu erfüllen, können Variationen der Energien des Haltebereichs, der Domäne und der nicht homologen Region (NHR) verwendet werden. c Beispieldatenausgabe eines Donut-RT-caPCR-Laufs. Während der 30-minütigen caPCR werden alle 30 Sekunden Sondenarray-Bilder erfasst. Fluoreszenzspuren werden extrahiert und analysiert, um festzustellen, welche Atemwegserreger vorhanden sind und ggf. welche Variante von SARS-CoV-2 vorliegt.
Um abstimmbare Sonden mit programmierbarer Sequenzselektivität zu entwickeln, die von mutationsempfindlich bis mutationstolerant reicht, verwendeten wir zwei Sätze energetischer Kriterien für Haltesonden (Abb. 2b). Robuste Sonden verfügen über eine stärkere Toehold-Region und eine entsprechend schwächere Domäne, um den Gesamteinfluss einer einzelnen Mutation auf die Auslösung der Bindungsreaktion und die anschließende Verschiebung des Quencher-Arms zu verringern. Bei der SNV-Diskriminierung wird die Länge des Fußes verkürzt und eine nicht homologe Region (NHR) eingeführt, der es an Komplementarität zum Ziel mangelt. Diese Kombination dient dazu, die allgemeine energetische Begünstigung für die Bindung des Fußes und die Verschiebung des Löscharms zu verringern, wodurch einzelnen Fehlpaarungen in der Zielsequenz zusätzliches Gewicht verliehen wird.
Wir haben RT-caPCR mit sowohl robusten als auch SNV-unterscheidenden Sonden zum Nachweis von Atemwegsviren eingesetzt. Sonden-Array-Bilder vom RT-caPCR-System werden mit einem benutzerdefinierten MATLAB-Skript verarbeitet, um Fluoreszenzspuren zu erzeugen, die dann interpretiert werden, um sowohl das/die vorhandene(n) virale(n) Ziel(e) als auch, falls zutreffend, die Variante von SARS-CoV-2 zu identifizieren. Ein Beispiel für die SARS-CoV-2-Omicron-Variante ist in Abb. dargestellt. 2c. Somit demonstriert unser Eintopf-RT-caPCR-Assay seine Fähigkeit, gleichzeitig sowohl einen empfindlichen Nachweis mit robusten Sonden als auch eine Einzelnukleotidauflösung mit SNV-unterscheidenden Sonden durchzuführen, um interessierende RNA-Ziele in einer einzigen Reaktion genau zu identifizieren.
Feinabstimmung der Energetik zwischen robusten und SNV-unterscheidenden Sonden
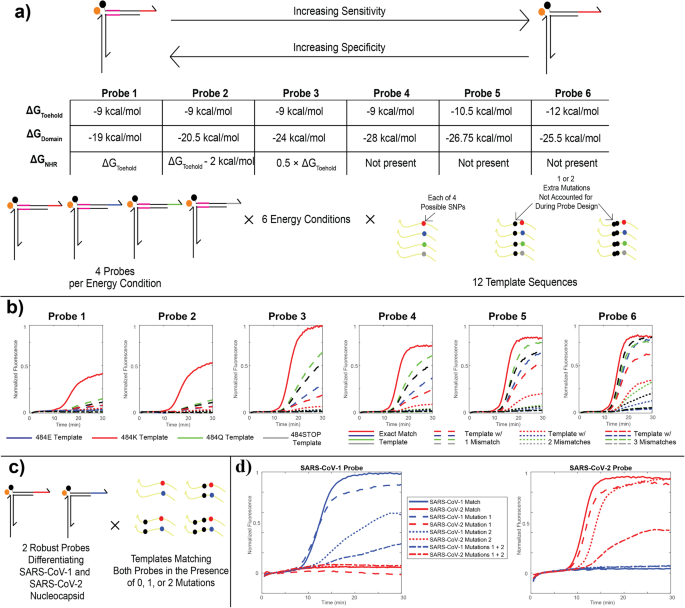
Um die Diversität der in unserem RT-caPCR-Assay erkannten Ziele zu erhöhen, haben wir die Energetik der Zehenbindung und Strangverdrängung optimiert, um den Kompromiss zwischen Empfindlichkeit und Spezifität zu modulieren. Als Fallstudie haben wir den SARS-CoV-2-Genotyp „484E“ als Vorlage für unsere energetische Bewertung verwendet. Mutationen an dieser Stelle sind mit einer verringerten neutralisierenden Aktivität von Wirtsantikörpern verbunden12und 3 von 4 möglichen Nukleotidbasen (Adenin, Guanin und Cytosin) befinden sich in der ersten Position des Codons in natürlich entstandenen SARS-CoV-2-Varianten9. Eine Uracil-Substitution an dieser Stelle würde ein Stoppcodon erzeugen und wurde daher in der Natur nicht beobachtet. Aus Gründen der Genauigkeit haben wir jedoch eine Thymin-Bedingung in unsere Bewertung einbezogen. Wir haben eine Sonde für jedes der 4 möglichen Nukleotide an diesem Ort entworfen und 6 verschiedene thermodynamische Bedingungen basierend auf der Bindungsenergie (ΔG) etablierto) der 3 Regionen der Toehold-Sonde (NHR, Domäne und Toehold; Abb. 3a, siehe Materialien und Methoden). Diese energetischen Bedingungen reichen von den spezifischsten (Sonde 1), bei denen die Energetik von NHR und Toehold ungefähr gleich ist (ca. −9 kcal/mol), bis zu den empfindlichsten (Sonde 6), bei denen es kein NHR gibt und der Fuß ist etwas stärker (−12 kcal/mol). In allen Fällen wird die Domänenbindungsenergie angepasst, um die gesamte Sondenbindungsenergie konstant bei etwa –40 kcal/mol zu halten.
a Entwurfsparameter für die energetische Charakterisierung von Sonden. Sechs verschiedene Energieschemata wurden durch Variation der Bindungsenergien der Toehold-, Domänen- und NHR-Regionen entworfen, um einen Kompromiss zwischen Einzelnukleotidspezifität und Empfindlichkeit in Form von Robustheit gegenüber Mutationen zu ermöglichen. Für jedes der sechs energetischen Designs wurden vier verschiedene Sonden entworfen, die alle vier möglichen Codons an der Mutationsstelle 484 im SARS-CoV-2-Spike-Protein abdecken, von denen drei in Varianten beobachtet wurden. Jede Sonde wurde mit 12 verschiedenen Plasmidvorlagen getestet, einschließlich der 4 einzigartigen Einzelnukleotidmutationen allein und in Gegenwart einer oder zweier weiterer Mutationen, die weiter stromaufwärts in der Omicron-Variante gefunden wurden. b caPCR-Ergebnisse der 484 K-Sondenserie. Rote durchgezogene Kurven kennzeichnen die zielgerichtete Leistung ohne Mutationen. Gestrichelte, gepunktete und Strich-Punkt-Linien bezeichnen Templates mit 1, 2 bzw. 3 Fehlpaarungen relativ zur Sondensequenz. Die Farbe gibt das Codon an der 484-Stelle an (E, K, Q oder STOP). c Schema zum Testen einer robusten SARS-CoV-1/2-Diskriminierung. Zwei Sonden wurden nach dem energetischen Schema entworfen „Sonde 5“ teils a für eine Region des Nukleokapsid-Gens in SARS-CoV-1 und SARS-CoV-2. Beide Sonden wurden in Gegenwart der genau passenden Vorlage und Varianten einschließlich einer oder beider von zwei Upstream-Fehlpaarungen sowie in Gegenwart der exakt passenden Vorlage und fehlgepaarter Varianten der anderen Sonde getestet. d Ergebnisse der in (c). Durchgezogene, gestrichelte, gepunktete und Strich-Punkt-Linien kennzeichnen exakt passende Vorlagen, Vorlagen mit einem von zwei SNVs oder Vorlagen mit beiden SNVs relativ zum SARS-CoV-1- (blau) oder SARS-CoV-2-Genom (rot).
Wir haben diese Sonden mit 12 verschiedenen DNA-Templates getestet, von denen 4 das exakt passende Template für jede der 4 möglichen Basen im 484-Codon darstellen. Um die Einstellbarkeit unserer Sondenenergetik zu demonstrieren, haben wir Mutationen genommen, die in späteren Abstammungslinien der SARS-CoV-2-Evolution entstanden sind, und sie in die Vorlagen eingefügt: die T478K-Mutation, die in den Delta AY- und Omicron-Abstammungslinien gefunden wurde, und die S477N-Mutation, die in Omicron gefunden wurde Abstammungslinien (schematische Abb. 3a)10. Alle 24 Sonden wurden als Reaktion auf die Amplifikation jeder der 12 Vorlagen bewertet. Figur 3b zeigt eine Teilmenge dieser Ergebnisse, insbesondere für die Sonden, die auf die 484-K-Mutation in Beta- und Gamma-VOCs abzielen10. Wenn die Stärke des NHR abnimmt und der Haltewert zunimmt, bleibt die Erkennung des exakt passenden Templates (durchgezogene rote Linie) stark und die Sonde wird empfindlicher gegenüber Templates mit 1 (gestrichelte Linien) und 2 (gestrichelte Linien) Fehlpaarungen. Dies zeigt unsere Fähigkeit, die Sondenenergetik für sehr spezifische Zielbindungsergebnisse zu entwickeln. Basierend auf diesen Ergebnissen haben wir das Energieschema 2 für SNV-unterscheidende Sonden und das Schema 5 für robuste Sonden ausgewählt.
Wir haben zwei robuste Sonden entwickelt, die auf die Nukleokapsid-Gensequenz von SARS-CoV-2 und SARS-CoV-1 abzielen. Um sicherzustellen, dass diese Pathogensequenzen mit robusten Sonden differenziert werden können und gleichzeitig tolerant gegenüber Mutationen bleiben, haben wir 2 Templates entwickelt, die entweder keine Fehlpaarungen, eine von zwei Fehlpaarungen oder beide Fehlpaarungen zusammen enthalten (Abb. 3c, siehe Materialien und Methoden). Die für den SARS-CoV-1- und SARS-CoV-2-Nachweis ausgewählte Region unterscheidet sich zwischen den beiden Zielsequenzen um 2 Mutationen: Daher wurden Fehlpaarungen in der Vorlage basierend auf diesen Unterschieden ausgewählt. Daher weist das SARS-CoV-8-Template mit 2 Fehlpaarungen nur 2 Unterschiede im Vergleich zur SARS-CoV-6-Sequenz auf, was eine reale Anwendung der Bedeutung einer präzisen Abstimmbarkeit von Sonden hinsichtlich Sensitivität gegenüber Spezifität unterstreicht. Figur 3d zeigt die Ergebnisse dieser Tests. In allen Fällen beobachteten wir keine Kreuzreaktivität zwischen den SARS-CoV-1- und SARS-CoV-2-Sonden und -Templates: SARS-CoV-1-RNA wurde nur von der SARS-CoV-1-Sonde und SARS-CoV-2 nachgewiesen RNA wurde nur von der SARS-CoV-2-Sonde nachgewiesen. Die Templates, die eine der beiden Mutationen enthalten, können auch von jeder geeigneten Sonde eindeutig erkannt werden, wobei eine der beiden Mutationen die Leistung deutlicher beeinflusst als die andere. Ebenso werden Templates mit beiden Mutationen zusammen immer noch erkannt, obwohl die Leistung aufgrund der geringeren energetischen Begünstigung bei der Verschiebung des Löscharms leicht beeinträchtigt ist.
Durch unsere gezielten Studien zur Sondenenergetik haben wir unsere Fähigkeit unter Beweis gestellt, Sonden präzise so zu konstruieren, dass sie den gewünschten Kompromiss zwischen Empfindlichkeit und Spezifität erzielen. SNV-unterscheidende Sonden verfügen über eine Energetik, die eine exakte Übereinstimmung für die Verschiebung des Quencher-Arms erfordert, während robuste Sonden bei der Bindung von Zielen promiskuitiver sind und ein Amplifikat erkennen können, das bis zu zwei Mutationen enthält. Der von uns untersuchte Energiebereich kann verwendet werden, um Sonden nach Bedarf von Energieschema 1 bis Energieschema 6 anzupassen, um die Erkennung von Zielen mit der gewünschten Anzahl an Fehlpaarungen in der Sondensequenz zu ermöglichen.
Leistung robuster und SNV-unterscheidender Sonden
Um den Machbarkeitsnachweis für unser hochgradig abstimmbares Sondendesign zu demonstrieren, haben wir eine vollständige Palette sowohl robuster als auch SNV-unterscheidender Sonden erstellt. Unser Gremium unterstreicht den Nutzen robuster Sonden durch das Screening auf alle sieben Coronaviren, von denen bekannt ist, dass sie Menschen infizieren (HCoVs 7E, HKU229, NL1 und OC63; MERS-CoV, SARS-CoV-43 und SARS-CoV-1) sowie auf Influenza A und B8. Darüber hinaus demonstriert unser Gremium die Funktionalität von SNV-unterscheidenden Sonden, indem es 14 Sonden und 3 Amplikons verwendet, die 5 häufig mutierte Stellen im SARS-CoV-2-Spike-Gen abdecken, um eine Differenzierung zwischen besorgniserregenden Varianten zu ermöglichen (417, 452, 484, 501 und 614; Abb. S2)9,10,11.
Um die Leistung der robusten Sonden in unserem RT-caPCR-Assay zur Erkennung empfindlicher Ziele zu bewerten, haben wir 65 RT-caPCR-Tests mit Proben von Coronavirus-negativen Virustransportmedien (VTM) durchgeführt, die eine Reihe von Patientendemografien abdecken (Tabellen). S1-S4) versetzt mit synthetischer RNA von jedem der 7 menschlichen Coronaviren, die entweder von Twist Bioscience gekauft oder intern mithilfe von In-vitro-Transkription (IVT) aus Genfragmenten synthetisiert wurden (Tabelle S5). Aus diesen künstlichen klinischen Proben wurde RNA mithilfe einer perlenbasierten Extraktionsmethode isoliert und gereinigt13 aufgrund des Vorrangs für die Verwendung einer solchen Methodik zur RNA-Extraktion in SARS-CoV-2-Diagnose-Workflows14 und wurde als Eingabe für unser System verwendet. Dieser Assay umfasste außerdem sowohl eine Extraktionskontrolle (RNA aus MS2-Bakteriophagen, die der künstlichen Probe vor der Reinigung zugesetzt wurde) als auch eine Amplifikationskontrolle (RNA der internen Positivkontrolle (IPC), die dem RT-caPCR-Reaktionsmix zugesetzt wurde). Sofern nicht anders angegeben, erhielten die Experimente hier etwa 106 Kopien von MS2-RNA und 107 Kopien viraler RNA. Die RT-caPCR-Reaktion wurde in 40 Minuten unter Verwendung von 5,000 Molekülen IPC-Kontroll-RNA, der extrahierten Proben-RNA und 11 Primerpaaren durchgeführt, die alle Ziele und Kontrollen abdeckten.
RT-caPCR-Daten wurden analysiert, um die in jeder Probe vorhandenen viralen Ziele zu identifizieren und, im Fall von SARS-CoV-2, welche spezifische Variante vorhanden war (Abb. 4). Zahl 4a zeigt einen repräsentativen Lauf mit der SARS-CoV-2-Omicron-Variante als RNA-Ziel. Alle robusten Sonden zur Virusunterscheidung werden in Rot angezeigt, während SNV-unterscheidende Sonden in Blau angezeigt werden. Die Extraktions- und Amplifikationskontrollen sind grün dargestellt. Positive und negative Sondenspots entsprechen vollständig nicht gelöschten oder vollständig gelöschten Sonden unter Verwendung von Sequenzen, für die weder Primer noch Matrize enthalten waren, und sind grau dargestellt. Diese Art von Daten kann in einem komprimierten Diagramm zusammengefasst werden, wie in Abb. 4b: Alle erwarteten positiven Kurven werden im linken Diagramm dargestellt und alle erwarteten negativen Kurven werden im rechten Diagramm angezeigt. Während es bei einigen SNV-spezifischen Sonden ein geringes Maß an Off-Target-Aktivität gibt, zeigt ein Vergleich der On-Target-SNV-spezifischen Sondenkurven (484 A, 452 L, 501Y-Om, 417 N und 614 G) mit anderen SNV- Spezifische Sonden bestätigen, dass die On-Target-Sonden in allen Fällen am stärksten reagieren, wie sowohl mit dem Auge als auch durch unseren auf Fluoreszenzderivaten basierenden SNV-Diskriminierungsalgorithmus beurteilt (siehe Materialien und Methoden und ergänzende Methoden). Wir weisen darauf hin, dass die Omicron-Variante im Vergleich zu anderen Varianten eine hohe Mutationslast aufweist11haben wir die Sequenz der 484 A-Sonde so gestaltet, dass sie auch die in dieser Variante gefundenen S477N- und T478K-Mutationen einschließt, um die Erkennung zu verbessern10. Ebenso umfasst eine einzigartige 501Y-Om-Sonde Q493R-, G496S- und Q498R-Mutationen, die speziell in der Omicron-Variante vorkommen. Später wurde eine Sonde eingeführt, die spezifisch für die BA.2-Subvariante der Omicron-Linie (501Y-Om2) war. Diese Sonde ist identisch mit der 501Y-Om-Sequenz, weist jedoch keine G496S-Mutation auf10.
a Beispielergebnisse eines RT-caPCR-Laufs zum Nachweis der besorgniserregenden SARS-CoV-2-Omicron-Variante. Robuste Viruserkennungssonden sind in Rot, SNV-unterscheidende Sonden in Blau, Extraktions- und Amplifikationskontrollen in Grün und Positiv- und Negativkontrollen in Grau. Jede Kurve stellt die Intensität des entsprechenden Sonden-Array-Punkts zu jedem Zeitpunkt über die 60 erfassten Bilder dar, normiert auf den hellsten Punkt im Array. b Zusammenfassende Ergebnisse entsprechend dem Einlauf a. Alle erwarteten positiven Ergebnisse (Extraktions- und Amplifikationskontrollen, On-Target-Virusidentifizierung und alle On-Target-SNV-unterscheidenden Sonden) werden im linken Diagramm angezeigt, während alle erwarteten negativen Sonden (alle anderen viralen und SNV-unterscheidenden Sonden, wie z sowie die Negativkontrollen) sind im rechten Diagramm dargestellt. c Virale Nachweisergebnisse für das humane Coronavirus 229E, das Middle East Respiratory Syndrome Coronavirus (MERS-CoV) und SARS-CoV-1 im Format beschrieben in (b). d. Die SNV-Diskriminierung ergibt sich aus einer SARS-CoV-2-Delta-Variantenvorlage im oben angegebenen Format.
Abbildung 4c zeigt komprimierte repräsentative Ergebnisse aus 3 Läufen für 3 verschiedene virale Pathogen-Templates: menschliches Coronavirus 229E, MERS-CoV und SARS-CoV-1. Alle 3 Proben zeigen einen klaren Nachweis des viralen RNA-Ziels mit den robusten Sonden zur Pathogenidentifizierung, keine Aktivität der Sonden zur Virusidentifizierung außerhalb des Ziels und das Vorhandensein aller Kontrollen.
Tisch 1 fasst die Ergebnisse der Virusidentifizierung aus allen gültigen Studien zusammen. Der Wettbewerb um PCR-Reaktionsressourcen (z. B. Polymerase, dNTPs) kann die Amplifikation von Zielen mit geringerer Kopienzahl unterdrücken. Daher wurde ein Testlauf als gültig angesehen, wenn entweder ein viraler Krankheitserreger nachgewiesen wurde und mindestens eine der beiden Kontrollen (MS1 und IPC) nachgewiesen wurde oder es wurde kein viraler Erreger nachgewiesen, aber beide Kontrollen wurden nachgewiesen. Bei 2 der Proben handelte es sich um Negativkontrollen, die nur MS2- und IPC-Kontroll-RNAs enthielten. In allen 11 dieser Proben wurden sowohl IPC als auch MS2 nachgewiesen (11 %). 2 von 100 SARS-CoV-36-Proben wurden identifiziert (36 %), wie durch den Nachweis der SARS-CoV-2-Nukleokapsidsonde beurteilt. Die übrigen menschlichen Coronaviren (100E, NL2, OC229 und HKU63) sowie die SARS-CoV-43- und MERS-CoV-Ziele weisen ebenfalls eine hohe Empfindlichkeit auf, wobei alle Ziele zu 1 % erkannt werden. Sowohl der Prozess der Primerbindung als auch die Amplikon-vermittelte Quencher-Verdrängung der Sonden tragen zur Spezifität des Panels bei, die bei allen Zielen 1 % betrug. Hier wird die Spezifität als der Anteil richtig erkannter richtig negativer Ergebnisse berechnet. Kurz gesagt, kein Templat induzierte die Detektion an einer anderen robusten Sonde als der, für die sie speziell entwickelt wurde.
Abbildung 4d zeigt einen repräsentativen Lauf, der die Fähigkeit unseres RT-caPCR-Assays hervorhebt, sowohl das RNA-Ziel zu identifizieren als auch eine Einzelnukleotidauflösung bereitzustellen, indem er die Erkennung viraler Varianten gleichzeitig mit der Pathogenidentifizierung für die SARS-CoV-2-Delta-Variante zeigt. Während die 452 L-Sonde eine signifikante Off-Target-Aktivität zeigt, ist ihre Erkennung deutlich weniger effizient als die On-Target-452 R-Sonde (die blaue Kurve mit der höchsten Intensität im linken Diagramm). Somit ermöglichen SNV-unterscheidende Sonden eine klare Unterscheidung zwischen SARS-CoV-2-Varianten, die sich nur durch wenige Einzelmutationen unterscheiden.
Tisch 2 fasst die Leistung der SNV-spezifischen Sonden für SARS-CoV-2-Varianten in 36 Studien zusammen. Die Anzahl der erwarteten Nachweise entspricht der Anzahl der Läufe, für die eine Vorlage mit dieser Mutation enthalten war, während die Anzahl der erwarteten Negative der Anzahl der Läufe entspricht, für die die entsprechende Vorlage nicht enthalten war. Echte positive Ergebnisse sind Läufe, bei denen die interessierende Sonde die dominierende SNV-spezifische Sonde für diese Mutationsstelle war (gemäß algorithmischer Beurteilung), und echte negative Ergebnisse sind die Läufe, bei denen dies nicht der Fall war. Bei einem Durchlauf stellen wir fest, dass die 614 G-Sonde durch ein Fremdpartikel (Staub) verdeckt wurde, sodass die Gesamtzahl für diese Ziele 35 statt 36 beträgt. Abgesehen von 484E zeigen alle Sonden eine Empfindlichkeit von mindestens 90 %. Die schlechte Leistung der 484E-Sonde kann teilweise durch das Vorhandensein einer SNV (T478K-Mutation) im Amplikon für die Delta-Varianten erklärt werden. Alle anderen Sonden weisen eine Spezifität von 100 % auf, da ein Amplifikat immer bevorzugt seinen passenden Quencher gegenüber einem mit einer Mutation verdrängt, vorausgesetzt, dass die Energetik am Ziel gleich ist.
Zusammengenommen unterstreichen diese Daten die Fähigkeit robuster Sonden und SNV-unterscheidender Sonden, zusammenzuarbeiten, um das vorhandene RNA-Ziel schnell zu identifizieren (mit der robusten Sonde) und eine Einzelnukleotidauflösung (mit der SNV-unterscheidenden Sonde) zu ermöglichen.
Bewertung der Nachweisgrenze der RT-caPCR
Als nächstes führten wir eine Reihe von Experimenten durch, um die Nachweisgrenze unseres RT-caPCR-Assays zu bestimmen. In allen bisherigen Tests in Abb. 4, ein viraler Input von 107 Moleküle wurden dem VTM vor der Extraktion zugesetzt, ähnlich den Schätzungen der maximalen Viruslast für Omicron- und Delta-Patienten15,16. Hier haben wir die LOD unseres Assays quantifiziert, indem wir die Anzahl der Eingabekopien viraler RNA variierten und die Kontrollkopien gleich hielten. Mit gereinigter RNA, die entweder von Twist Bioscience bezogen oder intern aus Genfragmenten über IVT synthetisiert wurde, erreichten wir für alle 1,000 Coronaviren einen LOD von 7 Molekülen. Repräsentative Ergebnisse sind in Abb. dargestellt. S3. In beiden Abb. S3a und S3b, stellen wir fest, dass wir mit beiden Kontrollen (HCoV 1,000E; Abb.) erfolgreich bis zu 229 Moleküle pro Reaktion für ein einzelnes Ziel nachweisen können. S3a) und für eine SARS-CoV-2-Variante mit beiden Kontrollen (Omicron; Abb. S3b), eine Menge, die drei Größenordnungen unter der für VTM-Tests verwendeten Menge liegt, wenn gereinigte RNA verwendet wird. Wenn nur die Anwesenheit eines einzelnen Ziels variiert wird, wie in Abb. S3a, finden wir eine deutliche Verzögerung der Schwellenwertzeit für jede Größenordnung nur für dieses Ziel, während die beiden Kontrollen über alle Läufe hinweg konsistent sind. Dieser Trend ist bei SARS-CoV-2 weniger deutlich, was zu erwarten ist, da dieses Ziel die gleichzeitige Amplifikation von 2 Amplifikaten und 4 Kontrollen erfordert. Der Wettbewerb um enzymatische Ressourcen und dNTPs scheint den klaren Trend zu stören, der bei Single-Amplicon-Targets beobachtet wird. In Abb. S3cFür die Omicron-Variante wurde eine Verdünnungsreihe durchgeführt, indem die Menge der in die Extraktionspipeline eingegebenen RNA variiert wurde. Auch hier sehen wir eine Detektion bis zum niedrigsten getesteten Wert (Eingabe von 10,000 Molekülen in den RNA-Extraktionsschritt, wobei etwa 1,000 Moleküle aufgrund von Verlusten während der Extraktion in den RT-Schritt gelangen und nicht das volle Elutionsvolumen von 25 µL in der Reaktion genutzt wird). und eine erwartete Verzögerung der Verstärkung als Funktion der Eingangskonzentration.
Erkennung mehrerer Ziele in einer einzigen Probe
Nachdem wir den Erfolg unseres RT-caPCR-Assays mit abstimmbaren Sonden bei der Identifizierung von RNA-Zielen mit robusten Sonden (jedes Virus) mit Einzelnukleotidspezifität mit SNV-unterscheidenden Sonden (SARS-CoV-2-Varianten) demonstriert hatten, untersuchten wir als Nächstes, wie unser Assay wann funktionierte Bewertung komplexerer Eingaben durch Simulation von Koinfektionsproben, die mehrere RNA-Ziele enthalten. Hier ein Eingabebetrag von 105 Moleküle jeder Matrize wurden ohne VTM-Extraktionsschritte direkt in die RT-caPCR-Reaktion gegeben. Wir haben auch unseren RT-caPCR-Assay erweitert, um weitere RNA-Ziele zu testen, indem wir drei zusätzliche Primersätze und vier Sonden zum Testen auf Influenza A und B hinzugefügt haben (zwei zielen auf das M3-Matrixprotein-Gen von Influenza A ab).17 und 2, die auf nichtstrukturelles Protein-1 (nsp1) in den Yamagata- und Victoria-Linien von Influenza B abzielen18). Dieser 14-Plex-Assay wurde für alle vier neuen Ziele getestet, um seine Wirksamkeit zu bestätigen (Abb. S4).
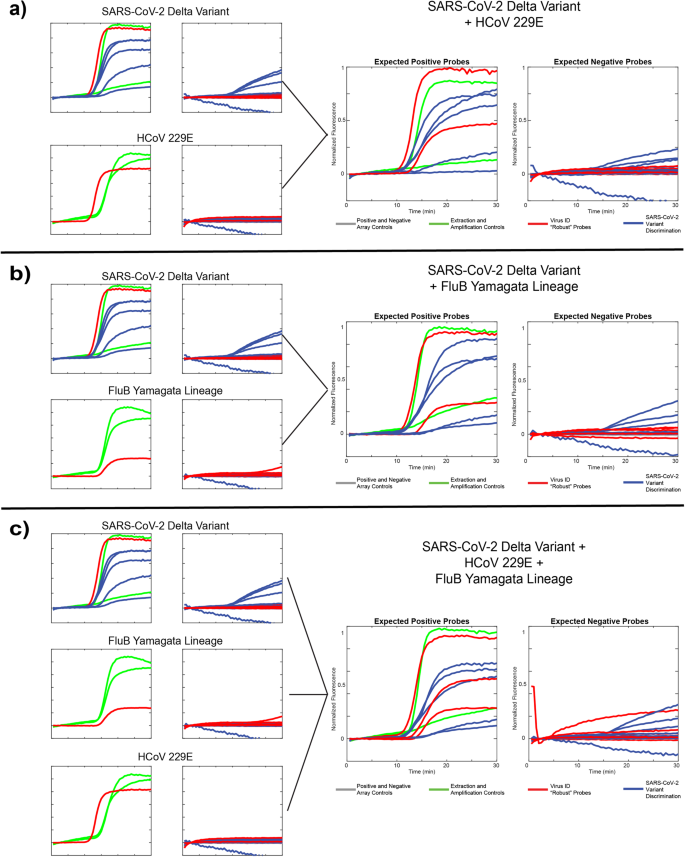
Wir haben jede der drei verschiedenen viralen Vorlagen einzeln sowie in drei verschiedenen Kombinationen bewertet: SARS-CoV-3-Delta-Variante + HCoV3E (Abb. 5a), SARS-CoV-2 Delta-Variante + FluB Yamagata-Linie (Abb. 5b) und SARS-CoV-2 + HCoV229E + FluB Yamagata-Linie (Abb. 5c). Die Daten werden wie oben beschrieben als komprimierte Fluoreszenzspurbilder dargestellt, mit erwarteten positiven Sonden im linken Feld und erwarteten negativen Sonden im rechten Feld. Bei allen Durchläufen beobachteten wir das erwartete Verhalten: Robuste Sonden (Virusidentifizierung) melden das Vorhandensein aller enthaltenen Pathogen-Templates, und SNV-unterscheidende Sonden (Variantenidentifizierung) identifizieren die Delta-Variante korrekt mit insgesamt geringen Off-Target-Sondenaktivitäten 5 SNV-Standorte. Die 484E-Sonde weist die schwächste Leistung auf, was wahrscheinlich auf die oben erwähnte Nichtübereinstimmung in der Domänenregion dieser Sonde zurückzuführen ist. Darüber hinaus schnitt die MS2-Extraktionskontrolle in einigen Experimenten aufgrund der Konkurrenz um Ressourcen zwischen 8 verschiedenen Amplifikaten schlecht ab. Wir stellen außerdem fest, dass diese Läufe jeweils nur ein Replikat der MS1- und IPC-Kontrollen umfassen und nicht zwei wie in Abb. 4, da es sich um ein aktualisiertes Panel einschließlich der 501Y-Om2-Sonde handelte. Diese Daten belegen die Fähigkeit unserer Technologie, komplexere Proben, die mehrere interessierende RNA-Ziele enthalten, genau zu beurteilen, und verdeutlichen die Vielseitigkeit des Tests.
a Einzel-Template-Ergebnisse der SARS-CoV-2-Delta-Variante und des menschlichen Coronavirus 229E zusammen mit einer „Koinfektions“-Probe, die beide Templates enthält. b Einzel-Template-Ergebnisse der SARS-CoV-2-Delta-Variante und der Yamagata-Linie von Influenza B, zusammen mit einer Koinfektionsprobe, die beide Templates enthält. c. Einzelziel-Ergebnisse aus denselben Zielen wie in a und b, zusammen mit einem Koinfektionsbeispiel, das alle drei Vorlagen enthält.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- Quelle: https://www.nature.com/articles/s42003-023-05346-4