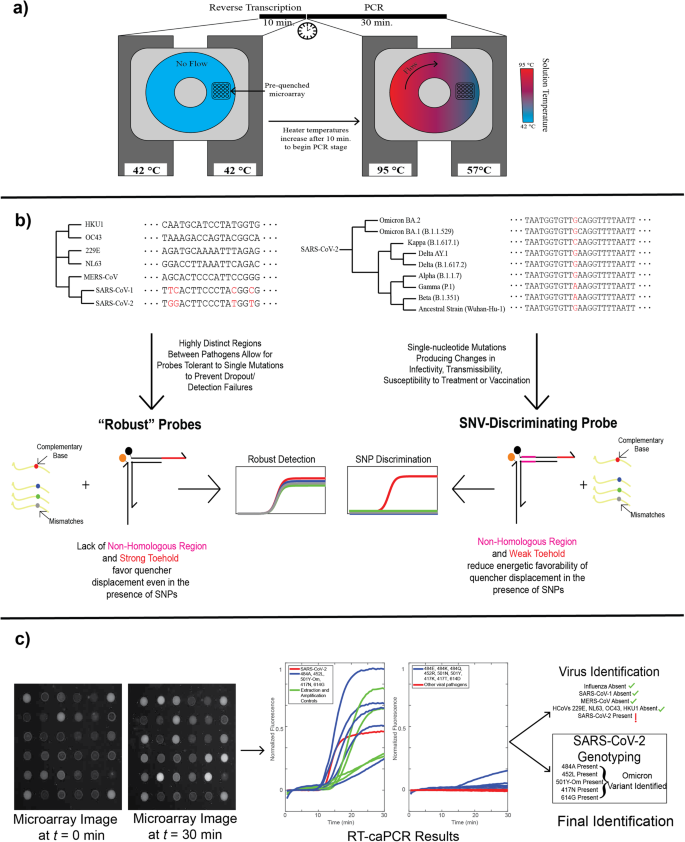
CaPCR'ye ters transkripsiyonun entegre edilmesi
İlk olarak standart caPCR tahliline bir RT adımı ekledik. caPCR, PCR reaksiyon karışımıyla doldurulmuş toroidal bir hazne içeren bir kartuşun her iki yüzüne kenetlenen 4 ısıtıcıdan oluşan bir set kullanılarak gerçekleştirilir.4. Odanın bir tarafındaki ısıtıcıların PCR tavlama sıcaklığına ve diğer tarafındaki denatürasyon sıcaklığına ayarlanmasıyla bir gradyan oluşturulur ve böylece PCR amplifikasyonunu yönlendiren pasif bir konvektif akış üretilir. Önceden söndürülmüş ayak ucu prob dizisi, kartuşun yüzeyinde hareketsiz hale getirilir ve spesifik PCR ürünleri sentezlenirken TMSD yoluyla yanıt verir. Başlangıçtaki ters transkripsiyon adımına uyum sağlamak için, ısıtıcıları cDNA sentezi için 42 dakika boyunca 10 °C'lik sabit bir sıcaklığı koruyacak şekilde programladık, ardından ısıtıcılar konvektif akışı başlatmak için sırasıyla 57 °C ve 95 °C'ye ayarlandı (Şekil XNUMX). 2a). İş akışındaki manuel işlem adımlarının sayısını azaltmak için tek kap RT-PCR yaklaşımı kullanılır. caPCR platformunda ayak ucu prob aracılı tespit, asimetrik PCR koşulları altında optimum şekilde çalışır: söndürücünün yerini alan ipliği üreten primerin fazlalığı, floresan prob dizisinde 1:1 ileri-geri primer oranından daha güçlü bir sinyal üretir (İncir. S1). Ters transkripsiyon ve PCR aşamaları için ayrı primerlerin tanıtılması ihtiyacını ortadan kaldırmak amacıyla aynı primerin aynı zamanda RNA hedeflerinden cDNA sentezine aracılık etmesini de sağladık.
a CaPCR iş akışı, ters transkriptaz enziminin eklenmesi yoluyla bir ters transkripsiyon adımı (RT-caPCR) ve tüm cihaz ısıtıcılarının oda sıcaklığına (42 °C) ayarlandığı özel bir oda sıcaklığı aşamasını içerecek şekilde genişletilebilir. Isıtıcı sıcaklıkları daha sonra denatürasyon (95 °C) ve tavlama (57 °C) için farklı sıcaklıklara yükseltilir ve cDNA şablonlarının konvektif akışını ve PCR amplifikasyonunu indükler. b DNA bağlanma termodinamiğine dayanan probların enerjik tasarım şeması, bulaşıcı hastalık teşhisinde iki ihtiyacı karşılamak için kullanılabilir: I) Viral patojen tespiti, yeni varyantların ortaya çıkması durumunda bırakmayı önlemek için nükleotit sekansındaki küçük değişikliklere karşı dayanıklı olmalıdır. II) Varyant ayrımcılığı tek nükleotid çözünürlüğünü gerektirir. Ayak parmağı, etki alanı ve homolog olmayan bölge (NHR) enerjilerinin değişimi, bu gereksinimlerin her ikisini de karşılamak için kullanılabilir. c Donut RT-caPCR çalıştırmasından örnek veri çıkışı. Prob dizisi görüntüleri, 30 dakikalık caPCR için her 30 saniyede bir alınır. Hangi solunum yolu patojenlerinin mevcut olduğunu ve mümkünse SARS-CoV-2'nin hangi varyantının mevcut olduğunu belirlemek için floresans izleri çıkarılır ve analiz edilir.
Mutasyona duyarlıdan mutasyona toleranslıya kadar programlanabilir dizi seçiciliğine sahip ayarlanabilir problar geliştirmek için, parmak probları için 2 set enerjik kriter kullandık (Şekil XNUMX). 2b). Sağlam problar, tek bir mutasyonun bağlanma reaksiyonunun başlatılması ve ardından söndürücü kolun yer değiştirmesi üzerindeki genel etkisini azaltmak için daha güçlü bir tutma bölgesine ve buna karşılık daha zayıf bir alana sahiptir. SNV ayrımcılığı için ayak parmağının uzunluğu azaltılır ve hedefe tamamlayıcılığı olmayan homolog olmayan bir bölge (NHR) eklenir. Bu kombinasyon, ayakucu bağlama ve söndürücü kolun yer değiştirmesi için genel enerji tercihliliğini azaltmaya hizmet eder, böylece hedef dizideki tekli uyumsuzluklara ekstra ağırlık sağlar.
Solunum yolu virüs tespitine hem sağlam hem de SNV ayırt edici problarla RT-caPCR uyguladık. RT-caPCR sisteminden gelen prob dizisi görüntüleri, floresans izleri üretmek için özel bir MATLAB komut dosyası kullanılarak işlenir ve bunlar daha sonra hem mevcut viral hedef(ler)i hem de uygunsa SARS-CoV-2 varyantını tanımlamak için yorumlanır. Şekil 2'de SARS-CoV-XNUMX Omicron varyantı için bir örnek gösterilmektedir. 2c. Bu nedenle, tek kaplı RT-caPCR tahlilimiz, tek bir reaksiyonda ilgilenilen RNA hedeflerini doğru bir şekilde tanımlamak için hem sağlam problarla hassas tespitin hem de SNV ayrımcı problarla tek nükleotid çözünürlüğünün aynı anda gerçekleştirilme yeteneğini gösterir.
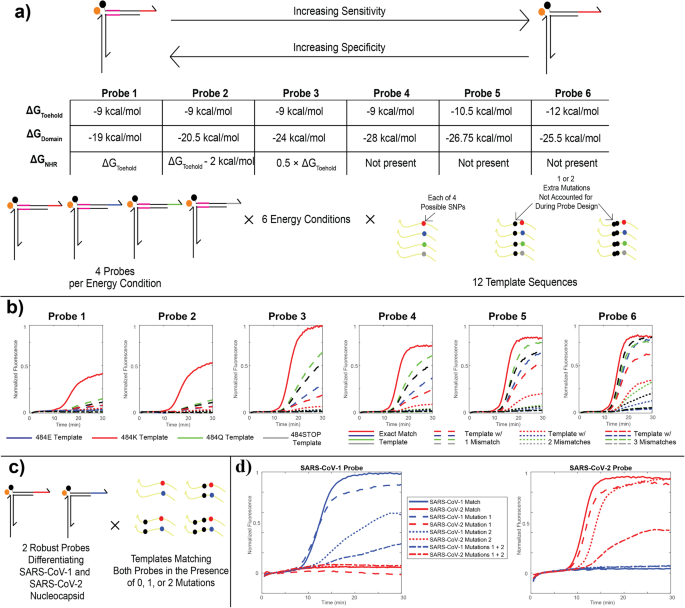
Sağlam ve SNV ayırt edici problar arasındaki enerjinin ince ayarlanması
RT-caPCR tahlilimizde tespit edilen hedeflerin çeşitliliğini arttırmak için, hassasiyet-özgüllük değiş tokuşunu modüle etmek amacıyla parmak bağlama ve iplikçik yer değiştirme enerjilerini optimize ettik. Bir vaka çalışması olarak enerjik değerlendirmemiz için şablon olarak SARS-CoV-2 '484E' genotipini kullandık. Bu bölgedeki mutasyonlar, konakçı antikorlarının nötrleştirme aktivitesinin azalmasıyla ilişkilidir.12ve 3 olası nükleotid bazından 4'ü (adenin, guanin ve sitozin), doğal olarak gelişen SARS-CoV-2 varyantlarında kodonun ilk konumunda bulunur.9. Bu bölgedeki urasil değişimi bir durdurma kodonu üretecektir ve bu nedenle doğada gözlemlenmemiştir, ancak kesinlik açısından değerlendirmemize bir timin durumunu dahil ettik. Bu lokustaki olası 4 nükleotidin her biri için bir prob tasarladık ve bağlanma enerjisine (ΔG) dayalı 6 farklı termodinamik koşul belirledik.o) ayak parmağı probunun 3 bölgesinin (NHR, alan ve ayak parmağı; Şekil XNUMX). 3a, bkz. Malzemeler ve Yöntemler). Bu enerjik koşullar, NHR ve ayak parmağının enerjilerinin yaklaşık olarak eşit olduğu (yaklaşık -1 kcal/mol) en spesifik (Prob 9) ile NHR'nin bulunmadığı ve en hassas (Prob 6) arasında değişir. ayak parmağı biraz daha güçlüdür (−12 kcal/mol). Her durumda, alan bağlama enerjisi, genel prob bağlama enerjisini yaklaşık -40 kcal/mol'de sabit tutacak şekilde ayarlanır.
a Prob enerjik karakterizasyonu için tasarım parametreleri. Tek nükleotid özgüllüğü ile mutasyonlara karşı sağlamlık biçiminde duyarlılık arasında bir değiş tokuşa izin vermek için ayak parmağı, alan ve NHR bölgelerinin bağlanma enerjileri değiştirilerek altı farklı enerji şeması tasarlandı. Altı enerjik tasarımın her biri için, SARS-CoV-484 spike proteinindeki 2 mutasyon bölgesindeki dört olası kodonun tümünü kapsayan dört farklı prob tasarlandı; bunlardan üçü varyantlarda gözlendi. Her bir prob, tek başına 12 eşsiz tek nükleotid mutasyonu dahil olmak üzere ve Omicron varyantının ilerisinde bulunan bir veya iki başka mutasyonun varlığında 4 farklı plazmit şablonuyla test edildi. b caPCR, 484 K serisi problardan elde edilir. Kırmızı katı eğriler, hiçbir mutasyon olmadan hedeflenen performansı gösterir. Kesikli, noktalı ve kesikli noktalı çizgiler, prob dizisine göre sırasıyla 1, 2 veya 3 uyumsuzluğa sahip şablonları belirtir. Renk, 484 bölgesindeki kodonu belirtir (E, K, Q veya STOP). c Sağlam SARS-CoV-1/2 ayrımcılığını test etme planı. Enerji şemasına göre iki prob tasarlandı “Sonda 5” kısmen a SARS-CoV-1 ve SARS-CoV-2'deki nükleokapsid geninin bir bölgesi için. Her iki prob da, tam eşleşme şablonu ve iki yukarı akış uyumsuzluğundan birini veya her ikisini içeren varyantların yanı sıra diğer probun tam eşleşme şablonu ve uyumsuz varyantlarının varlığında test edildi. d ('de açıklanan testlerin sonuçları)c). Düz, kesikli, noktalı ve noktalı çizgiler, SARS-CoV-1 (mavi) veya SARS-CoV-2 (kırmızı) genomuna göre tam eşleşme şablonlarını, iki SNV'den herhangi birine sahip şablonları veya her iki SNV'ye sahip şablonları belirtir.
Bu probları 12 farklı DNA şablonuyla test ettik; bunlardan 4'ü, 4 kodonundaki 484 olası bazın her biri için tam eşleşme şablonunu temsil ediyor. Sonda enerjilerimizin ayarlanabilirliğini göstermek için SARS-CoV-2 evriminin sonraki soylarında ortaya çıkan mutasyonları aldık ve bunları şablonlara dahil ettik: Delta AY ve Omicron soylarında bulunan T478K mutasyonu ve Omicron'da bulunan S477N mutasyonu. soylar (şematik Şek. 3a)10. 24 probun tümü, 12 şablonun her birinin amplifikasyonuna yanıt olarak değerlendirildi. Figür 3b özellikle Beta ve Gama VOC'lerde bulunan 484 K mutasyonunu hedeflemek üzere tasarlanmış problar için bu sonuçların bir alt kümesini gösterir10. NHR'nin gücü azaldıkça ve tutunma arttıkça, tam eşleşme şablonunun (düz kırmızı çizgi) tespiti güçlü kalır ve prob, 1 (kesikli çizgiler) ve 2 (noktalı çizgiler) uyumsuzluğu olan şablonlara karşı daha duyarlı hale gelir. Bu, çok spesifik hedef bağlama sonuçları için araştırma enerjilerini tasarlama yeteneğimizi göstermektedir. Bu sonuçlara dayanarak, SNV'yi ayırt eden problar için enerjik şema 2'yi ve sağlam problar için şema 5'i seçtik.
SARS-CoV-2 ve SARS-CoV-1'nin nükleokapsid gen dizisini hedef alan 2 sağlam prob tasarladık. Bu patojen dizilerinin, mutasyonlara karşı toleranslı kalırken sağlam problar kullanılarak farklılaştırılabilmesini sağlamak için, ya hiç uyumsuzluk, 8 uyumsuzluktan 1'i ya da her iki uyumsuzluğu bir arada içeren 2 şablon tasarladık (Şekil XNUMX). 3c, bkz. Malzemeler ve Yöntemler). SARS-CoV-1 ve SARS-CoV-2 tespiti için seçilen bölge, 2 hedef dizi arasında 8 mutasyon kadar farklılık göstermektedir: bu nedenle şablondaki uyumsuzluklar, bu farklılıklara göre seçilmiştir. Bu nedenle, 2 uyumsuzluğa sahip SARS-CoV-2 şablonu, SARS-CoV-6 dizisine göre yalnızca 1 farklılığa sahiptir, bu da probların duyarlılık ve özgüllük açısından hassas şekilde ayarlanabilmesinin öneminin gerçek hayattaki bir uygulamasını vurgulamaktadır. Figür 3d bu testlerin sonuçlarını gösterir. Tüm vakalarda SARS-CoV-1 ve SARS-CoV-2 probları ve şablonları arasında çapraz reaktivite gözlemlemedik: SARS-CoV-1 RNA'sı yalnızca SARS-CoV-1 probu tarafından tespit edildi ve SARS-CoV-2 RNA yalnızca SARS-CoV-2 probu tarafından tespit edildi. 2 mutasyondan herhangi birini içeren şablonlar, her bir uygun prob tarafından açık bir şekilde tespit edilebilir; 1 mutasyondan 2'i, performansı diğerine göre daha açık bir şekilde etkiler. Benzer şekilde, her iki mutasyonun birlikte olduğu şablonlar hala tespit edilse de, söndürücü kolunun yer değiştirmesindeki daha az enerjik tercih nedeniyle performans biraz tehlikeye girmiştir.
Prob enerjisine yönelik hedefli çalışmalarımız aracılığıyla, hassasiyet ve spesifiklik arasında arzu edilen dengeyi sağlayacak şekilde probları hassas bir şekilde tasarlama kapasitemizi gösterdik. SNV ayrımcı problar, söndürücü kolun yer değiştirmesi için tam bir eşleşme gerektiren enerjilere sahipken, sağlam problar, iki mutasyona kadar içeren bir amplikonu tespit etme yeteneği ile hedeflerin bağlanmasında daha karışıktır. Araştırdığımız enerji aralığı, prob dizisinden arzu edilen sayıda uyumsuzluğa sahip hedeflerin tespit edilmesine olanak sağlamak üzere enerji şeması 1'den enerji şeması 6'ya kadar probları gerektiği gibi ayarlamak için uygulanabilir.
Sağlam ve SNV ayrımcı probların performansı
Yüksek düzeyde ayarlanabilir prob tasarımımızın kavram kanıtını göstermek için hem sağlam hem de SNV ayrımcı problardan oluşan tam bir panel oluşturduk. Panelimiz, gribin yanı sıra insanları enfekte ettiği bilinen 7 koronavirüsün tamamını (HCoVs 229E, HKU1, NL63 ve OC43; MERS-CoV, SARS-CoV-1 ve SARS-CoV-2) tarayarak sağlam probların faydasını vurgulamaktadır. A ve B8. Ek olarak panelimiz, ilgili varyantlar arasında farklılaşmaya izin vermek için SARS-CoV-14 spike geninde yaygın olarak mutasyona uğramış 3 bölgeyi kapsayan 5 prob ve 2 amplikon kullanarak SNV ayrımcı probların işlevselliğini göstermektedir (417, 452, 484, 501 ve 614; Şek. S2)9,10,11.
Hassas hedef tespiti için RT-caPCR tahlilimizde sağlam probların performansını değerlendirmek amacıyla, çeşitli hasta demografik özelliklerini kapsayan koronavirüs negatif viral taşıma ortamı (VTM) numunelerini kullanarak 65 RT-caPCR testi gerçekleştirdik (Tablolar) S1-S4) Twist Bioscience'dan satın alınan veya gen parçalarından in vitro transkripsiyon (IVT) kullanılarak şirket içinde sentezlenen 7 insan koronavirüsünün her birinden sentetik RNA eklenmiş (Tablo) S5). Boncuk bazlı bir ekstraksiyon yöntemi kullanılarak bu hazırlanmış klinik örneklerden RNA izole edildi ve saflaştırıldı13 SARS-CoV-2 teşhis iş akışlarında RNA ekstraksiyonu için bu tür metodolojinin kullanılmasının önceliği nedeniyle14 sistemimize girdi olarak kullanılmıştır. Bu tahlil aynı zamanda hem bir ekstraksiyon kontrolünü (saflaştırmadan önce hazırlanmış numuneye eklenen MS2 bakteriyofajından RNA) hem de bir amplifikasyon kontrolünü (RT-caPCR reaksiyon karışımına eklenen dahili pozitif kontrol (IPC) RNA'sını) içeriyordu. Aksi belirtilmedikçe buradaki deneyler yaklaşık 106 MS2 RNA'nın kopyaları ve 107 viral RNA'nın kopyaları. RT-caPCR reaksiyonu, 40 IPC kontrol RNA molekülü, ekstrakte edilmiş örnek RNA ve tüm hedefleri ve kontrolleri kapsayan 5,000 primer çifti kullanılarak 11 dakikada gerçekleştirildi.
RT-caPCR verileri, her örnekte mevcut viral hedefleri ve SARS-CoV-2 durumunda hangi spesifik varyantın mevcut olduğunu belirlemek için analiz edildi (Şekil XNUMX). 4). şekil 4a RNA hedefi olarak SARS-CoV-2 Omicron varyantının kullanıldığı temsili bir çalışmayı gösterir. Viral ayrımcılık için tüm sağlam problar kırmızı renkle gösterilirken, SNV ayırt edici problar mavi renkle gösterilmiştir. Ekstraksiyon ve amplifikasyon kontrolleri yeşil renkle gösterilmiştir. Pozitif ve negatif prob noktaları, hiçbir primerin veya şablonun dahil edilmediği ve gri renkle gösterilen dizileri kullanan tamamen söndürülmemiş veya tamamen söndürülmüş problara karşılık gelir. Bu tür veriler, Şekil XNUMX'de gösterildiği gibi sıkıştırılmış bir grafikte özetlenebilir. 4b: beklenen tüm pozitif eğriler sol taraftaki grafikte gösterilmektedir ve beklenen tüm negatif eğriler sağ taraftaki grafikte gösterilmektedir. Bazı SNV'ye özgü problarda küçük düzeyde hedef dışı aktivite olsa da, hedefteki SNV'ye özgü prob eğrileri (484 A, 452 L, 501Y-Om, 417 N ve 614 G) diğer SNV- ile karşılaştırıldığında Spesifik problar, hedeflenen probların, hem gözle hem de floresans türevi bazlı SNV ayrımcılık algoritmamız aracılığıyla değerlendirildiği üzere tüm durumlarda en güçlü şekilde yanıt verdiğini doğrular (bkz. Malzemeler ve Yöntemler ve Ek Yöntemler). Omicron varyantının diğer varyantlara göre yüksek mutasyon yükü nedeniyle11tespitini iyileştirmek için 484 A probunun dizisini, bu varyantta bulunan S477N ve T478K mutasyonlarını da içerecek şekilde tasarladık.10. Benzer şekilde benzersiz bir 501Y-Om probu, Omicron varyantında spesifik olarak bulunan Q493R, G496S ve Q498R mutasyonlarını içerir. Omicron soyunun BA.2 alt değişkenine (501Y-Om2) özel bir prob daha sonra tanıtıldı. Bu prob 501Y-Om dizisiyle aynıdır ancak G496S mutasyonundan yoksundur10.
a Örnek, söz konusu SARS-CoV-2 Omicron varyantını tespit eden bir RT-caPCR çalışmasının sonuçlarıdır. Sağlam viral tespit probları kırmızı renktedir, SNV ayırt edici problar mavi renktedir, ekstraksiyon ve amplifikasyon kontrolleri yeşil renktedir ve pozitif ve negatif kontroller gri renktedir. Her eğri, dizideki en parlak noktaya normalize edilmiş, elde edilen 60 görüntü üzerinden her zaman noktasında karşılık gelen prob dizisi noktasının yoğunluğunu temsil eder. b Çalıştırmaya karşılık gelen özet sonuçlar a. Beklenen tüm pozitifler (ekstraksiyon ve amplifikasyon kontrolleri, hedef viral tanımlama ve tüm hedef SNV ayırt edici problar) soldaki grafikte gösterilirken beklenen tüm negatif problar (diğer tüm viral ve SNV ayırıcı problar, aşağıdaki şekilde) Negatif kontrollerin yanı sıra) sağ taraftaki grafikte gösterilmektedir. c İnsan koronavirüsü 229E, Orta Doğu Solunum Sendromu koronavirüsü (MERS-CoV) ve SARS-CoV-1 için ('de açıklanan formatta) viral tespit sonuçlarıb). d. SNV ayrımcılığı, yukarıda belirtilen formattaki SARS-CoV-2 Delta varyant şablonundan kaynaklanır.
şekil 4c 3 farklı viral patojen şablonu için 3 çalışmanın sıkıştırılmış temsili sonuçlarını gösterir: insan koronavirüsü 229E, MERS-CoV ve SARS-CoV-1. 3 numunenin tümü, güçlü patojen tanımlama probları ile viral RNA hedefinin net bir şekilde tespit edildiğini, hedef dışı viral tanımlama prob aktivitesinin bulunmadığını ve tüm kontrollerin varlığını göstermektedir.
tablo 1 tüm geçerli denemelerden elde edilen viral tanımlama sonuçlarını özetler. PCR reaksiyon kaynakları (örn. polimeraz, dNTP'ler) için rekabet, düşük kopya sayısı hedeflerinin amplifikasyonunu baskılayabilir; dolayısıyla bir viral patojen tespit edildiğinde ve 1 kontrolden en az 2'i (MS2 ve IPC) tespit edildiğinde bir test çalışması geçerli kabul edildi. veya hiçbir viral patojen tespit edilmedi ancak her iki kontrol de tespit edildi. Örneklerin 11'i, yalnızca MS2 ve IPC kontrol RNA'larını içeren negatif kontrollerdi. Bu numunelerin 11'inin tamamında hem IPC hem de MS2 tespit edildi (%100). 36 SARS-CoV-36 örneğinden 2'sı (%100) SARS-CoV-2 nükleokapsid probunun tespiti ile değerlendirilerek tanımlandı. Geriye kalan insan koronavirüsleri (229E, NL63, OC43 ve HKU1) ile SARS-CoV-1 ve MERS-CoV hedefleri de tüm hedefler için %100 tespitle yüksek hassasiyet göstermektedir. Hem primer bağlama işlemi hem de probların amplikon aracılı söndürücünün yer değiştirmesi, tüm hedeflerde %100 olan panelin özgüllüğüne katkıda bulunur. Burada özgüllük, doğru şekilde tespit edilen gerçek negatiflerin oranı olarak hesaplanır. Kısacası, hiçbir şablon, özel olarak tasarlandığı prob dışında sağlam bir probta tespite neden olmamıştır.
şekil 4d RT-caPCR tahlilimizin hem RNA hedefini tanımlama hem de SARS-CoV-2 Delta varyantı için patojen tanımlamayla eşzamanlı olarak viral varyant tespitini göstererek tek nükleotid çözünürlüğü sağlama yeteneğini vurgulayan temsili bir çalışmayı gösterir. 452 L probu önemli ölçüde hedef dışı aktivite gösterirken, tespiti hedeflenen 452 R probundan (sol taraftaki grafikte en yüksek yoğunluğa sahip mavi eğri) açıkça daha az verimlidir. Böylece, SNV'yi ayırt eden problar, yalnızca birkaç tek mutasyonla farklılık gösteren SARS-CoV-2 varyantları arasında net bir ayrım yapılmasına olanak tanır.
tablo 2 SARS-CoV-2 varyantları için SNV'ye özgü probların 36 denemedeki performansını özetlemektedir. Beklenen tespitlerin sayısı, söz konusu mutasyonu görüntüleyen bir şablonun dahil edildiği çalıştırma sayısına eşittir; beklenen negatiflerin sayısı ise karşılık gelen şablonun dahil edilmediği çalıştırma sayısıdır. Gerçek pozitifler, ilgilenilen probun o mutasyon bölgesi için baskın SNV'ye özgü prob olduğu çalışmalardır (algoritmik olarak değerlendirildiği üzere) ve gerçek negatifler ise, ilgili olmadığı çalışmalardır. Bir çalışmada, 614 G probunun yabancı bir parçacık (toz) tarafından engellendiğini, dolayısıyla bu hedeflerin toplamının 35 yerine 36 olduğunu fark ettik. 484E dışındaki tüm problar en az %90 hassasiyet gösteriyor. 484E probunun zayıf performansı, Delta varyantlarına yönelik amplikonda bir SNV'nin (T478K mutasyonu) varlığıyla kısmen açıklanabilir. Diğer tüm problar %100 özgüllük gösterir; çünkü bir amplikon, hedeflenen enerjilerin eşit olduğu varsayılarak her zaman mutasyona uğramış söndürücüye göre eşleşen söndürücüyü tercihli olarak değiştirir.
Birlikte, bu veriler, sağlam probların ve SNV ayrımcı probların, mevcut RNA hedefini hızlı bir şekilde tanımlamak (sağlam prob ile) ve tek nükleotid çözünürlüğü (SNV ayrımcı prob ile) sağlamak için birlikte çalışabilme yeteneğini vurgulamaktadır.
RT-caPCR'nin tespit sınırının değerlendirilmesi
Daha sonra RT-caPCR tahlilimizin tespit sınırını belirlemek için bir dizi deney gerçekleştirdik. Şekil XNUMX'deki önceki tüm testlerde. 4, 10'luk viral girdi7 Omicron ve Delta hastaları için en yüksek viral yük tahminlerine benzer şekilde moleküller ekstraksiyon öncesinde VTM'ye eklenmiştir.15,16. Burada viral RNA'nın giriş kopyalarının sayısını değiştirerek ve kontrol kopyalarını aynı tutarak testimizin LOD'sini ölçtük. Twist Bioscience'dan elde edilen veya IVT yoluyla gen parçalarından şirket içinde sentezlenen saflaştırılmış RNA'yı kullanarak, 1,000 koronavirüsün tümü için 7 moleküllük bir LOD elde ettik. Temsili sonuçlar Şekil XNUMX'de gösterilmektedir. S3. Her iki Şekilde de. S3a ve S3b, her iki kontrolle de tek bir hedef için reaksiyon başına 1,000'e kadar molekülü başarılı bir şekilde tespit edebildiğimizi bulduk (HCoV 229E; Şekil XNUMX). S3a) ve her iki kontrolü de içeren bir SARS-CoV-2 varyantı için (Omicron; Şekil XNUMX). S3b), saflaştırılmış RNA kullanılırken VTM testi için kullanılanın 3 büyüklüğünün altındaki bir miktar. Şekil XNUMX'de olduğu gibi yalnızca tek bir hedefin varlığı değiştirildiğinde. S3a, yalnızca bu hedef için her büyüklük sırası için eşik süresinde net bir gecikme buluyoruz, halbuki 2 kontrol çalıştırmalar arasında tutarlı. Bu hedefin 2 amplikon ve 4 kontrolün eşzamanlı amplifikasyonunu gerektirdiği göz önüne alındığında bu eğilim SARS-CoV-2 için daha az belirgindir. Enzimatik kaynaklar ve dNTP'ler için rekabet, tek amplikonlu hedefler için gözlemlenen net eğilimi bozuyor gibi görünüyor. İncirde. S3COmicron varyantı için ekstraksiyon boru hattına giriş RNA miktarını değiştirerek bir seyreltme serisi gerçekleştirildi. Burada da test edilen en düşük değere kadar tespit görüyoruz (RNA ekstraksiyon adımına 10,000 molekül girişi, ekstraksiyon sırasındaki kayıplar nedeniyle ve reaksiyonda 1,000 µL elüsyon hacminin tamamının kullanılmaması nedeniyle yaklaşık 25 molekül RT adımına giriyor) ve giriş konsantrasyonunun bir fonksiyonu olarak amplifikasyonda beklenen bir gecikme.
Tek bir örnekte birden fazla hedefin tespiti
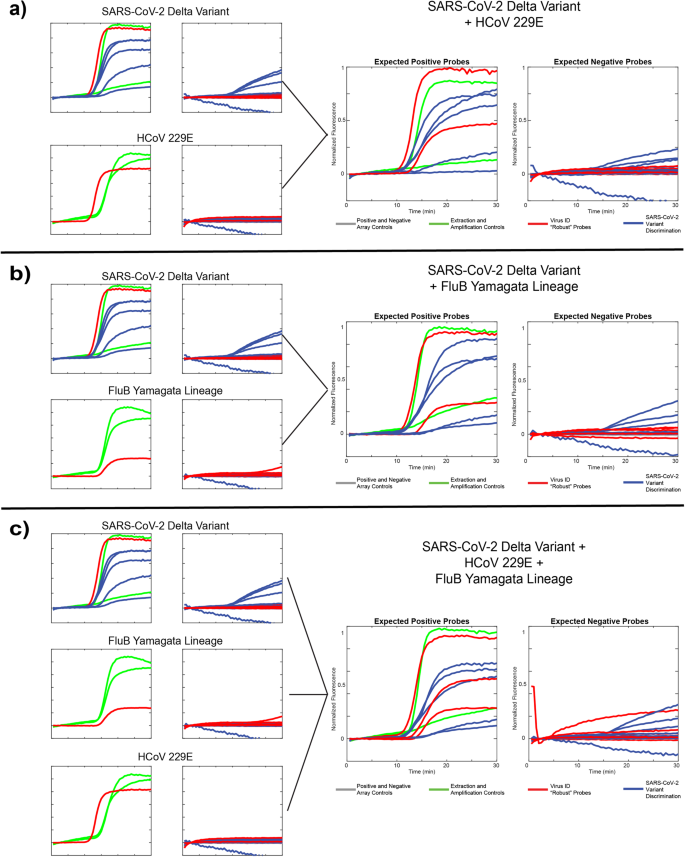
Ayarlanabilir problu RT-caPCR testimizin, SNV ayırt edici problarla (SARS-CoV-2 varyantları) tek nükleotid spesifikliğine sahip sağlam problarla (her virüs) RNA hedeflerini belirlemedeki başarısını gösterdikten sonra, daha sonra testimizin aşağıdaki durumlarda nasıl performans gösterdiğini araştırdık: Birden fazla RNA hedefi içeren ortak enfeksiyon örneklerini simüle ederek daha karmaşık girdilerin değerlendirilmesi. Burada giriş miktarı 105 Her şablonun molekülleri, hiçbir VTM ekstraksiyon aşaması olmadan doğrudan RT-caPCR reaksiyonuna eklendi. Ayrıca RT-caPCR tahlilimizi, influenza A ve B'yi test etmek için 3 ek primer seti ve 4 prob (2'si influenza A'nın M1 matris protein genini hedefleyen) dahil ederek daha fazla RNA hedefini test etmek üzere genişlettik.17 ve influenza B'nin Yamagata ve Victoria soylarındaki yapısal olmayan protein-2'i (nsp1) hedefleyen 118). Bu 14-plex tahlili, etkinliğini doğrulamak amacıyla 4 yeni hedefin tümü için test edildi (Şekil XNUMX). S4).
3 farklı viral şablonun her birini tek başına ve 3 farklı kombinasyon halinde değerlendirdik: SARS-CoV-2 Delta varyantı + HCoV229E (Şekil XNUMX). 5a), SARS-CoV-2 Delta varyantı + FluB Yamagata soyu (Şek. 5b) ve SARS-CoV-2 + HCoV229E + FluB Yamagata soyu (Şekil XNUMX). 5c). Veriler, sol panelde beklenen pozitif problar ve sağ panelde beklenen negatif problar ile yukarıda açıklandığı gibi sıkıştırılmış floresan iz görüntüleri olarak sunulur. Tüm çalışmalarda beklenen davranışı gözlemledik: sağlam (virüs tanımlama) problar, dahil edilen tüm patojen şablonlarının varlığını rapor eder ve SNV ayırt edici problar (varyant tanımlama), tümünde düşük düzeyde hedef dışı prob aktivitesi ile Delta varyantını doğru şekilde tanımlar. 5 SNV sitesi. 484E probu, muhtemelen bu probun etki alanı bölgesindeki yukarıda belirtilen uyumsuzluk nedeniyle en zayıf performansa sahip olanıdır. Ek olarak bazı deneylerde MS2 ekstraksiyon kontrolü, 8 farklı amplikon arasındaki kaynaklar için rekabet nedeniyle zayıf performans gösterdi. Ayrıca bu çalışmaların, Şekil 1'deki gibi 2 yerine, MS2 ve IPC kontrollerinin her birinin yalnızca XNUMX kopyasını içerdiğini de not ediyoruz. 4çünkü bu, 501Y-Om2 probunu içeren güncellenmiş bir paneldi. Bu veriler, teknolojimizin ilgilenilen birden fazla RNA hedefi içeren daha karmaşık numuneleri doğru bir şekilde değerlendirme yeteneğini ortaya koyarak testin çok yönlülüğünü ortaya koymaktadır.
a SARS-CoV-2 Delta varyantı ve insan koronavirüsü 229E'den tek şablon sonuçları ve her iki şablonu içeren bir "koenfeksiyon" örneği. b SARS-CoV-2 Delta varyantından ve influenza B'nin Yamagata soyundan tek şablon sonuçları ve her iki şablonu içeren bir koenfeksiyon örneği. c. ile aynı hedeflerden tek hedefli sonuçlar a ve b, üç şablonun tümünü içeren bir koenfeksiyon örneğiyle birlikte.
- SEO Destekli İçerik ve Halkla İlişkiler Dağıtımı. Bugün Gücünüzü Artırın.
- PlatoData.Network Dikey Üretken Yapay Zeka. Kendine güç ver. Buradan Erişin.
- PlatoAiStream. Web3 Zekası. Bilgi Genişletildi. Buradan Erişin.
- PlatoESG. karbon, temiz teknoloji, Enerji, Çevre, Güneş, Atık Yönetimi. Buradan Erişin.
- PlatoSağlık. Biyoteknoloji ve Klinik Araştırmalar Zekası. Buradan Erişin.
- Kaynak: https://www.nature.com/articles/s42003-023-05346-4