การสร้างและจำแนกลักษณะของ hiPSCs และ hiPSC-CMs เฉพาะผู้ป่วย
สาย hiPSC ถูกสร้างขึ้นจากผู้ป่วย TOF-DG สองราย ผู้ป่วย TOF-ND สองราย และการควบคุมสุขภาพสองรายการพร้อมเครื่องหมาย pluripotency และเครื่องหมายชั้นจมูกที่ตรวจสอบแล้ว (มะเดื่อเสริม 1 และ 2). การจัดลำดับจีโนมทั้งหมดได้รับการยืนยันตามลำดับการมีอยู่และไม่มีการลบ 22q11.2 ในสาย hiPSC จากผู้ป่วย TOF-DG และ TOF-ND (รูปที่ XNUMX เพิ่มเติม) 3). ไม่พบการกลายพันธุ์ที่ผิดพลาดหรือไร้สาระของยีนที่เกี่ยวข้องกับ CHD ที่รายงานก่อนหน้านี้จากสาย hiPSC ใด ๆ การแยกความแตกต่างของหัวใจทำได้สำเร็จโดย APLNR+ โปรโตคอลการคัดแยกที่พัฒนาโดยห้องปฏิบัติการของเรา32. สาย hiPSC ทั้งหมดแสดงประสิทธิภาพการแยกความแตกต่างที่น่าพอใจด้วยเซลล์ TNNT70+ มากกว่า 2% ในวันที่ 12 หลังการแยกความแตกต่าง hiPSC-CM ถูกสร้างขึ้นใน hCAS และปล่อยให้สุกต่อไปอีก 10 วันก่อนการประเมินการถอดเสียงและการทำงาน
TOF-DG เฉพาะผู้ป่วย hiPSC-CMs แสดงข้อกำหนดกระเป๋าหน้าท้องที่มีข้อบกพร่อง
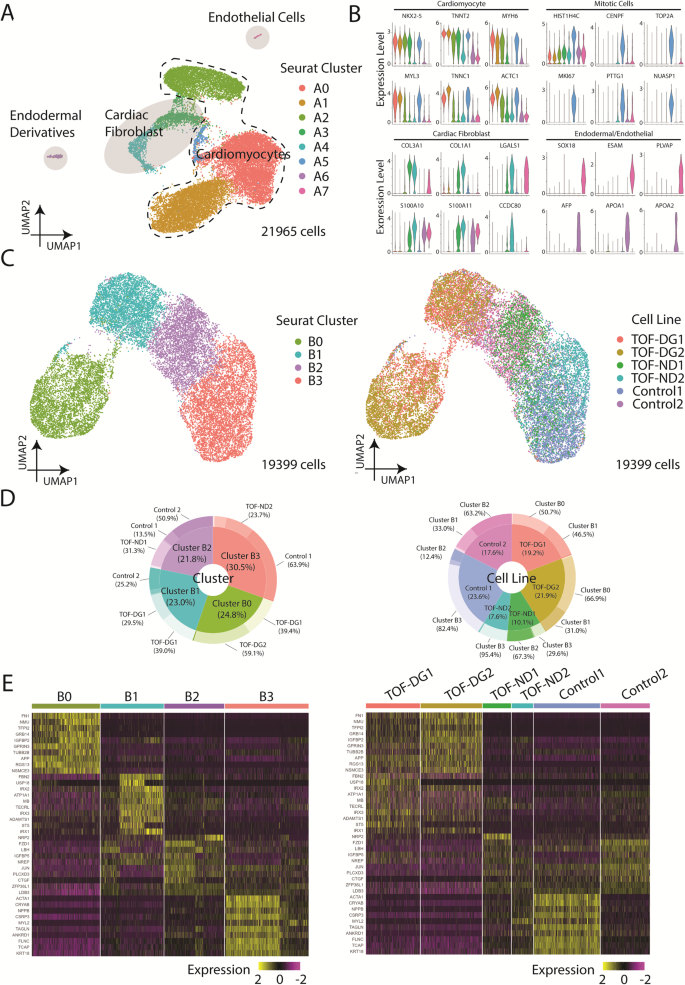
ก่อนอื่นเราได้พิจารณาโปรไฟล์การถอดเสียงของ hiPSC-CM ที่แตกต่างจากผู้ป่วยและการควบคุมโดย scRNA-seq โดยใช้แพลตฟอร์ม 10X Genomics การรวมกลุ่มของเซลล์ 21,965 เซลล์โดยใช้การประมาณค่าท่อร่วมหลายเท่าและการฉายภาพ (UMP) ระบุกระจุกซูรัตที่แตกต่างกัน 7 กลุ่ม (รูปที่ 1A). กลุ่ม Seurat A0, A1, A2, A5 แสดงระดับยีนหัวใจที่ค่อนข้างสูงรวมทั้ง เอ็นเคเอ็กซ์2-5, ไมโอซินสายเบา/หนัก และโทรโปนิน (รูปที่. 1B) ซึ่งระบุกลุ่มเหล่านี้เป็นคาร์ดิโอไมโอไซต์ คลัสเตอร์ A5 ยังแสดงยีนไมโทติสในระดับที่สูงขึ้นด้วย ซึ่งบ่งบอกถึงลักษณะการเพิ่มจำนวนของเซลล์คาร์ดิโอไมโอไซต์ กลุ่ม A3 และ A4 แสดงระดับยีนหัวใจค่อนข้างต่ำ (เอ็นเคเอ็กซ์2-5, ทีเอ็นที2) แต่เสริมสมรรถนะในการแสดงออกของยีนที่เกี่ยวข้องกับการเปลี่ยนแปลงเมทริกซ์นอกเซลล์ (ECM) (รูปที่. 1B) โปรไฟล์ที่เข้ากันได้กับไฟโบรบลาสต์หัวใจ คลัสเตอร์ A6 และ A7 ถูกกำหนดโดยเชื้อสายเอ็นโดเดอร์มัล (เอเอฟพี, APOA1, APOA2) และบุผนังหลอดเลือด (FLT1, ESAM, PLVAP) เครื่องหมาย ตามลำดับ (รูปที่. 1B). ดังนั้น ในบรรดาเซลล์ที่เรียงลำดับ ส่วนใหญ่เป็นคาร์ดิโอไมโอไซต์ (86%) บางส่วนเป็นไฟโบรบลาสต์ของหัวใจ (10%) และมีเพียงไม่กี่เซลล์ที่เป็นเอนโดเดอร์มอล (<1%) และอนุพันธ์ของเอนโดธีเลียม (<1%)
A การนำเสนอ UMAP ของเซลล์ทุกประเภทที่ระบุใน hCAS (TOF-DG, TOF-ND และส่วนควบคุม) การระบายสี: กลุ่ม Seurat; เส้นประ: cardiomyocytes; สีเทาอ่อน: ไม่ใช่ไมโอไซต์ B แผนไวโอลินของการแสดงออกของยีนของคาร์ดิโอไมโอไซต์ เซลล์ไมโทติค และเซลล์ที่ไม่ใช่มัยโอไซต์: ไฟโบรบลาสต์ของหัวใจ อนุพันธ์ของเอ็นโดเดอร์มอล และเซลล์บุผนังหลอดเลือด การระบายสี: กลุ่มเซลล์ Seurat ดังใน (A). C การนำเสนอ UMAP ของ hiPSC-CM ทั้งหมดที่ระบุใน hCAS (TOF-DG, TOF-ND และการควบคุม) ข้อมูลถูกระบายสีโดยกลุ่ม Seurat (แผงด้านซ้าย) และเส้นเซลล์ (แผงด้านขวา) ตามลำดับ D แผนโดนัทขององค์ประกอบในแต่ละคลัสเตอร์ Seurat (แผงด้านซ้าย) และเส้นเซลล์ (แผงด้านขวา) ตามลำดับ ข้อมูลคำนวณจาก hiPSC-CM เท่านั้น E การนำเสนอแผนที่ความร้อนของยีนที่ได้รับการควบคุม 10 อันดับแรกที่แสดงในแต่ละคลัสเตอร์ hiPSC-CMs-Seurat ข้อมูลถูกจัดกลุ่มตามคลัสเตอร์ Seurat (แผงด้านซ้าย) และเส้นเซลล์ (แผงด้านขวา) ตามลำดับ
ด้วยการมุ่งเน้นไปที่การตรวจสอบทรานสคริปต์โตมของหัวใจ เราได้แยก hiPSC-CMs (คลัสเตอร์ A0, A1, A2, A5) และดำเนินการจัดกลุ่มโดยใช้ UMAP (รูปที่. 1C). กระจุก Seurat ใหม่ (B0, B1, B2, B3) ถูกระบุ (รูปที่ 1C). hiPSC-CM ที่ได้มาจากผู้ป่วยและกลุ่มควบคุมมีการกระจายอย่างไม่สม่ำเสมอระหว่างกระจุก (รูปที่ 1D). คลัสเตอร์ B0 ประกอบด้วย hiPSC-CMs เกือบเฉพาะ (98%) ที่ได้มาจากผู้ป่วย TOF-DG คลัสเตอร์ B1 ประกอบด้วยเซลล์ส่วนใหญ่มาจากกลุ่มควบคุมและเซลล์ที่เหลือจากผู้ป่วย TOF-DG ในขณะที่คลัสเตอร์ B2 และ B3 ประกอบด้วย hiPSC-CM มาจากผู้ป่วย TOF-ND และกลุ่มควบคุม
พบว่าคลัสเตอร์ B0 มีการแสดงออกของยีนที่ไม่ใช่ไมโอไซต์ที่ถูกควบคุม โดยมียีนที่ได้รับการควบคุมสูงสุด 4 ใน 10 ยีนที่ไม่ค่อยพบ (<1%) ในกลุ่มอื่น (รูปที่. 1E). ยีนที่ไม่ใช่ไมโอไซต์เหล่านี้ไม่เกี่ยวข้องกับเอ็นโดเดอร์มอลหรือบุผนังหลอดเลือด การไม่มีการเพิ่มคุณค่าของยีนที่เกี่ยวข้องกับการเปลี่ยนแปลงเมทริกซ์นอกเซลล์ยังทำให้เซลล์ B0 คลัสเตอร์แตกต่างจากไฟโบรบลาสต์ของหัวใจ คลัสเตอร์ B1 ได้รับการเสริมสมรรถนะใน ไออาร์เอ็กซ์ ตระกูลยีน (IRX1, IRX2, IRX3) ในขณะที่คลัสเตอร์ B2 ถูกทำเครื่องหมายโดย LBH การแสดงออก (รูปที่ 1E). ออโธโลจีของ ไออาร์เอ็กซ์ ยีนและ LBH มีรายงานว่ามีการแสดงออกในช่วงต้นของ cardiomyocytes ของ murine ที่มีความแตกต่างในร่างกาย33,34. คลัสเตอร์ B2 เมื่อเปรียบเทียบกับคลัสเตอร์ B1 มีการควบคุมการแสดงออกของเครื่องหมายคาร์ดิโอไมโอไซต์ในกระเป๋าหน้าท้อง (MYH7, NPPB). กลุ่ม B3 แสดงการแสดงออกที่สูงที่สุดของตัวบ่งชี้คาร์ดิโอไมโอไซต์ที่มีกระเป๋าหน้าท้อง (มายแอล2) (รูปที่ 1E). การมีอยู่ของกลุ่ม B1, B2 และ B3 แนะนำข้อกำหนดแบบก้าวหน้าของ hiPSC-CMs ที่มีต่อโปรไฟล์การถอดเสียงของกระเป๋าหน้าท้อง
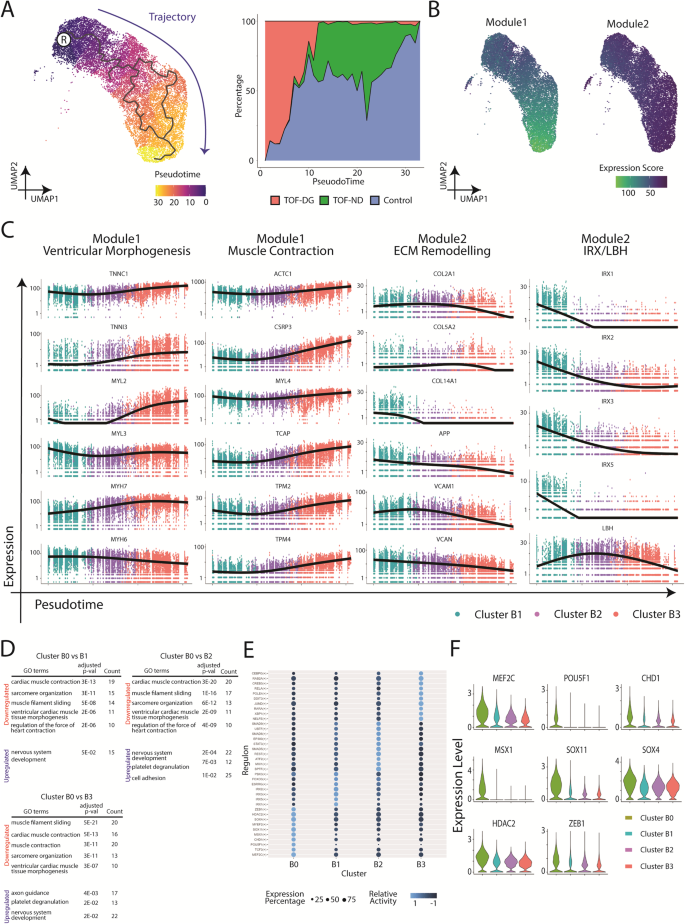
เพื่อตรวจสอบการเปลี่ยนแปลงของการถอดเสียงเพิ่มเติมในระหว่างข้อมูลจำเพาะของกระเป๋าหน้าท้อง เราทำการวิเคราะห์ pseudotime บนคลัสเตอร์ B1, B2 และ B3 ขึ้นอยู่กับการเสริมแต่งใน ไออาร์เอ็กซ์ ตระกูลยีน คลัสเตอร์ B1 ถูกเลือกด้วยตนเองเป็นโหนดรูทสำหรับการวิเคราะห์เวลาเทียม (รูปที่. 2A). ตามวิถีโคจร TOF-DG-hiPSC-CMs ส่วนใหญ่จะกระจายในช่วงเริ่มต้น ในขณะที่ TOF-ND-hiPSC-CMs ส่วนใหญ่จะกระจายในช่วงกลางถึงปลาย Control-hiPSC-CMs สามารถพบได้ตลอดวิถี (รูปที่. 2A).
A การวิเคราะห์ Pseudotime ของ hCAS-hiPSC-CMs การประสานงาน UMAP และการแสดงออกของยีนของกลุ่ม B1, B2 และ B3 (รูปที่. 1C) ถูกประมาณไว้สำหรับการคำนวณ Monocle3 โหนดรูทแสดงด้วย ® วิถีถูกย้อมด้วยสีน้ำเงิน B โมดูลยีนทำหน้าที่เป็นฟังก์ชันของการวิเคราะห์เวลาเทียมและการอนุมานวิถีของ hCAS-hiPSC-CM C การเปลี่ยนแปลงของการแสดงออกของยีนตามวิถีโคจร morphogenesis ของกระเป๋าหน้าท้อง การหดตัวของกล้ามเนื้อ และการเปลี่ยนแปลง ECM เป็นคำศัพท์ GO ที่เสริมสมรรถนะในโมดูลยีน X-แกน: เวลาหลอก ; Y-axis: ระดับการแสดงออก เมทริกซ์นอกเซลล์ ECM D การวิเคราะห์การเพิ่มคุณค่า GO ของคลัสเตอร์ B0 จาก hCAS-hiPSC-CMs มีการแสดงคำศัพท์ GO ที่ได้รับการเสริมสมรรถนะในยีนที่ควบคุม/ควบคุมจากคลัสเตอร์ B0 (เมื่อเปรียบเทียบกับคลัสเตอร์ B1/B2/B3) ค่า P ถูกปรับสำหรับการเปรียบเทียบหลายรายการของ Bonferroni E การวิเคราะห์ Regulon ของ hCAS-hiPSC-CMs หน่วยควบคุมสูงสุด (สูงสุด 10) ที่มีกิจกรรมสัมพัทธ์ที่สูงกว่าในแต่ละคลัสเตอร์ Seurat จะแสดงในแผนภาพดอทฮีทแมป F การแสดงออกของยีนของหน่วยงานควบคุมชั้นนำ (ระบุเป็น DEG) จากคลัสเตอร์ B0 การแสดงออกของยีนถูกแสดงไว้ในพล็อตเรื่องไวโอลิน
ยีนที่มีรูปแบบการแสดงออกคล้ายกันถูกจัดกลุ่มเป็นโมดูล (รูปที่. 2B) และอยู่ภายใต้การวิเคราะห์การเพิ่มคุณค่าของ Gene Ontology (GO) พบคำศัพท์ GO ที่สำคัญในทั้งโมดูล 1 และ 2 (รูปที่ 2C). โมดูล 1 ซึ่งแสดงคะแนนการแสดงออกสูงตลอดวิถี ได้รับการเสริมด้วยยีนที่เกี่ยวข้องกับการสร้างรูปร่างของกระเป๋าหน้าท้อง (TNNI3, MYL2, MYH7) และการหดตัวของกล้ามเนื้อ (ACTC1, CSRP3, TCAP). โมดูล 2 ซึ่งแสดงคะแนนการแสดงออกที่ลดลงตามวิถีโคจร ได้รับการเสริมสมรรถนะด้วยยีนที่เกี่ยวข้องกับการเปลี่ยนแปลงเมทริกซ์นอกเซลล์ (COL2A1, COL5A2, COL14A1) ไออาร์เอ็กซ์ ครอบครัวยีนและ LBH ยังได้รับมอบหมายให้อยู่ในโมดูลที่ 2 ซึ่งแสดงคะแนนการแสดงออกที่ลดลงตามวิถี
โดยสรุป การวิเคราะห์ pseudotime บน hiPSC-CM ที่ได้มาจากแพลตฟอร์ม hCAS ระบุข้อกำหนดกระเป๋าหน้าท้องแบบก้าวหน้าพร้อมการแสดงออกของยีนหัวใจที่เพิ่มขึ้น ส่วนควบคุมและ TOF-ND-hiPSC-CM สามารถเทียบเคียงได้ในข้อกำหนดขั้นสูงดังกล่าว อย่างไรก็ตาม TOF-DG-hiPSC-CM ส่วนใหญ่ยังคงรักษาโปรไฟล์การแสดงออกแบบดั้งเดิมไว้มากกว่า
ชุดย่อยของ hiPSC-CMs เฉพาะผู้ป่วย TOF-DG แสดงการลดลงของการแสดงออกของยีนหัวใจ แต่การควบคุมการแสดงออกของยีนประสาท
เพื่อวิเคราะห์การแสดงออกของยีนที่ไม่ใช่ไมโอไซต์เพิ่มเติมในคลัสเตอร์ B0 ยีนที่แสดงออกแตกต่างกัน (DEG) จะถูกระบุโดยการเปรียบเทียบคลัสเตอร์ B0 กับแต่ละคลัสเตอร์อื่น ๆ มีการระบุมากกว่า 200 DEGs ที่มีการควบคุมหรือลดระดับจากคลัสเตอร์ B0 (ข้อมูลเสริม 1). การวิเคราะห์การเพิ่มคุณค่า GO ระบุคำศัพท์ GO ที่สำคัญที่เกี่ยวข้องกับการหดตัวของกล้ามเนื้อหัวใจในยีนที่ควบคุมต่ำ และที่เกี่ยวข้องกับการพัฒนาระบบประสาทของยีนที่ควบคุมในกลุ่ม B0 อย่างต่อเนื่อง (รูปที่. 2D). ดังนั้นคลัสเตอร์ B0 จึงแตกต่างจากคลัสเตอร์ที่เหลือด้วยการแสดงออกของยีนการเต้นของหัวใจที่ลดลง เช่นเดียวกับการแสดงออกของยีนประสาทที่ได้รับการควบคุม อย่างไรก็ตาม ไม่สามารถแมป DEG ข้างต้นใด ๆ ซึ่งรวมถึงยีนหัวใจที่ลดลงและยีนประสาทที่ได้รับการควบคุม สามารถแมปกับภูมิภาคที่ไม่เพียงพอในสาย hiPSC ของ TOF-DG ทั้งสอง
นอกจากนี้เรายังพยายามระบุปัจจัยการถอดรหัส (TFs) และเครือข่ายการควบคุมยีนที่เกี่ยวข้องจากแต่ละคลัสเตอร์ผ่านการใช้งาน Python ของการอนุมานและการจัดกลุ่มเครือข่ายการควบคุมเซลล์เดียว (pySCENIC) ในบรรดา TF 10 อันดับแรกที่มีกิจกรรมสูงกว่าในคลัสเตอร์ B0 มี 4 รายการที่เกี่ยวข้องกับการพัฒนาระบบประสาท (TCF3, SOX11, SOX4 และ ZEB1) และ 2 ในด้านการพัฒนาหัวใจและระบบประสาท (MEF2C และ มายอีฟ2) (รูปที่ 2E). ระดับการแสดงออกของ TF ประสาททั้งห้า (SOX11, SOX4, ZEB1, MEF2C และ มายอีฟ2) ถูกปรับเพิ่มในคลัสเตอร์ B0 เมื่อเปรียบเทียบกับสิ่งเหล่านั้นในกลุ่มอื่น (รูปที่ 2F). การค้นพบการแสดงออกที่ได้รับการควบคุมของ TF ของระบบประสาทเหล่านี้สอดคล้องกับการแสดงออกของยีนประสาทที่เพิ่มขึ้นในคลัสเตอร์ B0 เช่นเดียวกับ DEGs TF ของระบบประสาทข้างต้นไม่ได้ถูกแมปกับภูมิภาคที่ไม่เพียงพอในเซลล์ TOF-DG ทั้งสอง
ดังนั้นส่วนสำคัญของ hiPSC-CMs จาก TOF-DG (คลัสเตอร์ B0) แสดงให้เห็นการลดลงของยีนหัวใจ และการควบคุมของยีนประสาทนอกมดลูก ความล้มเหลวในการแมป DEG และ TF ในคลัสเตอร์ B0 กับภูมิภาค 22q11.2 ชี้ให้เห็นว่าลายเซ็นการถอดเสียงนี้อาจเป็นผลทางอ้อมของความไม่เพียงพอของ haploins ซึ่งแตกต่างจาก hiPSC-CMs จากส่วนควบคุมและกลุ่ม TOF-ND ซึ่งแสดงให้เห็นความต่อเนื่องในข้อกำหนดกระเป๋าหน้าท้อง คลัสเตอร์ B0 ได้สร้างคลัสเตอร์ที่แตกต่างจากส่วนที่เหลือของ hiPSC-CMs จาก TOF-DG (คลัสเตอร์ B1) ต้นกำเนิดของคลัสเตอร์ B0 ได้รับการสำรวจเพิ่มเติมตามที่อธิบายไว้ด้านล่าง
การแยกความแตกต่างของหัวใจแบบแยกส่วนของต้นกำเนิดหัวใจ TOF-DG
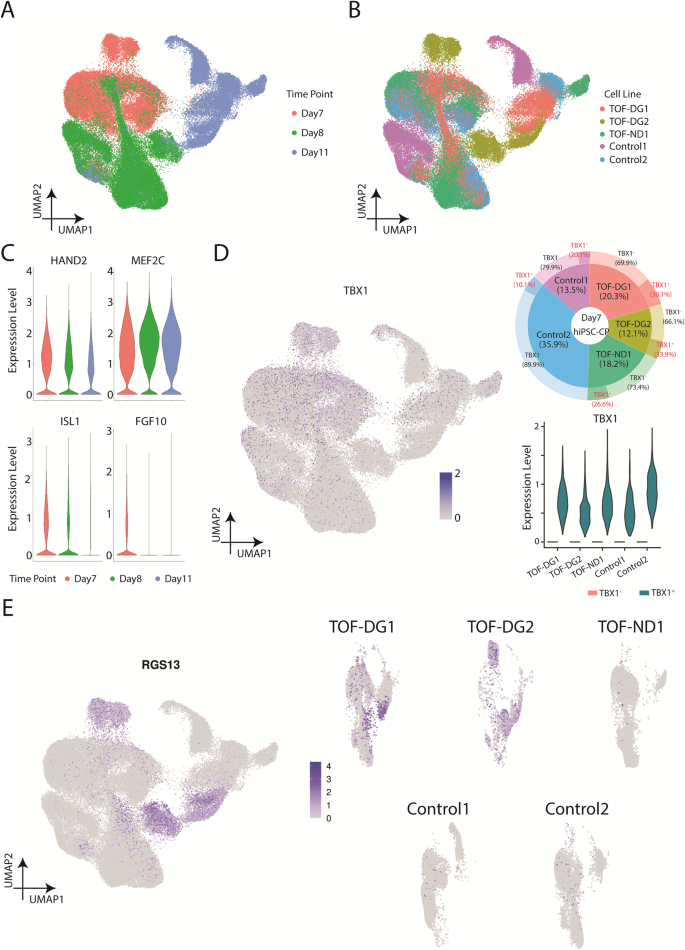
เพื่อให้เข้าใจถึงการเกิดขึ้นของ TOF-DG-cluster B0 เราได้ตรวจสอบโปรไฟล์การถอดเสียงของต้นกำเนิดหัวใจ ในหลอดทดลอง จากการศึกษาครั้งก่อนของเรา32การแสดงออกของยีนสนามหัวใจที่สอง (SHF) สูงสุดในวันที่หลังการสร้างความแตกต่าง (D) 7 และ 8 ดังนั้นเราจึงดำเนินการ scRNA-seq บนต้นกำเนิด hiPSC-cardiac (hiPSC-CPs) บน D7 และ D8 และ hiPSC-CMs ที่ยังไม่บรรลุนิติภาวะบน D11 จาก ตัวควบคุม 2 ตัว (ตัวควบคุม 1 และ 2) และผู้ป่วย TOF-DG (TOF-DG2 และ 1) 2 ตัว และผู้ป่วย TOF-ND (TOF-ND1) 1 ตัว (รูปที่. 3เอ, บี). โดยรวมแล้ว มีเซลล์ 87834 เซลล์ที่ถูกคงไว้หลังจากไม่รวมอนุพันธ์ของเอนโดเดอร์มอลและเซลล์บุผนังหลอดเลือดน้อยกว่า 1% การวิเคราะห์การเพิ่มคุณค่า Pseudotime และ GO ระบุการควบคุมในการแสดงออกของยีนหัวใจ และการลดการควบคุมในการถอดรหัสและการแปล และกิจกรรมวัฏจักรของเซลล์ตามแนววิถี (รูปที่ XNUMX เพิ่มเติม) 4A–D). คล้ายกับการศึกษาครั้งก่อนของเรา32,การแสดงออกของยีน SHF ได้แก่ ISL1, MEF2C, HAND2 และ FGF10พบได้ในจุดเวลาที่มีลำดับ (รูปที่. 3C). TBX1 ซึ่งเป็นหนึ่งในยีนภายในขอบเขตไมโครเดลิชั่น 22q11.2 ก็เป็นเครื่องหมายของ SHF เช่นกัน ท่ามกลางจุดเวลาตามลำดับ TBX1 พบการแสดงออกในวันที่ 7 โดยมีสัดส่วนแปรผันเท่ากับ TBX1+ hiPSC-CPs (10–30%) พบได้จากบรรทัดที่แตกต่างกัน (รูปที่. 3D). อย่างไรก็ตาม TBX1 ระดับการแสดงออกสามารถเทียบเคียงได้ระหว่าง D7-TBX1+ hiPSC-CP จากสายต่างๆ (รูปที่. 3D).
A การนำเสนอ UMAP ของ hiPSC-CP และ hiPSC-CM ทั้งหมด (TOF-DG, TOF-ND และการควบคุม) ข้อมูลถูกระบายสีตามจุดเวลาตามลำดับ B การนำเสนอ UMAP ของ hiPSC-CP และ hiPSC-CM ทั้งหมด (TOF-DG, TOF-ND และการควบคุม) ข้อมูลถูกระบายสีตามเส้นของเซลล์ C แปลงไวโอลินของการแสดงออกของยีน SHF ได้แก่ ISL1, MEF2C, แฮนด์2 และ เอฟจีเอฟ10. D TBX1 การแสดงออกใน hiPSC-CP TBX1 การแสดงออกในวันที่ 7/8/11 การสร้างความแตกต่างการเต้นของหัวใจภายนอกร่างกายถูกแสดงไว้ในพล็อต UMAP สูงสุด TBX1 พบการแสดงออกใน D7-hiPSC-CPs และสัดส่วนของ TBX1+ แสดงในแปลงโดนัทและมีป้ายกำกับสีแดง TBX1 แสดงออกมา TBX1+ D7-hiPSC-CPs ถูกแสดงในพล็อตไวโอลิน E แปลง UMAP ของ RGS13 การแสดงออกในวันที่ 7/8/11 ในหลอดทดลอง ความแตกต่างของหัวใจ RGS13 นิพจน์ถูกนำเสนอพร้อมกับบรรทัดของเซลล์ทั้งหมดพร้อมกัน (แผงด้านซ้าย) และแบ่งออกเป็นทุกบรรทัดของเซลล์ (แผงด้านขวา)
การเปรียบเทียบตามเวลากับส่วนควบคุมระบุเฉพาะ 20, 10 และ 22 DEG จาก D7-, D8- และ D11-TOF-DG-hiPSC-CP/CMs โดยไม่พบคำศัพท์ GO ที่มีนัยสำคัญ ไม่มีการแมป DEG เหล่านี้กับขอบเขตการลบไมโคร 22q11.2 ในสองบรรทัด TOF-DG-hiPSC จากนั้นตรวจสอบการแสดงออกของยีนนอกมดลูกจาก TOF-DG-cluster B0 ใน hiPSC-CPs และ hiPSC-CMs ที่ยังไม่บรรลุนิติภาวะ ต่างจาก TOF-DG-cluster B0 (hiPSC-CMs ที่แตกต่างกัน) ไม่พบยีนที่เกี่ยวข้องกับระบบประสาทใน TOF-DG-hiPSC- CPs (D7 และ D8) และ D11-immature hiPSC-CMs ในทางกลับกัน ชุดย่อยของเซลล์ที่ทำเครื่องหมายด้วย RGS13 การแสดงออก 1 ใน 10 ยีนที่ได้รับการควบคุมสูงสุดใน TOF-DG-คลัสเตอร์ B0 (รูปที่. 1E) ถูกพบอย่างสม่ำเสมอในเซลล์ที่ได้รับ TOF-DG ในช่วงเวลาที่มีลำดับทั้งหมด (D7, D8 และ D11) (รูปที่ 3E) แต่ไม่ใช่ใน TOF-ND1 และกลุ่มควบคุม
การแสดงออกของยีนนอกมดลูกได้รับการรายงานเมื่อเร็ว ๆ นี้ในต้นกำเนิดจาก Tbx1 เงื่อนไขเป็นโมฆะ (Tbx1-cKO) เมาส์35. เพื่อเปรียบเทียบข้อมูล hiPSC-CP และข้อมูล hiPSC-CM ที่ยังไม่บรรลุนิติภาวะของเรากับ Tbx1-เมาส์ cKO เราทำการเปรียบเทียบข้ามสายพันธุ์กับแพ็คเกจ R SingleCellNet36. มีการระบุกลุ่มสิบสี่กลุ่มจาก Tbx1ชุดข้อมูลเมาส์ -cKO (รูปที่ XNUMX เพิ่มเติม) 5A) ที่มีการแสดงออกของยีนที่รายงานในสิ่งพิมพ์35 (รูปที่เสริม 5B) ซึ่งรวมถึงต้นกำเนิดของหัวใจที่แตกต่างกันสี่ชนิด (ต้นกำเนิดหลายสายพันธุ์, MLP; SHF ล่วงหน้า, aSHF; SHF หลัง, pSHF และโพรเอพิคาร์เดียม; PEO) และคาร์ดิโอไมโอไซต์ D7-hiPSC-CPs ของเราส่วนใหญ่คล้ายกับ aSHF ในขณะที่ D11-hiPSC-immature cardiomyocytes ส่วนใหญ่คล้ายกับ cardiomyocytes (รูปที่ XNUMX เพิ่มเติม) 5C) ใน Tbx1-ชุดข้อมูลเมาส์ cKO พักซ์8ซึ่งเป็นยีนนอกมดลูกที่สำคัญที่รายงานโดย Nomaru และคณะ35พบเฉพาะใน MLP และต้นกำเนิดของปอด แต่ไม่พบใน aSHF และ cardiomyocytes (รูปที่ XNUMX เพิ่มเติม) 5D). เราไม่ได้ระบุ พักซ์ 8 การแสดงออกในชุดข้อมูล D7-D11 ของเรา อีกด้วย, RGS13ยีนนอกมดลูกที่พบในชุดข้อมูล D7-D11 ของเรา ไม่ได้ถูกระบุในชุดข้อมูล Tbx1-เมาส์ซีเคโอ กล่าวอีกนัยหนึ่งในขณะที่พบการแสดงออกของยีนนอกมดลูกในทั้งสองอย่าง Tbx1-เมาส์ cKO และ DG-hiPSC-CPs/CMs ของเรา ตัวตนและประชากรที่แสดงยีนนอกมดลูกนั้นแตกต่างกัน
เปลี่ยนแปลงพารามิเตอร์ทางไฟฟ้าสรีรวิทยาและเพิ่มภาวะหัวใจเต้นผิดจังหวะใน TOF-DG-hiPSC-CMs
นอกเหนือจากการทำโปรไฟล์การถอดเสียงแล้ว เรายังสำรวจสรีรวิทยาไฟฟ้าและภาวะผิดปกติของ hiPSC-CM ที่แตกต่างกันโดยใช้แพลตฟอร์ม hCAS ของเราเพื่อให้การประเมิน hiPSC-CM ที่ครอบคลุมมากขึ้น พารามิเตอร์ทางอิเล็กโทรสรีรวิทยา รวมถึงศักยะงาน (AP) การจัดการกับแคลเซียม และระยะเวลาการทนไฟที่มีประสิทธิผล (ERP) ถูกวัดภายใต้การกำหนดจังหวะทางไฟฟ้าที่ 1 เฮิร์ตซ์
เมื่อเปรียบเทียบกับสารควบคุมทั้งสอง TOF-DG2-hCAS แสดงระยะเวลา AP ที่สั้นลงอย่างมีนัยสำคัญ (50 และ 90% สู่การเกิดรีโพลาไรซ์, APD50 และ APD90) (รูปที่ 4A). ในทำนองเดียวกัน เมื่อเปรียบเทียบกับสารควบคุมทั้งสอง TOF-DG2-hCAS แสดงการสั้นลงที่มีนัยสำคัญของเวลาและเวลาในการสลาย AP ขึ้น (50 และ 90% จากพีค) (รูปที่ 4A). ดังนั้น คุณลักษณะ AP จึงแตกต่างกันอย่างมีนัยสำคัญระหว่าง TOF-DG2 และส่วนควบคุม
A คุณลักษณะ Dotplot of Action Potential (AP) ระยะเวลาที่เป็นไปได้ของการดำเนินการ APD B Dotplots ของคุณลักษณะแคลเซียมชั่วคราว (CaT) C Dotplot ของระยะเวลาทนไฟที่มีประสิทธิภาพ ข้อมูลถูกนำเสนอในค่าเฉลี่ย ± SD สำหรับรูปที่ 1A-C. การทดสอบทางสถิติ: การวิเคราะห์ความแปรปรวนทางเดียวแบบธรรมดาตามด้วยการทดสอบการเปรียบเทียบหลายรายการของ Tukey (APD50, AP90%Decay, CaT 50%Decay, CaT Up stroke และระยะเวลาทนไฟที่มีประสิทธิภาพ) การทดสอบ Kruskal–Wallis ตามด้วยการทดสอบเปรียบเทียบพหุคูณของ Dunn (APD90, AP up stroke, AP50%Decay และ CaT 90%Decay) การค้นพบที่สำคัญระหว่าง TOF(DG/ND) และกลุ่มควบคุมถูกทำเครื่องหมายด้วยเครื่องหมายดอกจัน * P <0.05; ** P <0.01; *** P < 0.001; **** P <0.0001. D เปอร์เซ็นต์ของเหตุการณ์การกลับเต้นผิดจังหวะของ TOF (DG/ND) และการควบคุม hCAS ระหว่างการกำหนดจังหวะในสภาวะคงตัว (SteadyState) และการกระตุ้นด้วยไฟฟ้าที่ตั้งโปรแกรมไว้ (PES) การทดสอบทางสถิติ: การทดสอบที่แน่นอนของฟิชเชอร์ *** P <0.001. E การติดตามศักยภาพในการดำเนินการของตัวแทน (AP) และแผนที่ไอโซโครนของเหตุการณ์การกลับเข้ามาใหม่ระหว่าง PES จาก TOF-DG2 แผงด้านบนแสดงการติดตาม AP ที่เป็นตัวแทนระหว่าง PES (S1S2) ตามด้วยอุบัติการณ์ของเหตุการณ์การกลับเข้าจังหวะอีกครั้ง แผงด้านล่างแสดงแผนที่ไอโซโครนสองแผนที่ซึ่งสอดคล้องกับ AP ปกติ (ซ้าย) และ AP จังหวะ (ขวา) F การลดลงของยีนหัวใจในคลัสเตอร์ B0-TOF-DG2 การวิเคราะห์การเพิ่มคุณค่า GO ถูกดำเนินการบนการเปรียบเทียบระหว่างคลัสเตอร์ B0-TOF-DG2 และคลัสเตอร์ B0-TOF-DG1 คำศัพท์ GO ที่ได้รับการเสริมสมรรถนะในยีนที่ควบคุมต่ำจะแสดงในตาราง (ซ้าย) และยีนที่เกี่ยวข้องจะแสดงในพล็อตไวโอลิน (ขวา)
การจัดการแคลเซียมได้รับการประเมินด้วยเวลาและเวลาในการสลายแคลเซียมชั่วคราว (CaT) ขึ้นไป (50 และ 90% จากจุดสูงสุด) พบการลดระยะเวลาการเคลื่อนตัวของ CaT ลงอย่างมีนัยสำคัญใน TOF-DG2-hCAS ในขณะที่การลดระยะเวลาลงอย่างมีนัยสำคัญในการสลายตัว (50% จากจุดสูงสุด) พบในทั้ง TOF-DG2-hCAS และ TOF-ND1-hCAS เมื่อเปรียบเทียบกับทั้งสองตัวควบคุม ( รูปที่. 4B).
เราประเมิน ERP ของ hiPSC-CM เพิ่มเติม ซึ่งการทำให้สั้นลงซึ่งมีแนวโน้มที่จะเกิดภาวะหัวใจเต้นผิดจังหวะ พบการหดตัวของ EPR อย่างมีนัยสำคัญใน TOF-DG2-hCAS ในขณะที่พบการยืดตัวของ ERP ใน TOF-ND2-hCAS (รูปที่. 4C). ที่สำคัญ TOF-DG2-hCAS แสดงอุบัติการณ์ที่สูงขึ้นอย่างมีนัยสำคัญของภาวะหัวใจเต้นผิดจังหวะซ้ำในระหว่างการกำหนดจังหวะทางไฟฟ้าที่ตั้งโปรแกรมไว้ (PES) (รูปที่ 4D). การติดตาม AP ที่เป็นตัวแทนพร้อมแผนที่ไอโซโครนของภาวะหัวใจเต้นผิดจังหวะกลับเข้ามาใหม่จาก TOF-DG2-hCAS ถูกแสดงไว้ในรูปที่ 4E.
ในขณะที่ TOF-DG2-hCAS แสดงระยะเวลา AP ที่สั้นลงอย่างมีนัยสำคัญ, เวลาและเวลาในการสลายของ CaT ที่เพิ่มขึ้น และ ERP, TOF-DG1-hCAS มีความคล้ายคลึงกับการควบคุมในแง่ของพารามิเตอร์ทางอิเล็กโตรฟิสิกส์วิทยาที่ประเมิน (รูปที่. 4A–D). ดังนั้นเราจึงสำรวจเพิ่มเติมถึงความแตกต่างในการถอดเสียงระหว่าง TOF-DG1 และ TOF-DG2 ในคลัสเตอร์ B0 เมื่อเปรียบเทียบกับคลัสเตอร์ B0-TOF-DG1 คลัสเตอร์ B0-TOF-DG2 แสดงการลดลงของยีนหัวใจที่เกี่ยวข้องกับการหดตัวของกล้ามเนื้อ (รูปที่. 4F). ในขณะที่ยีนการเต้นของหัวใจเหล่านี้ถูกลดการควบคุมลงในทั้งคลัสเตอร์ B0-TOF-DG1 และคลัสเตอร์ B0-TOF- DG2 เมื่อเปรียบเทียบกับคลัสเตอร์อื่น (B1, B2 และ B3) (รูปที่. 4F) ขอบเขตการปรับลดของพวกมันโดดเด่นมากขึ้นในคลัสเตอร์ B0-TOF-DG2 (รูปที่ 4F) นอกจากนี้ พีเคพี2มีรายงานว่าการกลายพันธุ์มีความเกี่ยวข้องกับภาวะ RV dysplasia ที่เกิดจากภาวะหัวใจเต้นผิดจังหวะ37พบว่าเป็นหนึ่งในยีนหัวใจที่มีการควบคุมลดลงในกลุ่ม B0-TOF-DG2 (รูปที่ 4F). ขอบเขตที่แตกต่างกันของการลดลงของยีนหัวใจอาจเป็นสาเหตุให้เกิดความคลาดเคลื่อนในการทำงานที่สังเกตได้ในกลุ่มเซลล์จากผู้ป่วยหลายรายในแพลตฟอร์ม hCAS
- เนื้อหาที่ขับเคลื่อนด้วย SEO และการเผยแพร่ประชาสัมพันธ์ รับการขยายวันนี้
- PlatoData.Network Vertical Generative Ai เพิ่มพลังให้กับตัวเอง เข้าถึงได้ที่นี่.
- เพลโตไอสตรีม. Web3 อัจฉริยะ ขยายความรู้ เข้าถึงได้ที่นี่.
- เพลโตESG. คาร์บอน, คลีนเทค, พลังงาน, สิ่งแวดล้อม แสงอาทิตย์, การจัดการของเสีย. เข้าถึงได้ที่นี่.
- เพลโตสุขภาพ เทคโนโลยีชีวภาพและข่าวกรองการทดลองทางคลินิก เข้าถึงได้ที่นี่.
- ที่มา: https://www.nature.com/articles/s42003-023-05344-6