Integrazione della trascrizione inversa in caPCR
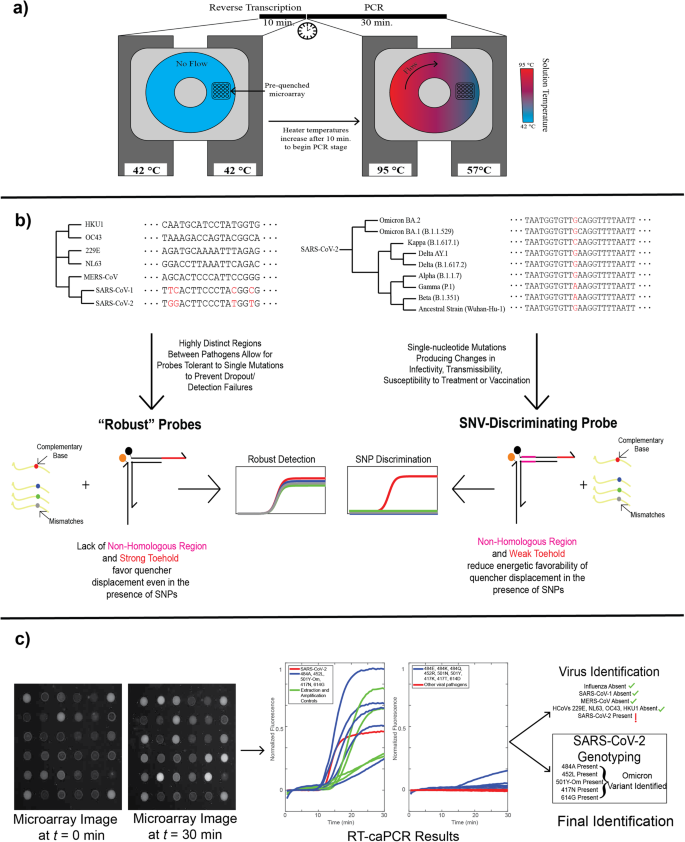
Per prima cosa abbiamo introdotto una fase RT nel test caPCR standard. La caPCR viene eseguita utilizzando un set di 4 riscaldatori che si fissano su entrambe le facce di una cartuccia contenente una camera toroidale riempita con la miscela di reazione PCR4. Impostando i riscaldatori su un lato della camera alla temperatura di ricottura della PCR e sull'altro alla temperatura di denaturazione, si stabilisce un gradiente, producendo così un flusso convettivo passivo che guida l'amplificazione della PCR. L'array di sonde pre-quenched è immobilizzato sulla superficie della cartuccia e risponde tramite TMSD mentre vengono sintetizzati prodotti PCR specifici. Per accogliere una fase iniziale di trascrizione inversa, abbiamo programmato i riscaldatori per mantenere una temperatura costante di 42 °C per la sintesi del cDNA per 10 minuti, dopodiché i riscaldatori si regolano rispettivamente a 57 °C e 95 °C per avviare il flusso convettivo (Fig. 2a). Per ridurre il numero di passaggi di gestione manuale nel flusso di lavoro viene utilizzato un approccio RT-PCR one-pot. Il rilevamento mediato dalla sonda Toehold sulla piattaforma caPCR funziona in modo ottimale in condizioni di PCR asimmetriche: un eccesso del primer che produce il filamento che sposta il quencher genera un segnale più forte sull'array di sonde fluorescenti rispetto a un rapporto di primer forward-to-reverse 1:1 (Fico. S1). Ci siamo assicurati che questo stesso primer media anche la sintesi del cDNA dai target dell'RNA per evitare la necessità di introdurre primer separati per le fasi di trascrizione inversa e PCR.
a Il flusso di lavoro caPCR può essere esteso per includere una fase di trascrizione inversa (RT-caPCR) attraverso l'aggiunta di un enzima di trascrittasi inversa e una fase RT dedicata con tutti i riscaldatori del dispositivo impostati sulla temperatura RT (42 °C). Le temperature del riscaldatore vengono quindi aumentate a temperature diverse per la denaturazione (95 °C) e la ricottura (57 °C), inducendo il flusso convettivo e l'amplificazione PCR dei modelli di cDNA. b Lo schema di progettazione energetica delle sonde, basato sulla termodinamica del legame del DNA, può essere utilizzato per soddisfare due esigenze nella diagnostica delle malattie infettive: I) Il rilevamento dei patogeni virali dovrebbe essere resistente a piccoli cambiamenti nella sequenza nucleotidica per prevenire l'abbandono se si presentano nuove varianti. II) La discriminazione delle varianti richiede la risoluzione di un singolo nucleotide. La variazione delle energie dell'appoggio, del dominio e della regione non omologa (NHR) può essere utilizzata per soddisfare entrambi questi requisiti. c Esempio di output di dati da una corsa RT-caPCR a ciambella. Le immagini dell'array di sonde vengono acquisite ogni 30 secondi per 30 minuti di caPCR. Le tracce di fluorescenza vengono estratte e analizzate per identificare quali agenti patogeni respiratori sono presenti e, se applicabile, quale variante di SARS-CoV-2 è presente.
Per sviluppare sonde sintonizzabili con selettività di sequenza programmabile, che vanno da sensibili alla mutazione a tolleranti alla mutazione, abbiamo utilizzato 2 serie di criteri energetici per le sonde di punta (Fig. 2b). Le sonde robuste presentano una regione di punta più forte e un dominio corrispondentemente più debole per ridurre l'influenza complessiva di una singola mutazione sull'inizio della reazione di legame e sul successivo spostamento del braccio del quencher. Per la discriminazione SNV, il punto d'appoggio viene ridotto in lunghezza e viene introdotta una regione non omologa (NHR) priva di complementarità con il bersaglio. Questa combinazione serve a ridurre la preferenza energetica complessiva per il legame del puntale e lo spostamento del braccio del quencher, fornendo così un peso extra ai singoli disallineamenti nella sequenza target.
Abbiamo applicato la RT-caPCR con sonde robuste e discriminanti per SNV al rilevamento dei virus respiratori. Le immagini dell'array di sonde dal sistema RT-caPCR vengono elaborate utilizzando uno script MATLAB personalizzato per produrre tracce di fluorescenza che vengono poi interpretate per identificare sia i target virali presenti che, se applicabile, la variante di SARS-CoV-2. Un esempio è mostrato per la variante SARS-CoV-2 Omicron in Fig. 2c. Pertanto, il nostro test RT-caPCR one-pot dimostra la sua capacità di eseguire simultaneamente sia il rilevamento sensibile con sonde robuste sia la risoluzione di un singolo nucleotide con sonde discriminatorie SNV per identificare accuratamente i target di RNA di interesse in una singola reazione.
Ottimizzazione dell'energetica tra sonde robuste e discriminanti SNV
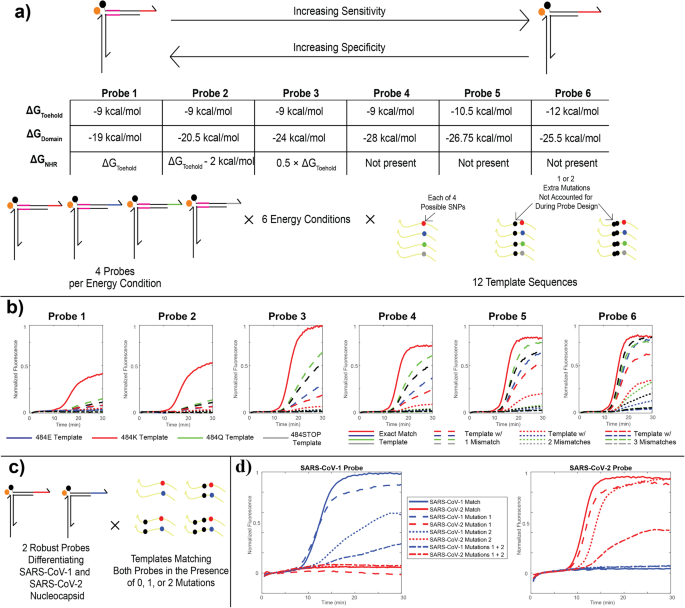
Per aumentare la diversità dei bersagli rilevati nel nostro test RT-caPCR, abbiamo ottimizzato l'energia del legame del puntale e dello spostamento del filamento per modulare il compromesso sensibilità-specificità. Come caso di studio, abbiamo utilizzato il genotipo SARS-CoV-2 “484E” come modello per la nostra valutazione energetica. Le mutazioni in questo sito sono associate ad una ridotta attività neutralizzante da parte degli anticorpi dell'ospite12e 3 delle 4 possibili basi nucleotidiche (adenina, guanina e citosina) si trovano nella prima posizione del codone nelle varianti SARS-CoV-2 naturalmente evolute9. La sostituzione dell'uracile in questo sito produrrebbe un codone di stop e quindi non è stata osservata in natura, ma per rigore abbiamo incluso una condizione di timina nella nostra valutazione. Abbiamo progettato una sonda per ciascuno dei 4 possibili nucleotidi in questo locus e stabilito 6 diverse condizioni termodinamiche basate sull'energia di legame (ΔGo) delle 3 regioni della sonda di appoggio (NHR, dominio e appoggio; Fig. 3a, vedere Materiali e metodi). Queste condizioni energetiche vanno dalla più specifica (Sonda 1), in cui l'energia di NHR e punto d'appoggio è approssimativamente uguale (circa −9 kcal/mol), alla più sensibile (Sonda 6), in cui non c'è NHR e l'appoggio è leggermente più forte (-12 kcal/mol). In tutti i casi, l'energia di legame del dominio viene regolata per mantenere costante l'energia di legame della sonda complessiva a circa −40 kcal/mol.
a Parametri di progetto per la caratterizzazione energetica delle sonde. Sei diversi schemi energetici sono stati progettati variando le energie di legame delle regioni di punta, dominio e NHR, per consentire un compromesso tra specificità del singolo nucleotide e sensibilità sotto forma di robustezza alle mutazioni. Sono state progettate quattro diverse sonde per ciascuno dei sei progetti energetici, coprendo tutti e quattro i possibili codoni nel sito di mutazione 484 nella proteina spike SARS-CoV-2, tre dei quali sono stati osservati in varianti. Ciascuna sonda è stata testata con 12 diversi modelli plasmidici, comprese le 4 uniche mutazioni a singolo nucleotide da sole e in presenza di una o due altre mutazioni trovate più a monte nella variante Omicron. b Risultati caPCR dalla serie di sonde 484 K. Le curve continue rosse indicano le prestazioni in linea con l'obiettivo senza mutazioni. Le linee tratteggiate, punteggiate e tratteggiate indicano rispettivamente modelli con 1, 2 o 3 disallineamenti relativi alla sequenza della sonda. Il colore specifica il codone nel sito 484 (E, K, Q o STOP). c Schema per testare una solida discriminazione SARS-CoV-1/2. Due sonde sono state progettate secondo lo schema energetico “Sonda 5” in parte a per una regione del gene del nucleocapside in SARS-CoV-1 e SARS-CoV-2. Entrambe le sonde sono state testate in presenza del modello di corrispondenza esatta e delle varianti comprendenti uno o entrambi i due disallineamenti a monte, nonché in presenza del modello di corrispondenza esatta e delle varianti non corrispondenti dell'altra sonda. d Risultati dei test descritti in (c). Le linee continue, tratteggiate, punteggiate e tratteggiate indicano modelli di corrispondenza esatta, modelli con uno dei due SNV o modelli con entrambi gli SNV relativi al genoma SARS-CoV-1 (blu) o SARS-CoV-2 (rosso).
Abbiamo testato queste sonde con 12 diversi modelli di DNA, 4 dei quali rappresentano il modello di corrispondenza esatta per ciascuna delle 4 possibili basi nel codone 484. Per dimostrare la sintonizzabilità dell'energia della nostra sonda, abbiamo preso mutazioni emerse nei lignaggi successivi dell'evoluzione SARS-CoV-2 e le abbiamo introdotte nei modelli: la mutazione T478K trovata nei lignaggi Delta AY e Omicron e la mutazione S477N trovata in Omicron lignaggi (schema Fig. 3a)10. Tutte le 24 sonde sono state valutate in risposta all'amplificazione di ciascuno dei 12 template. Figura 3b mostra un sottoinsieme di questi risultati, in particolare per le sonde progettate per individuare la mutazione 484 K riscontrata nei COV Beta e Gamma10. Man mano che la forza dell'NHR si riduce e l'appoggio aumenta, il rilevamento del modello di corrispondenza esatta (linea rossa continua) rimane forte e la sonda diventa più sensibile ai modelli con 1 (linee tratteggiate) e 2 (linee tratteggiate) non corrispondenti. Ciò dimostra la nostra capacità di progettare l’energia delle sonde per risultati molto specifici che vincolano il bersaglio. Sulla base di questi risultati, abbiamo selezionato lo schema energetico 2 per le sonde discriminanti SNV e lo schema 5 per le sonde robuste.
Abbiamo progettato 2 robuste sonde mirate alla sequenza del gene del nucleocapside di SARS-CoV-1 e SARS-CoV-2. Per garantire che queste sequenze di agenti patogeni potessero essere differenziate utilizzando sonde robuste pur rimanendo tolleranti alle mutazioni, abbiamo ideato 8 modelli contenenti nessun disadattamento, 1 di 2 disallineamenti o entrambi i disallineamenti insieme (Fig. 3c, vedere Materiali e metodi). La regione selezionata per il rilevamento di SARS-CoV-1 e SARS-CoV-2 differisce tra le 2 sequenze target di 8 mutazioni: pertanto, le discrepanze nel modello sono state selezionate in base a queste differenze. Pertanto, il modello SARS-CoV-2 con 2 disallineamenti presenta solo 6 differenze rispetto alla sequenza SARS-CoV-1, evidenziando così un’applicazione nella vita reale dell’importanza della precisa sintonizzazione delle sonde per sensibilità rispetto a specificità. Figura 3d mostra i risultati di questi test. In tutti i casi, non abbiamo osservato alcuna reattività crociata tra le sonde e i modelli SARS-CoV-1 e SARS-CoV-2: l’RNA di SARS-CoV-1 è stato rilevato solo dalla sonda SARS-CoV-1 e SARS-CoV-2 L’RNA è stato rilevato solo dalla sonda SARS-CoV-2. I modelli contenenti una delle 2 mutazioni possono anche essere rilevati in modo inequivocabile da ciascuna sonda appropriata, con 1 delle 2 mutazioni che influiscono più chiaramente sulle prestazioni rispetto all'altra. Allo stesso modo, i modelli con entrambe le mutazioni insieme vengono ancora rilevati sebbene le prestazioni siano leggermente compromesse a causa della minore preferenza energetica nello spostamento del braccio del quencher.
Attraverso i nostri studi mirati sull'energia delle sonde, abbiamo dimostrato la nostra capacità di progettare con precisione le sonde per ottenere il compromesso desiderato tra sensibilità e specificità. Le sonde discriminatorie SNV hanno componenti energetici che richiedono una corrispondenza esatta per lo spostamento del braccio quencher, mentre le sonde robuste sono più promiscue nel legame dei bersagli con la capacità di rilevare un amplicone contenente fino a due mutazioni. L'intervallo energetico che abbiamo esplorato può essere applicato per regolare le sonde secondo necessità dallo schema energetico 1 allo schema energetico 6 per consentire il rilevamento di target con il numero desiderato di discrepanze dalla sequenza della sonda.
Prestazioni di sonde robuste e discriminatorie SNV
Per dimostrare la prova di concetto del nostro design di sonda altamente personalizzabile, abbiamo creato un pannello completo di sonde robuste e discriminatorie SNV. Il nostro panel evidenzia l’utilità di sonde robuste effettuando lo screening di tutti e 7 i coronavirus noti per infettare l’uomo (HCoV 229E, HKU1, NL63 e OC43; MERS-CoV, SARS-CoV-1 e SARS-CoV-2) nonché dell’influenza A e B8. Inoltre, il nostro pannello dimostra la funzionalità delle sonde discriminatorie SNV utilizzando 14 sonde e 3 ampliconi che coprono 5 siti comunemente mutati nel gene del picco SARS-CoV-2 per consentire la differenziazione tra le varianti di interesse (417, 452, 484, 501 e 614; fig. S2)9,10,11.
Per valutare le prestazioni delle robuste sonde nel nostro test RT-caPCR per il rilevamento di target sensibili, abbiamo eseguito 65 test RT-caPCR utilizzando campioni di mezzi di trasporto virale (VTM) negativi al coronavirus che coprivano una gamma di dati demografici dei pazienti (Tabelle S1-S4) arricchito con RNA sintetico da ciascuno dei 7 coronavirus umani, acquistato da Twist Bioscience o sintetizzato internamente utilizzando la trascrizione in vitro (IVT) da frammenti di geni (Tabella S5). L'RNA è stato isolato e purificato da questi campioni clinici artificiali utilizzando un metodo di estrazione basato su perline13 a causa della precedenza nell’utilizzo di tale metodologia per l’estrazione dell’RNA nei flussi di lavoro diagnostici SARS-CoV-214 ed è stato utilizzato come input per il nostro sistema. Questo test includeva anche sia un controllo di estrazione (RNA del batteriofago MS2 aggiunto al campione artificiale prima della purificazione) sia un controllo di amplificazione (RNA del controllo positivo interno (IPC) aggiunto alla miscela di reazione RT-caPCR). Se non diversamente specificato, gli esperimenti qui ne hanno ricevuti circa 106 copie di RNA MS2 e 107 copie di RNA virale. La reazione RT-caPCR è stata eseguita in 40 minuti utilizzando 5,000 molecole di RNA di controllo IPC, l'RNA del campione estratto e 11 coppie di primer che coprivano tutti i target e i controlli.
I dati RT-caPCR sono stati analizzati per identificare i bersagli virali presenti in ciascun campione e, nel caso di SARS-CoV-2, quale variante specifica era presente (Fig. 4). figura 4a mostra una corsa rappresentativa con la variante SARS-CoV-2 Omicron come bersaglio dell'RNA. Tutte le sonde robuste per la discriminazione virale sono mostrate in rosso mentre le sonde discriminanti per SNV sono mostrate in blu. I controlli di estrazione e amplificazione sono mostrati in verde. Gli spot della sonda positiva e negativa corrispondono a sonde completamente non estinte o completamente estinte che utilizzano sequenze per le quali non sono stati inclusi primer né modello e sono mostrate in grigio. Questo tipo di dati possono essere riassunti in un grafico compresso come mostrato in Fig. 4b: tutte le curve positive attese sono rappresentate nel grafico a sinistra, mentre tutte le curve negative attese sono mostrate nel grafico a destra. Sebbene sia presente un piccolo livello di attività fuori target su alcune sonde specifiche per SNV, confrontando le curve delle sonde specifiche per SNV sul target (484 A, 452 L, 501Y-Om, 417 N e 614 G) con altre sonde SNV- sonde specifiche confermano che le sonde sul target rispondono in modo più forte in tutti i casi, come valutato a occhio e attraverso il nostro algoritmo di discriminazione SNV basato su derivati di fluorescenza (vedere Materiali e metodi e Metodi supplementari). Notiamo che, a causa dell'elevato carico mutazionale della variante Omicron rispetto ad altre varianti11, abbiamo progettato la sequenza della sonda 484 A in modo da includere anche le mutazioni S477N e T478K trovate in questa variante per migliorare il rilevamento10. Allo stesso modo, una sonda 501Y-Om unica include le mutazioni Q493R, G496S e Q498R che si trovano specificamente nella variante Omicron. Successivamente è stata introdotta una sonda specifica per la sottovariante BA.2 del lignaggio Omicron (501Y-Om2). Questa sonda è identica alla sequenza 501Y-Om ma manca della mutazione G496S10.
a Risultati di esempio di un'analisi RT-caPCR che rileva la variante preoccupante di SARS-CoV-2 Omicron. Le sonde di rilevamento virale robuste sono in rosso, le sonde discriminanti SNV sono in blu, i controlli di estrazione e amplificazione sono in verde e i controlli positivi e negativi sono in grigio. Ciascuna curva rappresenta l'intensità del punto corrispondente dell'array di sonde in ciascun punto temporale sulle 60 immagini acquisite, normalizzate al punto più luminoso dell'array. b Risultati riassuntivi corrispondenti al rodaggio a. Tutti i positivi attesi (controlli di estrazione e amplificazione, identificazione virale sul target e tutte le sonde discriminanti SNV sul target) sono mostrati nel grafico a sinistra, mentre tutte le sonde negative previste (tutte le altre sonde virali e discriminanti SNV, come così come i controlli negativi) sono mostrati nel grafico a destra. c Risultati del rilevamento virale per il coronavirus umano 229E, il coronavirus della sindrome respiratoria del Medio Oriente (MERS-CoV) e SARS-CoV-1 nel formato descritto in (b). d. La discriminazione SNV risulta da un modello di variante SARS-CoV-2 Delta nel formato sopra specificato.
figura 4c mostra risultati rappresentativi compressi di 3 analisi per 3 diversi modelli di agenti patogeni virali: coronavirus umano 229E, MERS-CoV e SARS-CoV-1. Tutti e 3 i campioni mostrano un chiaro rilevamento dell'RNA virale bersaglio con le robuste sonde di identificazione del patogeno, nessuna attività della sonda di identificazione virale fuori bersaglio e la presenza di tutti i controlli.
Table 1 riassume i risultati dell’identificazione virale di tutti gli studi validi. La competizione per le risorse di reazione della PCR (ad esempio, polimerasi, dNTP) può sopprimere l'amplificazione dei target con numero di copie inferiore, quindi un'analisi è stata considerata valida se è stato rilevato un patogeno virale e almeno 1 dei 2 controlli (MS2 e IPC) oppure non è stato rilevato alcun agente patogeno virale ma sono stati rilevati entrambi i controlli. 11 campioni erano controlli negativi, che includevano solo RNA di controllo MS2 e IPC. In tutti gli 11 campioni sono stati rilevati sia IPC che MS2 (100%). Sono stati identificati 36 campioni su 36 di SARS-CoV-2 (100%) valutati mediante rilevamento della sonda nucleocapside SARS-CoV-2. Anche i restanti coronavirus umani (229E, NL63, OC43 e HKU1) nonché i target SARS-CoV-1 e MERS-CoV mostrano un’elevata sensibilità, con un rilevamento del 100% per tutti i target. Sia il processo di legame dei primer che lo spostamento del quencher mediato dagli ampliconi delle sonde contribuiscono alla specificità del pannello, che è stata del 100% per tutti i target. In questo caso, la specificità viene calcolata come la proporzione dei veri negativi rilevati correttamente. In breve, nessun modello ha indotto il rilevamento su una sonda robusta diversa da quella per la quale è stata specificamente progettata.
figura 4d mostra un'esecuzione rappresentativa che evidenzia la capacità del nostro test RT-caPCR sia di identificare il bersaglio dell'RNA sia di fornire una risoluzione a singolo nucleotide mostrando il rilevamento della variante virale contemporaneamente all'identificazione del patogeno per la variante Delta SARS-CoV-2. Sebbene la sonda 452 L mostri una significativa attività fuori bersaglio, il suo rilevamento è chiaramente meno efficiente rispetto alla sonda 452 R sul bersaglio (la curva blu con la massima intensità nel grafico a sinistra). Pertanto, le sonde discriminanti SNV consentono una chiara distinzione tra le varianti SARS-CoV-2 che differiscono solo per poche singole mutazioni.
Table 2 riassume le prestazioni delle sonde specifiche per SNV per le varianti SARS-CoV-2 in 36 studi. Il numero di rilevamenti attesi è uguale al numero di analisi per le quali è stato incluso un modello che mostra quella mutazione, mentre il numero di negativi attesi è il numero di analisi per le quali non è stato incluso il modello corrispondente. I veri positivi sono le corse per le quali la sonda di interesse era la sonda specifica del SNV dominante per quel sito di mutazione (come valutato algoritmicamente), mentre i veri negativi sono quelle corse per le quali non lo era. In un'analisi, notiamo che la sonda 614 G era oscurata da una particella estranea (polvere), quindi il totale di questi target è 35, anziché 36. A parte 484E, tutte le sonde mostrano almeno il 90% di sensibilità. Le scarse prestazioni della sonda 484E possono essere parzialmente spiegate dalla presenza di un SNV (mutazione T478K) nell'amplicone delle varianti Delta. Tutte le altre sonde mostrano una specificità del 100%, poiché un amplicone sposterà sempre preferenzialmente il suo quencher corrispondente rispetto a uno con una mutazione, presupponendo che i valori energetici sul bersaglio siano uguali.
Insieme, questi dati evidenziano la capacità delle sonde robuste e delle sonde discriminatorie SNV di lavorare in tandem per identificare rapidamente il bersaglio dell'RNA presente (con la sonda robusta) e fornire una risoluzione a singolo nucleotide (con la sonda discriminatoria SNV).
Valutazione del limite di rilevamento di RT-caPCR
Successivamente abbiamo eseguito una serie di esperimenti per determinare il limite di rilevamento del nostro test RT-caPCR. In tutti i test precedenti in Fig. 4, un input virale di 107 molecole sono state aggiunte al VTM prima dell'estrazione, in modo simile alle stime della carica virale di picco per i pazienti Omicron e Delta15,16. Qui abbiamo quantificato il LOD del nostro test variando il numero di copie di input dell'RNA virale e mantenendo le stesse copie di controllo. Utilizzando RNA purificato ottenuto da Twist Bioscience o sintetizzato internamente da frammenti di geni tramite IVT, abbiamo raggiunto un LOD di 1,000 molecole per tutti e 7 i coronavirus. I risultati rappresentativi sono mostrati in Fig. S3. In entrambe le Fig. S3 bis ed S3b, scopriamo che possiamo rilevare con successo fino a 1,000 molecole per reazione per un singolo target con entrambi i controlli (HCoV 229E; Fig. S3 bis) e per una variante SARS-CoV-2 con entrambi i controlli (Omicron; Fig. S3b), una quantità 3 ordini di grandezza inferiore a quella utilizzata per i test VTM quando si utilizza RNA purificato. Quando viene variata solo la presenza di un singolo bersaglio, come in Fig. S3 bis, troviamo un chiaro ritardo nel tempo soglia per ciascun ordine di grandezza solo per questo obiettivo, mentre i 2 controlli sono coerenti tra le esecuzioni. Questa tendenza è meno chiara per SARS-CoV-2, cosa prevedibile dato che questo target richiede l’amplificazione simultanea di 4 ampliconi e 2 controlli. La competizione per le risorse enzimatiche e i dNTP sembra interrompere la chiara tendenza osservata per i target a singolo amplicone. Nella fig. S3C, è stata eseguita una serie di diluizioni variando la quantità di RNA immesso nel condotto di estrazione per la variante Omicron. Anche in questo caso vediamo un rilevamento fino al valore più basso testato (ingresso di 10,000 molecole nella fase di estrazione dell'RNA con circa 1,000 molecole che entrano nella fase RT a causa delle perdite durante l'estrazione e del mancato utilizzo dell'intero volume di eluizione di 25 µL nella reazione) e un ritardo previsto nell'amplificazione in funzione della concentrazione in ingresso.
Rilevamento di più target in un singolo campione
Avendo dimostrato il successo del nostro test RT-caPCR con sonda sintonizzabile nell'identificare target di RNA con sonde robuste (ciascun virus) con specificità a singolo nucleotide con sonde discriminanti SNV (varianti SARS-CoV-2), abbiamo successivamente esplorato come il nostro test si è comportato quando valutare input più complessi simulando campioni di coinfezione contenenti più target di RNA. Qui, un importo di input pari a 105 le molecole di ciascun modello sono state aggiunte direttamente nella reazione RT-caPCR senza passaggi di estrazione VTM. Abbiamo anche ampliato il nostro test RT-caPCR per testare più target di RNA includendo 3 set di primer aggiuntivi e 4 sonde per testare l'influenza A e B (2 mirate al gene della proteina della matrice M1 dell'influenza A17 e 2 mirati alla proteina non strutturale 1 (nsp1) nei lignaggi Yamagata e Victoria dell'influenza B18). Questo test a 14 plex è stato testato per tutti e 4 i nuovi target per confermarne l'efficacia (Fig. S4).
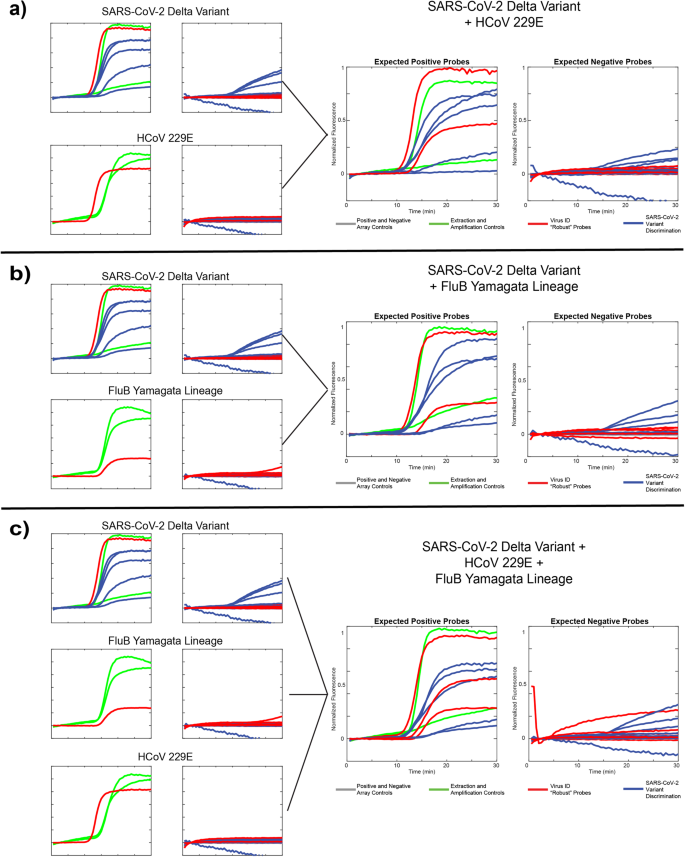
Abbiamo valutato ciascuno dei 3 diversi modelli virali singolarmente e in 3 diverse combinazioni: variante Delta SARS-CoV-2 + HCoV229E (Fig. 5a), variante Delta SARS-CoV-2 + lineage FluB Yamagata (Fig. 5b) e il lignaggio SARS-CoV-2 + HCoV229E + FluB Yamagata (Fig. 5c). I dati vengono presentati come immagini di tracce fluorescenti compresse come descritto sopra con sonde positive attese nel pannello di sinistra e sonde negative attese nel pannello di destra. In tutte le analisi, abbiamo osservato il comportamento previsto: sonde robuste (identificazione del virus) segnalano la presenza di tutti i modelli di agenti patogeni inclusi e sonde discriminanti SNV (identificazione delle varianti) identificano correttamente la variante Delta con bassi livelli di attività della sonda fuori bersaglio in tutte le analisi. 5 siti SNV. La sonda 484E ha le prestazioni più deboli, probabilmente a causa della già citata discrepanza nella regione del dominio di questa sonda. Inoltre, in alcuni esperimenti, il controllo dell'estrazione MS2 ha funzionato male a causa della competizione per le risorse tra 8 diversi ampliconi. Notiamo inoltre che queste analisi includono solo 1 replica di ciascuno dei controlli MS2 e IPC, anziché 2 come in Fig. 4, poiché si trattava di un pannello aggiornato che includeva la sonda 501Y-Om2. Questi dati dimostrano la capacità della nostra tecnologia di valutare accuratamente campioni più complessi contenenti più target di RNA di interesse, dimostrando la versatilità del test.
a Risultati a modello singolo della variante SARS-CoV-2 Delta e del coronavirus umano 229E insieme a un campione di “coinfezione” comprendente entrambi i modelli. b Risultati a modello singolo della variante SARS-CoV-2 Delta e del lignaggio Yamagata dell'influenza B, insieme a un campione di coinfezione comprendente entrambi i modelli. c. Risultati a target singolo dagli stessi target di in a ed b, insieme a un campione di coinfezione comprendente tutti e tre i modelli.
- Distribuzione di contenuti basati su SEO e PR. Ricevi amplificazione oggi.
- PlatoData.Network Generativo verticale Ai. Potenzia te stesso. Accedi qui.
- PlatoAiStream. Intelligenza Web3. Conoscenza amplificata. Accedi qui.
- PlatoneESG. Carbonio, Tecnologia pulita, Energia, Ambiente, Solare, Gestione dei rifiuti. Accedi qui.
- Platone Salute. Intelligence sulle biotecnologie e sulle sperimentazioni cliniche. Accedi qui.
- Fonte: https://www.nature.com/articles/s42003-023-05346-4