इस पोस्ट में, हम प्रदर्शित करते हैं कि प्रोटीन उपकोशिकीय स्थानीयकरण की भविष्यवाणी करने के लिए एक अत्याधुनिक प्रोटीन भाषा मॉडल (पीएलएम) को कुशलतापूर्वक कैसे ठीक किया जाए अमेज़न SageMaker.
प्रोटीन शरीर की आणविक मशीनें हैं, जो आपकी मांसपेशियों को हिलाने से लेकर संक्रमणों पर प्रतिक्रिया करने तक हर चीज के लिए जिम्मेदार हैं। इस विविधता के बावजूद, सभी प्रोटीन अमीनो एसिड नामक अणुओं की दोहराई जाने वाली श्रृंखलाओं से बने होते हैं। मानव जीनोम 20 मानक अमीनो एसिड को एनकोड करता है, जिनमें से प्रत्येक की रासायनिक संरचना थोड़ी भिन्न होती है। इन्हें वर्णमाला के अक्षरों द्वारा दर्शाया जा सकता है, जो हमें टेक्स्ट स्ट्रिंग के रूप में प्रोटीन का विश्लेषण और अन्वेषण करने की अनुमति देता है। प्रोटीन अनुक्रमों और संरचनाओं की विशाल संभावित संख्या ही प्रोटीन को उनके व्यापक प्रकार के उपयोग प्रदान करती है।
प्रोटीन औषधि विकास में, संभावित लक्ष्य के रूप में, बल्कि उपचारात्मक के रूप में भी महत्वपूर्ण भूमिका निभाते हैं। जैसा कि निम्नलिखित तालिका में दिखाया गया है, 2022 में सबसे अधिक बिकने वाली कई दवाएं या तो प्रोटीन (विशेष रूप से एंटीबॉडी) थीं या शरीर में प्रोटीन में अनुवादित एमआरएनए जैसे अन्य अणु थे। इस वजह से, कई जीवन विज्ञान शोधकर्ताओं को प्रोटीन के बारे में प्रश्नों का उत्तर तेजी से, सस्ता और अधिक सटीक रूप से देने की आवश्यकता है।
| नाम | उत्पादक | 2022 वैश्विक बिक्री ($अरबों अमेरिकी डॉलर) | संकेत |
| कोमिरनाटी | फाइजर / BioNTech | $40.8 | COVID -19 |
| स्पाइकवैक्स | आधुनिक | $21.8 | COVID -19 |
| Humira | AbbVie | $21.6 | गठिया, क्रोहन रोग, और अन्य |
| कीट्रेटुडा | मर्क | $21.0 | विभिन्न कैंसर |
डेटा स्रोत: उर्कहार्ट, एल. 2022 में बिक्री के हिसाब से शीर्ष कंपनियां और दवाएं. नेचर रिव्यूज़ ड्रग डिस्कवरी 22, 260-260 (2023)।
क्योंकि हम प्रोटीन को वर्णों के अनुक्रम के रूप में प्रस्तुत कर सकते हैं, हम मूल रूप से लिखित भाषा के लिए विकसित तकनीकों का उपयोग करके उनका विश्लेषण कर सकते हैं। इसमें विशाल डेटासेट पर पहले से प्रशिक्षित बड़े भाषा मॉडल (एलएलएम) शामिल हैं, जिन्हें बाद में पाठ सारांश या चैटबॉट जैसे विशिष्ट कार्यों के लिए अनुकूलित किया जा सकता है। इसी तरह, पीएलएम को बिना लेबल वाले, स्व-पर्यवेक्षित शिक्षण का उपयोग करके बड़े प्रोटीन अनुक्रम डेटाबेस पर पूर्व-प्रशिक्षित किया जाता है। हम उन्हें प्रोटीन की 3डी संरचना या यह अन्य अणुओं के साथ कैसे बातचीत कर सकता है जैसी चीजों की भविष्यवाणी करने के लिए अनुकूलित कर सकते हैं। शोधकर्ताओं ने नए सिरे से नए प्रोटीन को डिजाइन करने के लिए पीएलएम का भी उपयोग किया है। ये उपकरण मानव वैज्ञानिक विशेषज्ञता को प्रतिस्थापित नहीं करते हैं, लेकिन उनमें पूर्व-नैदानिक विकास और परीक्षण डिजाइन को गति देने की क्षमता है।
इन मॉडलों के साथ एक चुनौती उनका आकार है। पिछले कुछ वर्षों में एलएलएम और पीएलएम दोनों में परिमाण के आधार पर वृद्धि हुई है, जैसा कि निम्नलिखित आंकड़े में दिखाया गया है। इसका मतलब यह है कि उन्हें पर्याप्त सटीकता के साथ प्रशिक्षित करने में लंबा समय लग सकता है। इसका मतलब यह भी है कि आपको मॉडल मापदंडों को संग्रहीत करने के लिए बड़ी मात्रा में मेमोरी के साथ हार्डवेयर, विशेष रूप से जीपीयू का उपयोग करने की आवश्यकता है।
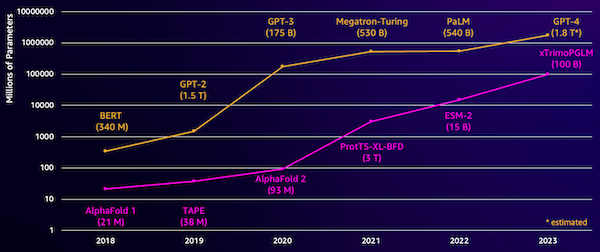
लंबे प्रशिक्षण समय, साथ ही बड़े उदाहरण, उच्च लागत के बराबर होते हैं, जो इस काम को कई शोधकर्ताओं की पहुंच से बाहर कर सकता है। उदाहरण के लिए, 2023 में, ए खोजी दल 100 दिनों के लिए 768 ए100 जीपीयू पर 164 बिलियन-पैरामीटर पीएलएम के प्रशिक्षण का वर्णन किया गया! सौभाग्य से, कई मामलों में हम मौजूदा पीएलएम को अपने विशिष्ट कार्य के लिए अनुकूलित करके समय और संसाधन बचा सकते हैं। इस तकनीक को कहा जाता है फ़ाइन ट्यूनिंग, और हमें अन्य प्रकार के भाषा मॉडलिंग से उन्नत उपकरण उधार लेने की भी अनुमति देता है।
समाधान अवलोकन
इस पोस्ट में हम जिस विशिष्ट समस्या का समाधान कर रहे हैं वह है उपकोशिकीय स्थानीयकरण: प्रोटीन अनुक्रम को देखते हुए, क्या हम एक ऐसा मॉडल बना सकते हैं जो यह अनुमान लगा सके कि यह कोशिका के बाहर (कोशिका झिल्ली) पर रहता है या अंदर? यह जानकारी का एक महत्वपूर्ण हिस्सा है जो हमें कार्य को समझने में मदद कर सकता है और क्या यह एक अच्छा दवा लक्ष्य होगा।
हम एक सार्वजनिक डेटासेट का उपयोग करके डाउनलोड करना शुरू करते हैं अमेज़ॅन सैजमेकर स्टूडियो. फिर हम एक कुशल प्रशिक्षण पद्धति का उपयोग करके ईएसएम-2 प्रोटीन भाषा मॉडल को ठीक करने के लिए सेजमेकर का उपयोग करते हैं। अंत में, हम मॉडल को वास्तविक समय अनुमान समापन बिंदु के रूप में तैनात करते हैं और कुछ ज्ञात प्रोटीनों का परीक्षण करने के लिए इसका उपयोग करते हैं। निम्नलिखित चित्र इस वर्कफ़्लो को दर्शाता है।

निम्नलिखित अनुभागों में, हम आपका प्रशिक्षण डेटा तैयार करने, एक प्रशिक्षण स्क्रिप्ट बनाने और सेजमेकर प्रशिक्षण कार्य चलाने के चरणों के बारे में जानेंगे। इस पोस्ट में प्रदर्शित सभी कोड यहां उपलब्ध हैं GitHub.
प्रशिक्षण डेटा तैयार करें
हम का हिस्सा उपयोग करते हैं डीपलॉक-2 डेटासेट, जिसमें प्रयोगात्मक रूप से निर्धारित स्थानों के साथ कई हजार स्विसप्रोट प्रोटीन शामिल हैं। हम 100-512 अमीनो एसिड के बीच उच्च गुणवत्ता वाले अनुक्रमों को फ़िल्टर करते हैं:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
इसके बाद, हम अनुक्रमों को टोकनाइज़ करते हैं और उन्हें प्रशिक्षण और मूल्यांकन सेटों में विभाजित करते हैं:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
अंत में, हम संसाधित प्रशिक्षण और मूल्यांकन डेटा अपलोड करते हैं अमेज़न सरल भंडारण सेवा (अमेज़न S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)एक प्रशिक्षण स्क्रिप्ट बनाएं
सेजमेकर स्क्रिप्ट मोड आपको AWS द्वारा प्रबंधित अनुकूलित मशीन लर्निंग (ML) फ्रेमवर्क कंटेनर में अपना कस्टम प्रशिक्षण कोड चलाने की अनुमति देता है। इस उदाहरण के लिए, हम एक को अनुकूलित करते हैं पाठ वर्गीकरण के लिए मौजूदा स्क्रिप्ट गले मिलते चेहरे से. यह हमें अपने प्रशिक्षण कार्य की दक्षता में सुधार के लिए कई तरीकों को आजमाने की अनुमति देता है।
विधि 1: भारित प्रशिक्षण वर्ग
कई जैविक डेटासेट की तरह, डीपलॉक डेटा असमान रूप से वितरित किया जाता है, जिसका अर्थ है कि झिल्ली और गैर-झिल्ली प्रोटीन की समान संख्या नहीं है। हम अपने डेटा का पुन: नमूनाकरण कर सकते हैं और बहुसंख्यक वर्ग से रिकॉर्ड हटा सकते हैं। हालाँकि, इससे कुल प्रशिक्षण डेटा कम हो जाएगा और संभावित रूप से हमारी सटीकता को नुकसान पहुँचेगा। इसके बजाय, हम प्रशिक्षण कार्य के दौरान कक्षा के वजन की गणना करते हैं और नुकसान को समायोजित करने के लिए उनका उपयोग करते हैं।
हमारी प्रशिक्षण स्क्रिप्ट में, हम उपवर्ग बनाते हैं Trainer से कक्षा transformers पंजीकरण शुल्क WeightedTrainer वह वर्ग जो क्रॉस-एन्ट्रॉपी हानि की गणना करते समय वर्ग भार को ध्यान में रखता है। इससे हमारे मॉडल में पूर्वाग्रह को रोकने में मदद मिलती है:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossविधि 2: क्रमिक संचय
ग्रेडिएंट संचय एक प्रशिक्षण तकनीक है जो मॉडलों को बड़े बैच आकारों पर प्रशिक्षण अनुकरण करने की अनुमति देती है। आमतौर पर, बैच का आकार (एक प्रशिक्षण चरण में ग्रेडिएंट की गणना करने के लिए उपयोग किए जाने वाले नमूनों की संख्या) GPU मेमोरी क्षमता द्वारा सीमित होता है। ग्रेडिएंट संचय के साथ, मॉडल पहले छोटे बैचों पर ग्रेडिएंट की गणना करता है। फिर, मॉडल वेट को तुरंत अपडेट करने के बजाय, ग्रेडिएंट कई छोटे बैचों में जमा हो जाते हैं। जब संचित ग्रेडिएंट लक्ष्य के बड़े बैच आकार के बराबर होते हैं, तो मॉडल को अद्यतन करने के लिए अनुकूलन चरण निष्पादित किया जाता है। यह मॉडलों को GPU मेमोरी सीमा को पार किए बिना प्रभावी ढंग से बड़े बैचों के साथ प्रशिक्षित करने देता है।
हालाँकि, छोटे बैच के आगे और पीछे के पास के लिए अतिरिक्त गणना की आवश्यकता होती है। ग्रेडिएंट संचय के माध्यम से बैच आकार में वृद्धि से प्रशिक्षण धीमा हो सकता है, खासकर यदि बहुत अधिक संचय चरणों का उपयोग किया जाता है। इसका उद्देश्य जीपीयू उपयोग को अधिकतम करना है लेकिन बहुत अधिक अतिरिक्त ग्रेडिएंट गणना चरणों से अत्यधिक मंदी से बचना है।
विधि 3: ग्रेडिएंट चेकपॉइंटिंग
ग्रेडिएंट चेकपॉइंटिंग एक ऐसी तकनीक है जो कम्प्यूटेशनल समय को उचित रखते हुए प्रशिक्षण के दौरान आवश्यक मेमोरी को कम करती है। बड़े तंत्रिका नेटवर्क बहुत अधिक मेमोरी लेते हैं क्योंकि उन्हें बैकवर्ड पास के दौरान ग्रेडिएंट्स की गणना करने के लिए फॉरवर्ड पास से सभी मध्यवर्ती मानों को संग्रहीत करना पड़ता है। इससे स्मृति संबंधी समस्याएं हो सकती हैं. एक समाधान यह है कि इन मध्यवर्ती मूल्यों को संग्रहीत न किया जाए, लेकिन फिर उन्हें बैकवर्ड पास के दौरान पुनर्गणना करनी होगी, जिसमें बहुत समय लगता है।
ग्रेडिएंट चेकपॉइंटिंग एक संतुलित दृष्टिकोण प्रदान करता है। यह केवल कुछ मध्यवर्ती मानों को सहेजता है, जिन्हें कहा जाता है चौकियों, और आवश्यकतानुसार अन्य की पुनर्गणना करता है। इसलिए, यह हर चीज़ को संग्रहीत करने की तुलना में कम मेमोरी का उपयोग करता है, लेकिन हर चीज़ की पुनर्गणना करने की तुलना में कम गणना भी करता है। रणनीतिक रूप से चेकपॉइंट पर कौन सी सक्रियता का चयन करके, ग्रेडिएंट चेकपॉइंटिंग बड़े तंत्रिका नेटवर्क को प्रबंधनीय मेमोरी उपयोग और गणना समय के साथ प्रशिक्षित करने में सक्षम बनाता है। यह महत्वपूर्ण तकनीक बहुत बड़े मॉडलों को प्रशिक्षित करना संभव बनाती है जो अन्यथा स्मृति सीमाओं में चलेंगे।
हमारी प्रशिक्षण स्क्रिप्ट में, हम आवश्यक पैरामीटर जोड़कर ग्रेडिएंट सक्रियण और चेकपॉइंटिंग चालू करते हैं TrainingArguments वस्तु:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)विधि 4: एलएलएम का निम्न-रैंक अनुकूलन
ESM-2 जैसे बड़े भाषा मॉडल में अरबों पैरामीटर हो सकते हैं जिन्हें प्रशिक्षित करना और चलाना महंगा है। शोधकर्ताओं इन विशाल मॉडलों को और अधिक कुशल बनाने के लिए लो-रैंक एडाप्टेशन (एलओआरए) नामक एक प्रशिक्षण पद्धति विकसित की गई।
LoRA के पीछे मुख्य विचार यह है कि किसी विशिष्ट कार्य के लिए मॉडल को ठीक करते समय, आपको सभी मूल मापदंडों को अपडेट करने की आवश्यकता नहीं होती है। इसके बजाय, LoRA मॉडल में नए छोटे मैट्रिक्स जोड़ता है जो इनपुट और आउटपुट को बदल देता है। फ़ाइन-ट्यूनिंग के दौरान केवल इन छोटे मैट्रिसेस को अपडेट किया जाता है, जो बहुत तेज़ होता है और कम मेमोरी का उपयोग करता है। मूल मॉडल पैरामीटर स्थिर रहते हैं।
LoRA के साथ फ़ाइन-ट्यूनिंग के बाद, आप छोटे अनुकूलित मैट्रिसेस को वापस मूल मॉडल में मर्ज कर सकते हैं। या यदि आप पिछले कार्यों को भूले बिना अन्य कार्यों के लिए मॉडल को शीघ्रता से ठीक करना चाहते हैं तो आप उन्हें अलग रख सकते हैं। कुल मिलाकर, LoRA एलएलएम को सामान्य लागत के एक अंश पर नए कार्यों के लिए कुशलतापूर्वक अनुकूलित करने की अनुमति देता है।
हमारी प्रशिक्षण स्क्रिप्ट में, हम LoRA का उपयोग करके कॉन्फ़िगर करते हैं PEFT हगिंग फेस से लाइब्रेरी:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)सेजमेकर प्रशिक्षण कार्य सबमिट करें
अपनी प्रशिक्षण स्क्रिप्ट परिभाषित करने के बाद, आप सेजमेकर प्रशिक्षण कार्य को कॉन्फ़िगर और सबमिट कर सकते हैं। सबसे पहले, हाइपरपैरामीटर निर्दिष्ट करें:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}इसके बाद, परिभाषित करें कि प्रशिक्षण लॉग से कौन से मेट्रिक्स कैप्चर करने हैं:
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]अंत में, एक हगिंग फेस अनुमानक को परिभाषित करें और इसे ml.g5.2xlarge इंस्टेंस प्रकार पर प्रशिक्षण के लिए सबमिट करें। यह एक लागत प्रभावी उदाहरण प्रकार है जो कई AWS क्षेत्रों में व्यापक रूप से उपलब्ध है:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)निम्न तालिका हमारे द्वारा चर्चा की गई विभिन्न प्रशिक्षण विधियों और हमारे काम के रनटाइम, सटीकता और जीपीयू मेमोरी आवश्यकताओं पर उनके प्रभाव की तुलना करती है।
| विन्यास | बिल करने योग्य समय (मिनट) | मूल्यांकन सटीकता | अधिकतम GPU मेमोरी उपयोग (GB) |
| आधार मॉडल | 28 | 0.91 | 22.6 |
| बेस + जीए | 21 | 0.90 | 17.8 |
| बेस + जीसी | 29 | 0.91 | 10.2 |
| बेस + लोरा | 23 | 0.90 | 18.6 |
सभी विधियों ने उच्च मूल्यांकन सटीकता के साथ मॉडल तैयार किए। LoRA और ग्रेडिएंट सक्रियण का उपयोग करने से रनटाइम (और लागत) में क्रमशः 18% और 25% की कमी आई। ग्रेडिएंट चेकपॉइंटिंग का उपयोग करने से अधिकतम GPU मेमोरी उपयोग में 55% की कमी आई। आपकी बाधाओं (लागत, समय, हार्डवेयर) के आधार पर, इनमें से एक दृष्टिकोण दूसरे की तुलना में अधिक सार्थक हो सकता है।
इनमें से प्रत्येक विधियाँ अपने आप में अच्छा प्रदर्शन करती हैं, लेकिन जब हम उन्हें संयोजन में उपयोग करते हैं तो क्या होता है? निम्न तालिका परिणामों का सारांश प्रस्तुत करती है।
| विन्यास | बिल करने योग्य समय (मिनट) | मूल्यांकन सटीकता | अधिकतम GPU मेमोरी उपयोग (GB) |
| सभी विधियाँ | 12 | 0.80 | 3.3 |
इस मामले में, हम सटीकता में 12% की कमी देखते हैं। हालाँकि, हमने रनटाइम को 57% और GPU मेमोरी के उपयोग को 85% तक कम कर दिया है! यह एक बड़ी कमी है जो हमें लागत प्रभावी उदाहरण प्रकारों की एक विस्तृत श्रृंखला पर प्रशिक्षित करने की अनुमति देती है।
क्लीन अप
यदि आप अपने स्वयं के AWS खाते में अनुसरण कर रहे हैं, तो आगे के शुल्कों से बचने के लिए आपके द्वारा बनाए गए किसी भी वास्तविक समय अनुमान समापन बिंदु और डेटा को हटा दें।
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()निष्कर्ष
इस पोस्ट में, हमने दिखाया कि वैज्ञानिक रूप से प्रासंगिक कार्य के लिए ESM-2 जैसे प्रोटीन भाषा मॉडल को कुशलतापूर्वक कैसे ठीक किया जाए। पीएलएमएस को प्रशिक्षित करने के लिए ट्रांसफॉर्मर और पीईएफटी लाइब्रेरी का उपयोग करने के बारे में अधिक जानकारी के लिए पोस्ट देखें प्रोटीन के साथ गहन शिक्षा और ईएसएमबाइंड (ईएसएमबी): प्रोटीन बाइंडिंग साइट भविष्यवाणी के लिए ईएसएम-2 का निम्न रैंक अनुकूलन हगिंग फेस ब्लॉग पर। आप प्रोटीन गुणों की भविष्यवाणी करने के लिए मशीन लर्निंग का उपयोग करने के और भी उदाहरण पा सकते हैं एडब्ल्यूएस पर विस्मयकारी प्रोटीन विश्लेषण गिटहब भंडार।
लेखक के बारे में
 ब्रायन लॉयल Amazon Web Services में ग्लोबल हेल्थकेयर और लाइफ साइंसेज टीम में एक वरिष्ठ AI/ML समाधान वास्तुकार हैं। उनके पास बायोटेक्नोलॉजी और मशीन लर्निंग में 17 से अधिक वर्षों का अनुभव है, और ग्राहकों को जीनोमिक और प्रोटिओमिक चुनौतियों को हल करने में मदद करने का जुनून है। अपने खाली समय में वह अपने दोस्तों और परिवार के साथ खाना बनाना और खाना पसंद करते हैं।
ब्रायन लॉयल Amazon Web Services में ग्लोबल हेल्थकेयर और लाइफ साइंसेज टीम में एक वरिष्ठ AI/ML समाधान वास्तुकार हैं। उनके पास बायोटेक्नोलॉजी और मशीन लर्निंग में 17 से अधिक वर्षों का अनुभव है, और ग्राहकों को जीनोमिक और प्रोटिओमिक चुनौतियों को हल करने में मदद करने का जुनून है। अपने खाली समय में वह अपने दोस्तों और परिवार के साथ खाना बनाना और खाना पसंद करते हैं।
- एसईओ संचालित सामग्री और पीआर वितरण। आज ही प्रवर्धित हो जाओ।
- प्लेटोडेटा.नेटवर्क वर्टिकल जेनरेटिव एआई। स्वयं को शक्तिवान बनाएं। यहां पहुंचें।
- प्लेटोआईस्ट्रीम। Web3 इंटेलिजेंस। ज्ञान प्रवर्धित। यहां पहुंचें।
- प्लेटोईएसजी. कार्बन, क्लीनटेक, ऊर्जा, पर्यावरण, सौर, कचरा प्रबंधन। यहां पहुंचें।
- प्लेटोहेल्थ। बायोटेक और क्लिनिकल परीक्षण इंटेलिजेंस। यहां पहुंचें।
- स्रोत: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



