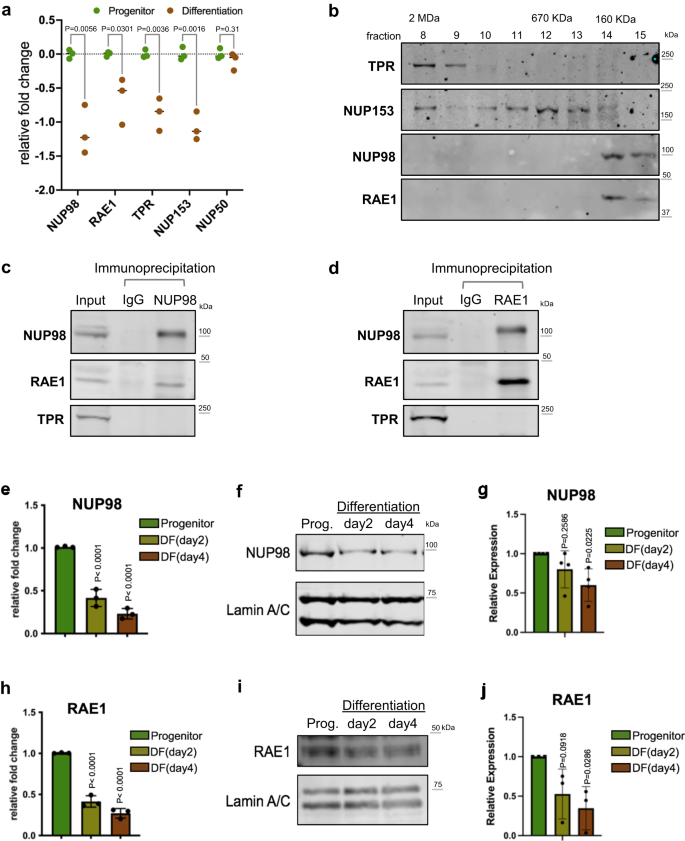
NUP98 et RAE1 sont enrichis en progéniteurs et constituent un complexe distinct
Entre les kératinocytes à l'état progéniteur et à l'état de différenciation, nous avons identifié que 4 NUP du panier nucléaire sur 5 sont significativement régulés à la baisse dans la différenciation (Fig. 1a), tirant parti des données RNA-seq que nous avons récemment générées20. Pour déterminer comment l'enrichissement de ces NUP du panier nucléaire peut influencer le maintien des progéniteurs, nous avons demandé si ces NUP existent dans d'autres complexes à l'intérieur du noyau. Nous avons extrait les fractions solubles des noyaux des kératinocytes à l'état progéniteur et effectué une chromatographie d'exclusion de taille. Alors que TPR et NUP153 éluaient dans une gamme de fractions antérieures, correspondant à des tailles de complexes protéiques plus grandes jusqu'à 2MDa, NUP98 et RAE1 n'éluaient que dans les fractions ultérieures autour du marqueur 160 KDa (Fig. 1b). La co-élution de NUP98 et RAE1, dans les mêmes fractions correspondant à de petits complexes protéiques, suggère que ces deux protéines peuvent s'associer indépendamment l'une de l'autre NUPs. Pour tester cela, nous avons effectué une co-immunoprécipitation à l'aide d'une extraction nucléaire à partir de kératinocytes à l'état progéniteur. L'anticorps NUP98 a co-immunoprécipité à la fois NUP98 et RAE1 ; l'anticorps RAE1 a également co-immunoprécipité à la fois RAE1 et NUP98. Les deux co-immunoprécipitations n'ont pas enrichi d'autres nucléoporines telles que la TPR (Fig. 1c, d). Puisque l'ARN pourrait médier les interactions de NUP98 avec d'autres protéines23, nous avons étudié cela en comparant les co-immunoprécipitations NUP98-RAE1 avec ou sans traitement à la RNase. Fait intéressant, l'association entre NUP98 et RAE1 a été peu affectée par la RNase (Fig. 1a), suggérant que l'interaction NUP98-RAE1 ne nécessite pas de composant ARN dans ce contexte. De plus, nous avons sondé les protéines immunoprécipitées par l'anticorps NUP98 ou RAE1 en utilisant mAb414, qui reconnaît plusieurs NUP contenant le domaine FG. Bien que mAb414 ait détecté plusieurs bandes dans le lysat d'entrée, ce motif de bandes n'a pas été observé dans l'immunoprécipitation par l'anticorps NUP98 ou RAE1 (Fig. 1b). Ces données suggèrent que NUP98 et RAE1 peuvent jouer un rôle dans l'état progéniteur indépendamment des autres NUP.
a Expression relative de l'ARNm des NUP du panier nucléaire, comparant l'état progéniteur aux kératinocytes humains primaires différenciés (jour 4), sur la base des données ARN-seq (test t multiple non apparié, N = 3 réplicats biologiques). b Western blots montrant la distribution des NUP du panier nucléaire dans les fractions de la chromatographie d'exclusion de taille (SEC), en utilisant l'extraction soluble des noyaux de kératinocytes à l'état progéniteur. Les fractions correspondant aux normes protéiques pour SEC sont étiquetées en haut. c, d Western blots montrant la co-immunoprécipitation entre NUP98 et RAE1 dans l'extraction soluble des noyaux (kératinocytes à l'état progéniteur). NUP98 et RAE1 se co-immunoprécipitent, mais pas d'autres sous-unités de pores nucléaires telles que TPR. e RT-qPCR comparant l'expression relative de NUP98 au niveau de l'ARNm dans l'état progéniteur, l'état de différenciation précoce (jour2) et moyen (jour4) des kératinocytes (ANOVA à une voie avec test post-hoc, N = 3 réplicats biologiques, les données sont représentées sous forme de moyenne ± écart type). f, g Western blots et quantifications comparant l'expression de la protéine NUP98 dans la différenciation des kératinocytes, avec Lamin A/C utilisé comme contrôle de charge (ANOVA unidirectionnelle avec test post-hoc, N = 4, les données sont représentées sous forme de moyenne ± écart type). h RT-qPCR comparant l'expression relative de l'ARNm de RAE1 dans la différenciation des kératinocytes (ANOVA unidirectionnelle avec test post-hoc, les données sont représentées sous forme de moyenne ± écart type). i, j Western blots et quantifications comparant les niveaux d'expression de la protéine RAE1 dans la différenciation des kératinocytes, avec Lamin A/C utilisé comme contrôle de charge (ANOVA unidirectionnelle avec test post-hoc, N = 3, les données de quantification sont représentées sous forme de moyenne ± écart type).
Pour mieux comprendre l'expression temporelle de NUP98 et RAE1, passant de l'état progéniteur à la différenciation terminale, nous avons effectué à la fois la qRT-PCR et le Western blot pour quantifier leurs niveaux d'ARNm et de protéines au cours du temps de la différenciation des kératinocytes induite par le calcium. Une réduction significative de NUP98 et RAE1 a été détectée au jour 2 (différenciation précoce), et cette réduction s'est étendue au jour 4 (différenciation moyenne) de l'évolution temporelle de la différenciation (Fig. 1e-j). Ces données indiquent que la régulation à la baisse de NUP98 et RAE1 est un événement précoce dans la différenciation des kératinocytes, qui peut jouer un rôle dans la régulation du passage de l'état progéniteur à la différenciation.
L'inactivation de NUP98 ou RAE1 altère la capacité de régénération des progéniteurs
Pour déterminer si la régulation à la baisse de NUP98 ou de RAE1 favorise le passage de l'état progéniteur à la différenciation, nous avons exploité l'inactivation médiée par le shRNA dans les kératinocytes à l'état progéniteur, en ajustant leur expression à un niveau comparable à l'état de différenciation. Trois shRNA indépendants pour NUP98 ou RAE1 ont été validés au niveau de l'ARNm et des protéines (Fig. 2a, b, Fig. Supplémentaire 2a à d). La réduction de NUP98 ou de RAE1 par ces 6 shARN individuellement était suffisante pour diminuer la clonogénicité des kératinocytes (Fig. 2c–f). Pour évaluer les rôles de NUP98 ou RAE1 dans l'influence de la capacité de régénération des progéniteurs, nous avons effectué une compétition progénitrice dans la régénération épidermique de la peau. Un mélange 50:50 de kératinocytes à l'état progéniteur exprimant GFP ou DsRed a été ensemencé sur du derme humain dévitalisé, élevé dans une interface liquide-air. Ce processus de régénération organotypique se termine en une semaine, formant un épiderme de peau humaine fidèle à l'architecture18,19. Les kératinocytes exprimant la GFP co-expriment le shRNA non ciblant le contrôle ; les kératinocytes exprimant DsRed ont co-exprimé l'un des trois shARN : shARN contrôle non ciblant, shARN ciblant NUP98 ou shARN ciblant RAE1. Pour l'épiderme régénéré à l'aide de kératinocytes exprimant tous deux les shARN de contrôle non ciblant, les kératinocytes fluorescents rouges et verts ont montré une représentation comparable dans le tissu, indiquant que l'expression de GFP ou de DsRed n'a pas influencé différemment la capacité de régénération des progéniteurs. En revanche, les kératinocytes rouges co-exprimant le shRNA NUP98 ou RAE1 ont été supplantés par les kératinocytes verts co-exprimant le shRNA contrôle, avec une représentation réduite dans le compartiment progéniteur basal (Fig. 2g, h). Ces résultats suggèrent que le niveau d'expression élevé de NUP98 et de RAE1 est essentiel pour le maintien des progéniteurs.
a, b RT-qPCR montrant l'efficacité de knockdown de 3 shRNA indépendants ciblant NUP98 ou RAE1 (ANOVA unidirectionnelle avec test post-hoc, N = 3, les données sont représentées sous forme de moyenne ± écart type). c-f Images représentatives et quantification du test de clonogénicité comparant les kératinocytes avec NUP98 ou RAE1 knockdown par rapport au contrôle (ANOVA unidirectionnelle avec test post-hoc, N = 3, les données de quantification sont représentées sous forme de moyenne ± écart type.) g Images représentatives du test de compétition dans la régénération des tissus épidermiques. Un nombre égal de kératinocytes exprimant DsRed ou GFP ont été mélangés ensemencés sur du derme humain. Les kératinocytes exprimant la GFP co-expriment le shRNA de contrôle et les kératinocytes exprimant le DsRed co-expriment le shRNA de contrôle ou le shRNA ciblant NUP98 ou RAE1 (barre d'échelle = 125 μm). h Quantification des kératinocytes exprimant DsRed ou GFP dans la couche basale de l'épiderme régénéré, les données sont représentées sous forme de moyenne ± écart type. i Diagramme de Venn montrant le chevauchement des gènes NUP98 et RAE1 knockdown différentiellement exprimés (test exact de Fisher, p = 1 × 10-321). j Heatmap montrant l'expression relative des gènes partagés avec NUP98 ou RAE1 knockdown. k Les trois principaux termes d'ontologie génique (GO) pour les gènes régulés positivement ou négativement partagés par NUP98 et RAE1 knockdown, identifiés par des analyses RNA-seq.
Pour identifier les processus cellulaires clés influencés par l'inactivation de NUP98 ou RAE1, nous avons effectué un profilage du transcriptome à l'aide d'ARN-seq. Au total, nous avons identifié 1493 gènes modifiés de manière significative avec le knockdown de NUP98 et 1401 gènes modifiés significatifs avec le knockdown de RAE1 (p <0.05, bilatéral, test de Wald, changement de pli moyen pour trois shRNA indépendants> 2 et changement de pli de shRNA individuel> 1.5, Données supplémentaires 1, Données supplémentaires 2). Ces deux ensembles partagent 597 gènes (p = 1 × 10-321, test exact de Fishers), qui sont modifiés dans le même sens (régulé à la hausse ou à la baisse) avec NUP98 ou RAE1 knockdown (Fig. 2i,j). Les principaux termes d'ontologie des gènes (GO) des gènes régulés positivement partagés incluent le développement épidermique et la différenciation des kératinocytes ; Les principaux termes GO des gènes partagés régulés à la baisse sont liés à la division cellulaire (Fig. 2k). Nous avons en outre identifié que 77% de ces gènes partagés sont également significativement modifiés dans la différenciation induite par le calcium (Fig. S2e). De plus, nous avons recherché si l'inactivation de NUP98 ou de RAE1 était suffisante pour déclencher l'apoptose, en tirant parti de deux colorants différents sensibles au potentiel des mitochondries. Alors que la coloration était abolie dans les kératinocytes témoins positifs traités avec H2O2, les kératinocytes avec NUP98 ou RAE1 knockdown ont conservé la coloration similaire au knockdown témoin (Fig. 2f,g), suggérant que la stratégie de knockdown n'a pas déclenché l'apoptose. Ainsi, ces résultats suggèrent que l'enrichissement de NUP98 et RAE1 à l'état progéniteur est essentiel pour maintenir la prolifération et réprimer la différenciation.
NUP98 se lie à proximité des TSS des régulateurs clés dans la maintenance des progéniteurs
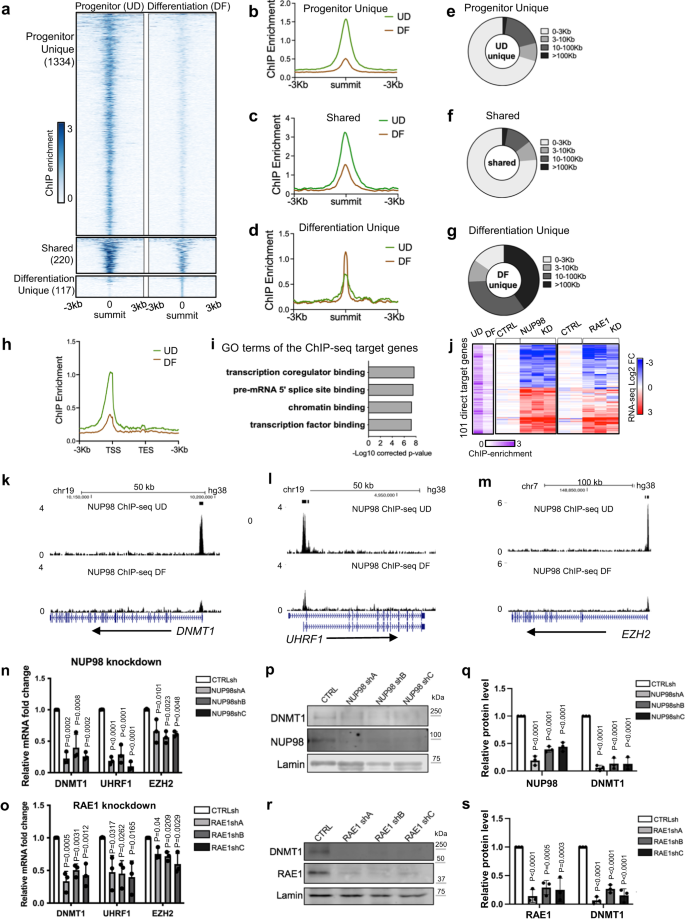
Il a été rapporté que NUP98 se lie à différentes régions génomiques dans le contexte de différents types de cellules14,24. Pour étudier comment la liaison génomique NUP98 pourrait influencer l'expression des gènes dans le maintien des progéniteurs épidermiques, nous avons effectué NUP98 ChIP-seq dans les kératinocytes. Dans les kératinocytes à l'état progéniteur, nous avons identifié un total de 1554 pics NUP98 ChIP. Dans l'état de différenciation, cependant, la liaison de NUP98 est réduite dans toutes ces régions. La majorité (86%, 1334 pics) de ces pics NUP98 ChIP-seq sont uniques pour l'état progéniteur, mais pas pour l'état de différenciation. Seuls 14 % de ces pics (220 pics) ont également été appelés à l'état de différenciation, mais l'enrichissement de la puce au niveau de ces régions est également réduit en différenciation. Un petit nombre (117) de pics de puce NUP98 ont été identifiés comme uniques à l'état de différenciation, dont la plupart (74.3 %) sont situés à au moins 10 kb des TSS. En revanche, la plupart des pics (71%) identifiés à l'état progéniteur sont situés à moins de 3 kb des sites de début de transcription (Fig. 3a à g, Données supplémentaires 3). Ainsi, le passage de l'état progéniteur à la différenciation a impliqué une réduction globale de la liaison génomique de NUP98, en particulier près des sites de début de transcription (Fig. 3h).
a-d Cartes thermiques centrées sur le sommet et diagrammes moyens comparant l'enrichissement de la puce NUP98 entre l'état progéniteur (UD) et l'état de différenciation (DF). e-g Graphiques circulaires montrant la distribution des pics NUP98 ChIP-seq, en fonction de leurs distances par rapport aux sites de début de transcription (TSS) les plus proches. h Diagramme moyen comparant l'enrichissement de NUP98 ChIP-seq près des sites de début et de fin de transcription des gènes cibles. i Top Termes d'ontologie génétique (GO) des gènes les plus proches associés aux pics NUP98 ChIP-seq. j Heatmap montrant les 101 gènes, avec l'enrichissement NUP98 ChIP-seq et sont considérablement modifiés avec le knockdown NUP98. L'enrichissement relatif de la puce NUP98 en UD et DF associés à ces gènes, et leur expression relative avec NUP98 ou RAE1 knockdown, sont inclus pour chacun de ces gènes côte à côte. k-m Exemples de suivi de navigateur d'enrichissement NUP98 ChIP-seq, comparant UD et DF. n, o Validation qRT-PCR de gènes cibles représentatifs avec knockdown NUP98 ou RAE1 (ANOVA unidirectionnelle avec test post-hoc, N = 3 réplicats biologiques, les données sont représentées sous forme de moyenne ± écart type). p-s Western blot et quantification comparant les niveaux de protéine DNMT1 avec NUP98 ou RAE1 knockdown, avec Lamin utilisé comme contrôle de chargement (ANOVA unidirectionnelle avec test post-hoc, N = 3, les données de quantification sont représentées sous forme de moyenne ± écart type).
Nous avons ensuite annoté ces gènes, qui sont associés à des pics de liaison NUP98 ChIP-seq à l'état progéniteur. Les principaux termes GO sont liés à la liaison au co-régulateur de transcription, à la liaison à la chromatine et à la liaison au facteur de transcription (Fig. 3i), suggérant que ces gènes pourraient être des régulateurs en amont de processus biologiques clés. L'intersection des cibles ChIP-seq avec les données NUP98 RNA-seq a identifié un total de 101 gènes cibles directs. La majorité de ces 101 gènes cibles directs présentent également une régulation à la hausse ou à la baisse similaire avec l'inactivation de RAE1 (Fig. 3j). Fait intéressant, les gènes cibles directs sous-régulés comprennent des régulateurs clés régissant la maintenance des progéniteurs épidermiques, y compris l'ADN méthyltransférase DNMT1 et son recruteur URHF16, la protéine du groupe polycomb EZH23 (Figue. 3k-o), en plus des régulateurs de réplication de l'ADN CDT1 et RRM2. Ces gènes cibles directs incluent également des gènes régulés positivement tels que DUSP10 et JARID2, qui sont moins bien caractérisés dans le contexte de la différenciation des kératinocytes (Données supplémentaires 4).
Pour déterminer si RAE1 se lie à la chromatine avec NUP98, nous avons généré une construction RAE1 étiquetée HA et l'avons exprimée dans des kératinocytes, car les anticorps disponibles dans le commerce que nous avons criblés n'ont pas donné de données ChIP-seq de haute qualité. HA-RAE1 co-immunoprécipite NUP98 dans les kératinocytes à l'état progéniteur (Fig. 3a), confirmant que la balise HA n'interfère pas avec l'association de RAE1 avec NUP98. À l'aide de ChIP-seq à double réticulation, nous avons identifié que RAE1 est enrichi dans 83 % des régions de pointe NUP98 ChIP-seq. Les pics NUP98-RAE1 chevauchant ChIP-seq, mais pas les pics uniques NUP98, sont principalement situés près des TSS (Fig. 3b-f). Parmi les 101 gènes cibles directs de NUP98, 96 de ces gènes sont également associés à l'enrichissement RAE1 ChIP-seq, y compris DNMT1 et EZH2 (Fig. 3g–je). Ces résultats suggèrent que NUP98 et RAE1 se lient directement près des TSS pour réguler l'expression des gènes.
DNMT1 a déjà été caractérisé comme un régulateur clé pour la maintenance des progéniteurs épidermiques humains6. Nous avons confirmé la régulation à la baisse drastique de DNMT1 dans la différenciation des kératinocytes en utilisant le Western Blot (Fig. 4a, b). Tirer parti des données publiées sur le profilage du transcriptome de l'inactivation de DNMT1 dans les kératinocytes à l'état progéniteur6, nous avons comparé les gènes exprimés de manière différentielle lors de l'inactivation de DNMT1 avec les gènes exprimés de manière différentielle lors de l'inactivation de NUP98 ou de RAE1. L'intersection de ces ensembles de données a identifié un total de 236 gènes partagés (Fig. 4c, d). Les principaux termes GO des gènes partagés régulés positivement sont associés au développement épidermique et à la différenciation des kératinocytes ; les gènes régulés à la baisse sont liés à la division cellulaire et à la réplication de l'ADN (Fig. 4e, f). Nous avons en outre validé à l'aide d'un transfert Western que les niveaux de protéine DNMT1 sont considérablement réduits avec l'inactivation de NUP98 ou RAE1, dans les kératinocytes cultivés dans la condition progénitrice (Fig. 3p–s). Ainsi, la régulation à la baisse de DNMT1 explique au moins partiellement l'induction de la différenciation et l'inhibition de la prolifération observées avec l'inactivation de NUP98 ou de RAE1. Prises ensemble, ces données suggèrent que la liaison à la chromatine de NUP98 et RAE1 est impliquée dans la maintenance des progéniteurs, en contrôlant directement l'expression de régulateurs clés de prolifération/différenciation tels que DNMT1.
NUP98 co-localise avec HDAC1 sur la chromatine et coopère avec HDAC dans la régulation des gènes
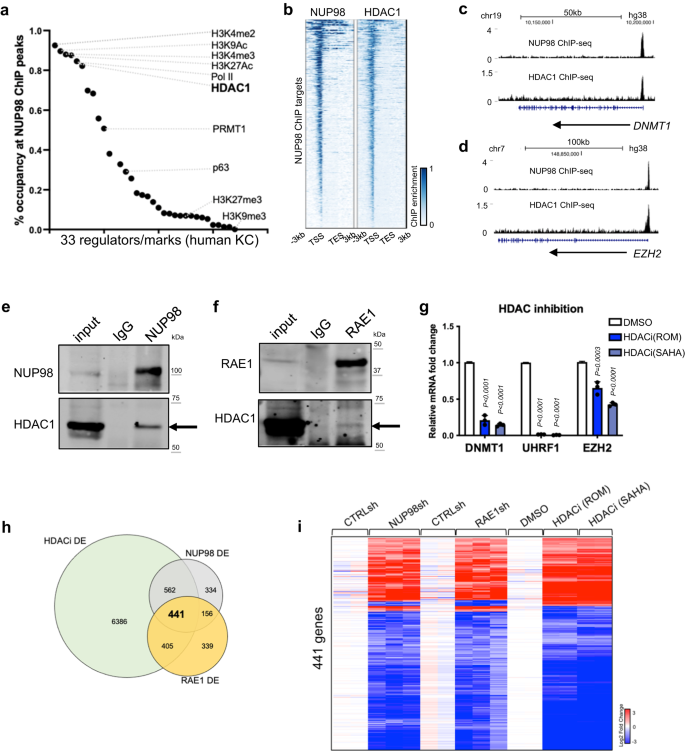
Compte tenu de la liaison de la chromatine de NUP98 aux gènes codant pour les régulateurs clés du processus de prolifération/différenciation, nous avons étudié les mécanismes potentiels facilitant la liaison de la chromatine de NUP98 à ces régions génomiques spécifiques. La recherche de motifs n'a pas permis de découvrir des facteurs de transcription spécifiques pouvant expliquer la majorité des sites de liaison génomique. Nous avons ensuite comparé les pics NUP98 ChIP-seq avec les fichiers de pics ChIP-seq d'autres régulateurs transcriptionnels et les marques d'histones générées à l'aide du même type cellulaire de kératinocytes humains primaires (Fig. 4a, Données supplémentaires 5). Conformément à la liaison de NUP98 près des sites de début de transcription, les régions de pointe NUP98 ChIP-seq sont fortement (> 80 %) co-occupées par les marques d'histone (H3K4me2/3, H3K27Ac, H3K9Ac) et Pol II. Fait intéressant, HDAC1 co-occupe également 82% de tous les pics NUP98 ChIP-seq, comparables à Pol II (85%) et beaucoup plus élevés que le facteur de transcription spécifique à la lignée p63 (29%). Semblable à NUP98, HDAC1 se lie également près des TSS des gènes cibles de NUP98, tels que DNMT1 et EZH2 (Fig. 4b-d). Ainsi, HDAC1 s'est imposé comme un candidat pouvant coopérer avec NUP98 dans la liaison à la chromatine et la régulation des gènes.
a Occupation d'autres marques épigénétiques ou régulateurs dans les régions de pointe NUP98 ChIP-seq. b Heatmap comparant NUP98 ChIP-seq et HDAC1 ChIP-seq sur les pics NUP98. c, d Pistes de navigateur montrant la co-occupation de NUP98 et HDAC1 sur des gènes cibles représentatifs. e, f Co-immunoprécipitation par réticulation utilisant l'anticorps NUP98 ou RAE1 pour détecter HDAC1. g qRT-PCR montrant l'expression relative des gènes cibles RAE1-NUP98 avec inhibition de l'HDAC (ANOVA unidirectionnelle avec test post-hoc, N = 3, les données sont représentées sous forme de moyenne ± écart-type). hDiagramme de Venn montrant le chevauchement des gènes NUP98, RAE1 et HDACi exprimés de manière différentielle. i Carte thermique montrant l'expression des gènes exprimés de manière différentielle partagés entre l'inactivation de NUP98, l'inactivation de RAE1 et l'inhibition de HDAC.
Ce chevauchement étendu des pics NUP98 et HDAC1 ChIP-seq a suggéré que ces deux protéines pourraient s'associer physiquement. Nous avons confirmé cela en utilisant l'immunoprécipitation par réticulation, que l'anticorps NUP98 ou RAE1 co-immunoprécipitait HDAC1 dans le lysat de kératinocytes (Fig. 4e, f). Pour déterminer si l'HDAC influence l'expression génique similaire à NUP98 et RAE1, nous avons utilisé les inhibiteurs d'HDAC Romidepsin (ROM) et SAHA. Lorsqu'ils sont ajoutés à des kératinocytes cultivés à l'état progéniteur, ces inhibiteurs régulent constamment à la baisse les gènes cibles directs représentatifs de NUP98, tels que DNMT1, UHRF1 et EZH2 (Fig. 4g). En utilisant RNA-seq, nous avons en outre identifié que 74% des gènes cibles NUP98-RAE1 sont également significativement modifiés par l'inhibition de HDAC (Fig. 4h, je, Données supplémentaires 6). Étant donné que p300 a également été identifié comme une protéine interagissant avec les protéines de fusion NUP98 dans les tumeurs malignes hématopoïétiques25, nous avons constaté que l'inhibition de p300 ne modifiait pas radicalement l'expression du gène cible de NUP98 dans les kératinocytes (Fig. 5a), soutenant que cette HDAC est spécifiquement impliquée dans la modulation des gènes cibles contrôlés par NUP98 et RAE1 dans le maintien des progéniteurs épidermiques.
En nous appuyant sur les modifications de l'expression génique des gènes cibles de NUP98 et RAE1 induites par l'inhibition de HDAC, nous avons étudié plus en détail les rôles de HDAC1. Nous avons conçu et validé un total de 3 shRNA indépendants ciblant HDAC1. Fait intéressant, ces trois shARN ont systématiquement régulé à la baisse les gènes cibles de NUP98 et RAE1, tels que DNMT1 et UHRF1 (Fig. 5b). Ainsi, HDAC1 est impliqué dans la régulation de l'expression des gènes cibles de NUP98 et RAE1 à l'état progéniteur. Dans le processus de différenciation des kératinocytes, le niveau de protéine de HDAC1 est légèrement réduit, avec une expression relative moyenne de 54 % au jour de différenciation 4 par rapport à l'état progéniteur (Fig. 5c). De manière constante, l'enrichissement de la puce HDAC1 n'a été que modérément réduit dans l'état de différenciation (Fig. 5d). Nous avons en outre comparé l'enrichissement de HDAC1 ChIP-seq dans les sites de liaison NUP98 entre l'état progéniteur et l'état de différenciation (jour 4, Fig. 5e–h). La majorité (92.1%) de ces sites n'ont pas montré de réduction drastique de la liaison HDAC1 dans la différenciation. Seulement dans une très petite fraction (7.7 %) de ces sites, l'enrichissement en HDAC1 a montré une réduction significative (changement de facteur > 2, p < 0.05). Ainsi, l'enrichissement de HDAC1 ChIP n'a été que modestement réduit en différenciation dans les sites de liaison NUP98, contrairement à la réduction drastique de NUP98 dans ces sites en différenciation, ce qui suggère que NUP98 n'est pas nécessaire pour maintenir la liaison à la chromatine de HDAC1 dans leurs sites de liaison partagés.
La liaison de la chromatine NUP98 dépend de l'activité HDAC
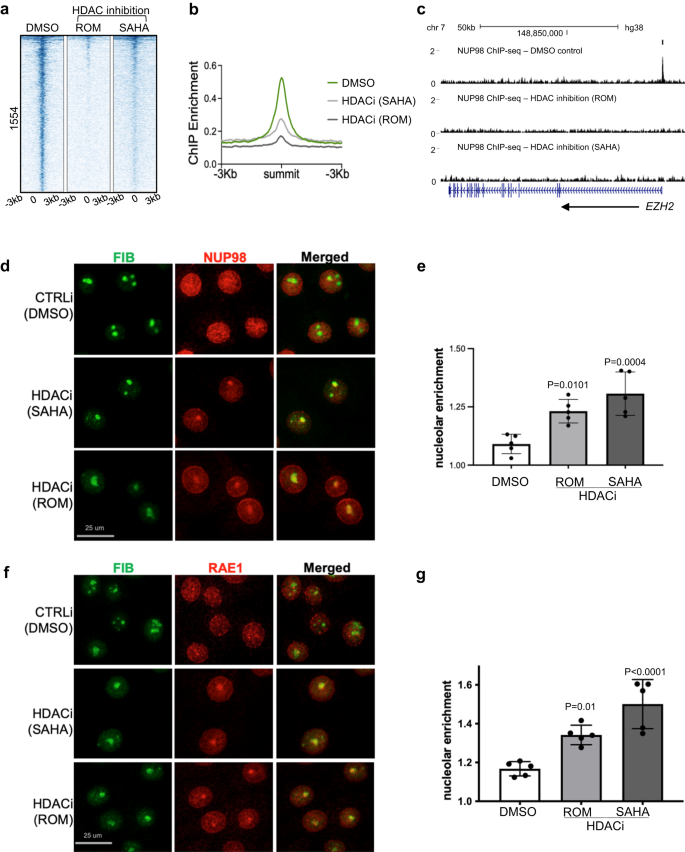
Compte tenu du chevauchement entre NUP98 et HDAC1 dans les sites de liaison génomique et dans la régulation des gènes, nous avons demandé si l'activité HDAC fonctionnait en amont pour influencer la liaison génomique de NUP98. Nous avons effectué NUP98 ChIP-seq dans des kératinocytes avec ou sans inhibition de HDAC. Remarquablement, les signaux NUP98 ChIP-seq ont été diminués avec l'inhibition de HDAC en utilisant SAHA ou Romidepsin (Fig. 5a – c), suggérant que la liaison génomique de NUP98 à ses gènes cibles dépend de l'activité HDAC.
a Carte thermique montrant l'enrichissement de la puce NUP98 avec l'inhibition de l'HDAC (à l'aide de ROM ou de SAHA) par rapport au contrôle DMSO. b Profil moyen montrant l'enrichissement de la puce NUP98 avec inhibition de HDAC (en utilisant ROM ou SAHA) par rapport au contrôle DMSO. c Piste du navigateur montrant un gène cible représentatif EZH2, comparant l'enrichissement de la puce NUP98 avec l'inhibition de l'HDAC versus le contrôle DMSO, dans des kératinocytes cultivés à l'état progéniteur. d Images représentatives montrant des kératinocytes co-immunocolorés avec des anticorps ciblant NUP98 et le marqueur nucléole Fibrillarine (FIB), comparant l'inhibition HDAC au contrôle DMSO (barre d'échelle = 25 μm). e Quantification de l'enrichissement relatif en NUP98 dans le nucléole dans l'inhibition de l'HDAC par rapport au contrôle DMSO (ANOVA unidirectionnelle avec test post-hoc, N = 5, les données sont représentées sous forme de moyenne ± écart type). f, g Co-immunomarquage de RAE1 et FIB dans les kératinocytes traités avec les inhibiteurs d'HDAC versus le contrôle DMSO (barre d'échelle = 25 μm), et quantification (ANOVA à une voie avec test post-hoc, N = 5, les données de quantification sont représentées en moyenne ± standard déviation).
Pour déterminer si la localisation subcellulaire de NUP98 était également affectée par l'inhibition de l'HDAC, nous avons effectué une coloration par immunofluorescence NUP98 dans les kératinocytes avec inhibition de l'HDAC par rapport au contrôle DMSO. Alors que NUP98 présentait une coloration diffuse à l'intérieur du noyau dans la condition de contrôle, en plus de son enrichissement de la périphérie nucléaire, l'inhibition de HDAC par SAHA ou Romidespin entraînait systématiquement 1 à 2 foyers agrégés de NUP98 dans chaque noyau. La co-coloration avec le marqueur nucléole fibrilline a confirmé que le NUP98 était localisé au nucléole (Fig. 5d, e). Semblable à NUP98, RAE1 s'est également agrégé au nucléole lors de l'inhibition de HDAC (Fig. 5f,g).
Un article récent décrit le ciblage FUS vers le nucléole sous stress transcriptionnel26. Nous avons demandé si FUS est impliqué dans l'enrichissement du nucléole de NUP98 et RAE1. Une coloration par co-immunofluorescence a été réalisée pour NUP98 et FUS avec inhibition de HDAC, par rapport au témoin. Alors que NUP98 était constamment enrichi dans le nucléole avec l'inhibition de HDAC, aucun enrichissement en FUS n'a été détecté (Fig. 6). Ces données suggèrent que le ciblage nucléolaire de NUP98 et RAE1 est indépendant des mécanismes impliqués dans le ciblage du FUS vers le nucléole.
La localisation nucléolaire de NUP98 et RAE1 est équilibrée par les activités HDAC et HAT
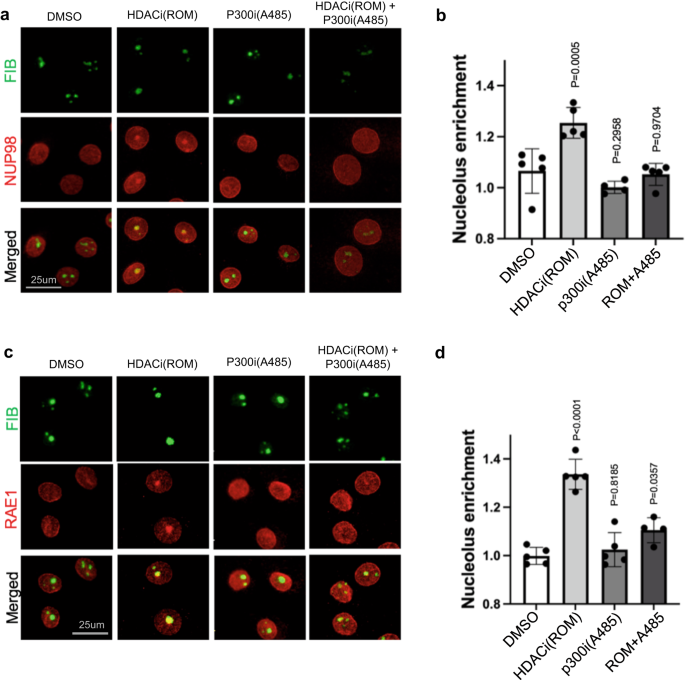
L'enrichissement nucléolaire de NUP98 et RAE1, induit par l'inhibition de HDAC, suggère que l'acétylation des protéines pourrait être impliquée dans ce processus. Pour tester cela, nous avons conçu une expérience pour inhiber l'acétylation des protéines en combinaison avec l'inhibition de l'HDAC, en tirant parti des inhibiteurs p300/CBP HAT A485 et C64627,28. Comme le montre la Fig. 6 et Fig supplémentaire. 7, l'inhibition de HAT seule n'a pas modifié de manière significative la localisation sous-nucléaire de NUP98 ou RAE1 ; cependant, lorsque les kératinocytes ont été prétraités avec des inhibiteurs de la HAT pendant 6 h, l'inhibition de l'HDAC n'a plus été en mesure d'induire la localisation nucléolaire de NUP98 ou de RAE1. Cet antagonisme entre l'inhibition de la HAT et l'inhibition de l'HDAC a été systématiquement observé parmi les deux inhibiteurs indépendants de p300/CBP (A485 ou C646). Ces données soutiennent un rôle de l'acétylation des protéines dans la médiation de la localisation nucléolaire de NUP98 ou RAE1, en conséquence de l'inhibition de HDAC.
a Co-immunomarquage de NUP98 et du marqueur nucléole FIB dans les kératinocytes traités avec l'inhibiteur HDAC, ROM et/ou l'inhibiteur HAT A485 (barre d'échelle = 25 μm). b quantification de l'enrichissement du nucléole NUP98 dans ces conditions de traitement (ANOVA unidirectionnelle avec test post-hoc, N = 5, les données sont représentées sous forme de moyenne ± écart type). c Co-immunomarquage de RAE1 et du marqueur nucléole FIB dans les kératinocytes traités avec l'inhibiteur HDAC, ROM et/ou l'inhibiteur HAT A485 (barre d'échelle = 25 μm). d quantification de l'enrichissement du nucléole RAE1 parmi ces conditions (ANOVA unidirectionnelle avec test post-hoc, N = 5, les données sont représentées sous forme de moyenne ± écart type).
Interdépendance entre NUP98 et RAE1 dans la localisation nucléolaire lors de l'inhibition de HDAC
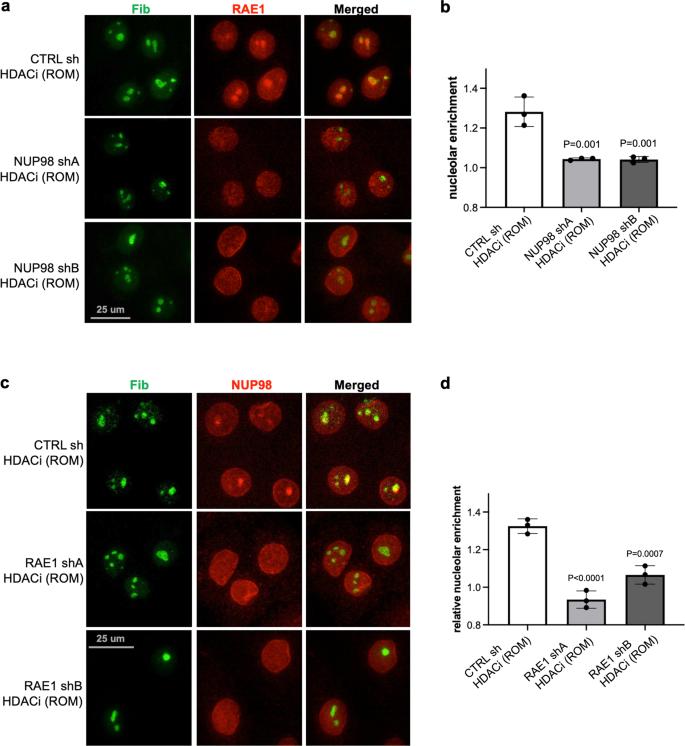
Compte tenu de l'association entre NUP98 et RAE1, nous avons en outre demandé si le ciblage nucléolaire de NUP98 lors de l'inhibition de HDAC dépendait de RAE1. Alors que les kératinocytes exprimant des shARN témoins non ciblant montraient toujours une agrégation de NUP98 avec inhibition de HDAC, cette agrégation nucléolaire était abolie dans les kératinocytes exprimant des shARN ciblant RAE1 (Fig. 7a, b). Ainsi, l'enrichissement nucléolaire de NUP98 nécessite la fonction intacte de RAE1. Nous avons ensuite étudié si le ciblage nucléolaire de RAE1 lors de l'inhibition de HDAC dépendait également de NUP98. Nous avons traité des kératinocytes exprimant soit NUP98 shA, NUP98 shB ou contrôle shRNA non ciblant avec l'inhibiteur de HDAC ROM. Le ciblage nucléolaire de RAE1 a été aboli dans les kératinocytes exprimant le shRNA NUP98, mais pas le contrôle non ciblant. En utilisant deux shARN indépendants ciblant NUP98, RAE1 ne ciblait pas non plus le nucléole (Fig. 7c, d). L'inactivation de RAE1 seule n'a pas induit de localisation nucléolaire de NUP98, et l'inactivation de NUP98 n'a pas induit de localisation nucléolaire de RAE1, sans inhibition de HDAC (Fig. 8). Ces résultats indiquent que NUP98 et RAE1 dépendent l'un de l'autre pour se localiser dans le nucléole lors de l'inhibition de HDAC.
a, b Images représentatives montrant la co-immunocoloration de RAE1 et du marqueur nucléole FIB dans les kératinocytes traités avec NUP98 knockdown versus contrôle knockdown (barre d'échelle = 25 μm), et quantifications de l'enrichissement nucléolaire RAE1 parmi ces conditions (ANOVA unidirectionnelle avec test post-hoc, N = 3, les données de quantification sont représentées sous forme de moyenne ± écart type). c, d Co-immunomarquage de NUP98 et du marqueur nucléole FIB dans les kératinocytes traités avec RAE1 knockdown versus contrôle knockdown (barre d'échelle = 25 μm), et quantifications de l'enrichissement nucléolaire NUP98 parmi ces conditions (ANOVA à une voie avec test post-hoc, N = 3 , les données de quantification sont représentées sous forme de moyenne ± écart type).
Pris ensemble, ces résultats suggèrent un modèle selon lequel NUP98 et RAE1 favorisent le maintien des progéniteurs en se liant directement et en contrôlant l'expression des principaux régulateurs épigénétiques sous deux conditions : expression élevée et activité HDAC. L'expression élevée à l'état progéniteur permet la présence d'un pool intranucléaire soluble de NUP98 et RAE1, permettant la liaison à la chromatine en plus de leur association noyau-pore ; L'activité HDAC est également essentielle pour contrarier le ciblage nucléolaire de NUP98 et RAE1, favorisant le ciblage génomique de NUP98 vers les principaux régulateurs épigénétiques pour l'auto-renouvellement (Fig. 8).
NUP98 et RAE1 soutiennent le maintien des progéniteurs épidermiques en se liant à la chromatine près des sites de début de transcription des gènes cibles clés tels que le répresseur épigénétique DNMT1. Les rôles de NUP98 et RAE1 dans la maintenance des progéniteurs dépendent de deux facteurs : une expression élevée et une activité HDAC. L'expression élevée de NUP98 et RAE1 fournit le pool de liaison à la chromatine, en plus de leur incorporation de pores nucléaires ; L'activité HDAC soutient la liaison de NUP98 sur la chromatine près des TSS des gènes cibles clés, antagonisant la localisation nucléolaire de NUP98 et RAE1.
- Contenu propulsé par le référencement et distribution de relations publiques. Soyez amplifié aujourd'hui.
- PlatoData.Network Ai générative verticale. Autonomisez-vous. Accéder ici.
- PlatoAiStream. Intelligence Web3. Connaissance Amplifiée. Accéder ici.
- PlatonESG. Automobile / VE, Carbone, Technologie propre, Énergie, Environnement, Solaire, La gestion des déchets. Accéder ici.
- Décalages de bloc. Modernisation de la propriété des compensations environnementales. Accéder ici.
- La source: https://www.nature.com/articles/s42003-023-05043-2