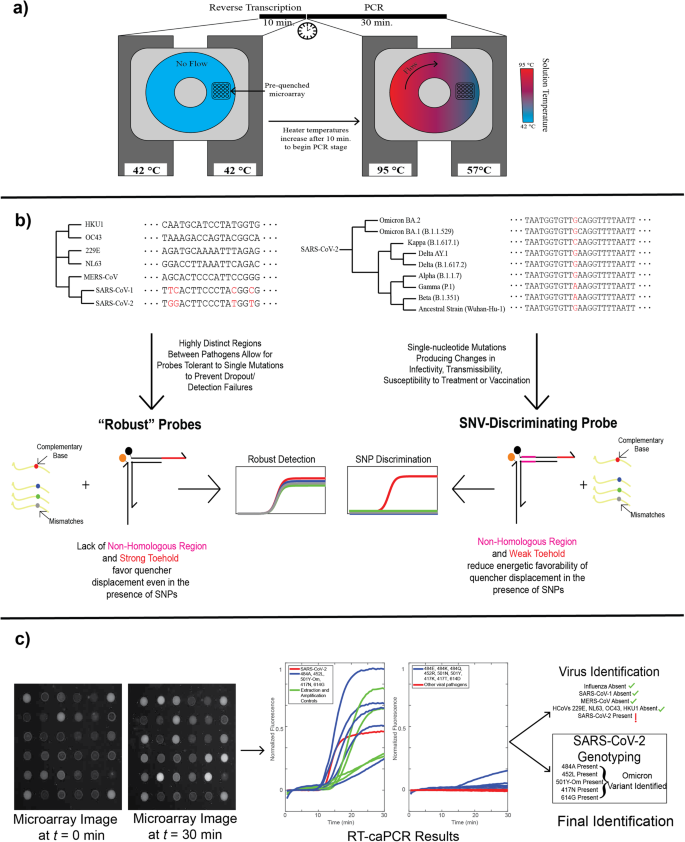
Intégration de la transcription inverse dans caPCR
Nous avons d’abord introduit une étape RT dans le test caPCR standard. La caPCR est réalisée à l'aide d'un ensemble de 4 éléments chauffants qui se fixent sur chaque face d'une cartouche contenant une chambre toroïdale remplie du mélange réactionnel PCR.4. En réglant les radiateurs d'un côté de la chambre à la température d'hybridation PCR et de l'autre à la température de dénaturation, un gradient est établi, produisant ainsi un flux convectif passif qui entraîne l'amplification PCR. Le réseau de sondes pré-trempées est immobilisé sur la surface de la cartouche et répond via TMSD à mesure que des produits PCR spécifiques sont synthétisés. Pour permettre une première étape de transcription inverse, nous avons programmé les réchauffeurs pour maintenir une température constante de 42 °C pour la synthèse de l'ADNc pendant 10 minutes, après quoi les réchauffeurs s'ajustent à 57 °C et 95 °C respectivement pour initier un flux convectif (Fig. 2a). Une approche RT-PCR one-pot est utilisée pour réduire le nombre d’étapes de manipulation manuelle dans le flux de travail. La détection médiée par la sonde Toehold sur la plateforme caPCR fonctionne de manière optimale dans des conditions de PCR asymétriques : un excès d'amorce qui produit le brin qui déplace l'extincteur génère un signal plus fort sur le réseau de sondes fluorescentes qu'un rapport d'amorce avant/arrière de 1 : 1. (Figue. S1). Nous avons veillé à ce que cette même amorce médie également la synthèse de l'ADNc à partir des cibles d'ARN afin d'éviter d'avoir à introduire des amorces distinctes pour les étapes de transcription inverse et de PCR.
a Le flux de travail caPCR peut être étendu pour inclure une étape de transcription inverse (RT-caPCR) grâce à l’ajout d’une enzyme transcriptase inverse et une étape RT dédiée avec tous les radiateurs de l’appareil réglés à la température RT (42 °C). Les températures de chauffage sont ensuite augmentées à différentes températures pour la dénaturation (95 °C) et l'hybridation (57 °C), induisant un flux convectif et une amplification PCR des matrices d'ADNc. b Le schéma de conception énergétique des sondes, basé sur la thermodynamique de liaison à l'ADN, peut être utilisé pour répondre à deux besoins en matière de diagnostic des maladies infectieuses : I) La détection des agents pathogènes viraux doit être robuste aux petits changements dans la séquence nucléotidique pour éviter l'abandon si de nouvelles variantes apparaissent. II) La discrimination des variantes nécessite une résolution sur un seul nucléotide. La variation des énergies de pied, de domaine et de région non homologue (NHR) peut être utilisée pour répondre à ces deux exigences. c Exemple de sortie de données d’une analyse RT-caPCR en beignet. Les images du réseau de sondes sont acquises toutes les 30 secondes pendant 30 minutes de caPCR. Les traces de fluorescence sont extraites et analysées pour identifier quels agents pathogènes respiratoires sont présents et, le cas échéant, quelle variante du SRAS-CoV-2 est présente.
Pour développer des sondes accordables avec une sélectivité de séquence programmable, allant de sensible aux mutations à tolérante aux mutations, nous avons utilisé 2 ensembles de critères énergétiques pour les sondes de pied (Fig. 2b). Les sondes robustes comportent une région d'appui plus forte et un domaine correspondant plus faible pour réduire l'influence globale d'une seule mutation sur l'initiation de la réaction de liaison et le déplacement ultérieur du bras d'extinction. Pour la discrimination SNV, la longueur de la pointe est réduite et une région non homologue (NHR) manquant de complémentarité avec la cible est introduite. Cette combinaison sert à réduire la favorabilité énergétique globale pour la fixation de la pointe et le déplacement du bras d'extincteur, fournissant ainsi un poids supplémentaire aux mésappariements uniques dans la séquence cible.
Nous avons appliqué la RT-caPCR avec des sondes à la fois robustes et discriminantes du SNV à la détection des virus respiratoires. Les images du réseau de sondes du système RT-caPCR sont traitées à l'aide d'un script MATLAB personnalisé pour produire des traces de fluorescence qui sont ensuite interprétées pour identifier à la fois la ou les cibles virales présentes et, le cas échéant, la variante du SRAS-CoV-2. Un exemple est présenté pour la variante SARS-CoV-2 Omicron sur la figure. 2c. Ainsi, notre test RT-caPCR one-pot démontre sa capacité à effectuer simultanément une détection sensible avec des sondes robustes ainsi qu'une résolution mononucléotidique avec des sondes discriminatoires SNV pour identifier avec précision les cibles d'ARN d'intérêt dans une seule réaction.
Affiner l'énergie entre les sondes robustes et discriminantes SNV
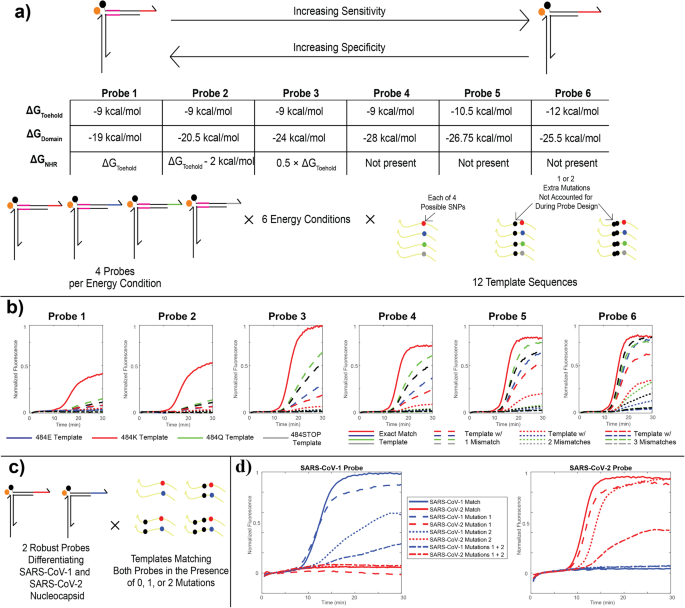
Pour augmenter la diversité des cibles détectées dans notre test RT-caPCR, nous avons optimisé l'énergie de la liaison au pied et du déplacement des brins pour moduler le compromis sensibilité-spécificité. À titre d'étude de cas, nous avons utilisé le génotype « 2E » du SRAS-CoV-484 comme modèle pour notre évaluation énergétique. Les mutations sur ce site sont associées à une activité neutralisante réduite des anticorps de l'hôte12, et 3 des 4 bases nucléotidiques possibles (adénine, guanine et cytosine) se trouvent en première position du codon dans les variantes naturellement évoluées du SRAS-CoV-29. La substitution de l'uracile sur ce site produirait un codon d'arrêt et n'a donc pas été observée dans la nature, mais par souci de rigueur, nous avons inclus une condition de thymine dans notre évaluation. Nous avons conçu une sonde pour chacun des 4 nucléotides possibles à ce locus et établi 6 conditions thermodynamiques différentes basées sur l'énergie de liaison (ΔGo) des 3 régions de la sonde du pied (NHR, domaine et pied ; Fig. 3a, voir Matériels et méthodes). Ces conditions énergétiques vont des plus spécifiques (Sonde 1), dans lesquelles les énergies du NHR et du pied sont approximativement égales (environ -9 kcal/mol), aux plus sensibles (Sonde 6), dans lesquelles il n'y a pas de NHR et la prise du pied est légèrement plus forte (−12 kcal/mol). Dans tous les cas, l’énergie de liaison au domaine est ajustée pour maintenir l’énergie de liaison globale de la sonde constante à environ -40 kcal/mol.
a Paramètres de conception pour la caractérisation énergétique des sondes. Six schémas énergétiques différents ont été conçus en faisant varier les énergies de liaison des régions de pied, de domaine et de NHR, pour permettre un compromis entre la spécificité d'un seul nucléotide et la sensibilité sous forme de robustesse aux mutations. Quatre sondes différentes ont été conçues pour chacune des six conceptions énergétiques, couvrant les quatre codons possibles au niveau du site de mutation 484 dans la protéine de pointe du SRAS-CoV-2, dont trois ont été observées dans des variantes. Chaque sonde a été testée avec 12 matrices plasmidiques différentes, incluant les 4 mutations uniques mononucléotidiques seules et en présence d'une ou deux autres mutations trouvées plus en amont dans le variant Omicron. b Résultats caPCR de la série de sondes 484 K. Les courbes pleines rouges indiquent les performances sur la cible sans mutation. Les lignes pointillées, pointillées et tirets-points désignent les modèles avec 1, 2 ou 3 disparités par rapport à la séquence de sonde, respectivement. La couleur spécifie le codon au site 484 (E, K, Q ou STOP). c Schéma de test de la discrimination robuste du SRAS-CoV-1/2. Deux sondes ont été conçues selon le schéma énergétique "Sonde 5" en partie a pour une région du gène de la nucléocapside dans le SRAS-CoV-1 et le SRAS-CoV-2. Les deux sondes ont été testées en présence de la matrice de correspondance exacte et de variantes comprenant l'un ou les deux mésappariements en amont, ainsi qu'en présence de la matrice de correspondance exacte et de variantes mésappariées de l'autre sonde. d Résultats des tests décrits dans (c). Les lignes pleines, pointillées, pointillées et tirets indiquent des modèles de correspondance exacte, des modèles avec l'un des deux SNV ou des modèles avec les deux SNV par rapport au génome du SRAS-CoV-1 (bleu) ou du SRAS-CoV-2 (rouge).
Nous avons testé ces sondes avec 12 matrices d'ADN différentes, dont 4 représentent la matrice exacte pour chacune des 4 bases possibles dans les 484 codons. Pour démontrer l'accordabilité de l'énergétique de notre sonde, nous avons pris des mutations apparues dans les lignées ultérieures de l'évolution du SRAS-CoV-2 et les avons introduites dans les modèles : la mutation T478K trouvée dans les lignées Delta AY et Omicron, et la mutation S477N trouvée dans Omicron. lignées (figure schématique. 3a)10. Les 24 sondes ont été évaluées en réponse à l'amplification de chacune des 12 matrices. Chiffre 3b montre un sous-ensemble de ces résultats, spécifiquement pour les sondes conçues pour cibler la mutation 484 K trouvée dans les COV bêta et gamma10. À mesure que la force du NHR diminue et que la prise augmente, la détection du modèle de correspondance exacte (ligne rouge continue) reste forte et la sonde devient plus sensible aux modèles avec des disparités de 1 (lignes pointillées) et 2 (lignes pointillées). Cela démontre notre capacité à concevoir l’énergie des sondes pour obtenir des résultats de liaison à des cibles très spécifiques. Sur la base de ces résultats, nous avons sélectionné le schéma énergétique 2 pour les sondes discriminantes SNV et le schéma 5 pour les sondes robustes.
Nous avons conçu 2 sondes robustes ciblant la séquence génétique de la nucléocapside du SARS-CoV-1 et du SARS-CoV-2. Pour garantir que ces séquences pathogènes puissent être différenciées à l'aide de sondes robustes tout en restant tolérantes aux mutations, nous avons conçu 8 modèles ne contenant soit aucun mésappariement, soit 1 mésappariement sur 2, soit les deux mésappariements ensemble (Fig. 3c, voir Matériels et méthodes). La région sélectionnée pour la détection du SARS-CoV-1 et du SARS-CoV-2 diffère entre les 2 séquences cibles de 8 mutations : en tant que tels, les mésappariements dans la matrice ont été sélectionnés en fonction de ces différences. En tant que tel, le modèle SARS-CoV-2 avec 2 mésappariements ne présente que 6 différences par rapport à la séquence SARS-CoV-1, mettant ainsi en évidence une application réelle de l'importance d'un accordage précis des sondes pour la sensibilité par rapport à la spécificité. Chiffre 3d montre les résultats de ces tests. Dans tous les cas, nous n'avons observé aucune réactivité croisée entre les sondes et matrices SARS-CoV-1 et SARS-CoV-2 : l'ARN du SARS-CoV-1 n'a été détecté que par la sonde SARS-CoV-1, et le SARS-CoV-2 L’ARN n’a été détecté que par la sonde SARS-CoV-2. Les matrices contenant l'une ou l'autre des 2 mutations peuvent également être détectées sans ambiguïté par chaque sonde appropriée, l'une des 1 mutations affectant plus clairement les performances que l'autre. De même, les modèles contenant les deux mutations ensemble sont toujours détectés, bien que les performances soient légèrement compromises en raison d'une moindre faveur énergétique dans le déplacement du bras d'extinction.
Grâce à nos études ciblées sur l’énergétique des sondes, nous avons démontré notre capacité à concevoir avec précision des sondes pour obtenir le compromis souhaité entre sensibilité et spécificité. Les sondes discriminatoires SNV ont une énergie qui nécessite une correspondance exacte pour le déplacement du bras extincteur, tandis que les sondes robustes sont plus légères dans leur liaison aux cibles avec la capacité de détecter un amplicon contenant jusqu'à deux mutations. La plage énergétique que nous avons explorée peut être appliquée pour ajuster les sondes selon les besoins, du schéma énergétique 1 au schéma énergétique 6 afin de permettre la détection de cibles présentant le nombre souhaité de mésappariements par rapport à la séquence de sondes.
Performances des sondes robustes et discriminatoires SNV
Pour démontrer la preuve de concept de notre conception de sonde hautement réglable, nous avons créé un panel complet de sondes à la fois robustes et discriminatoires SNV. Notre panel met en évidence l'utilité de sondes robustes en dépistant les 7 coronavirus connus pour infecter les humains (HCoV 229E, HKU1, NL63 et OC43 ; MERS-CoV, SARS-CoV-1 et SARS-CoV-2) ainsi que la grippe. A et B8. De plus, notre panel démontre la fonctionnalité des sondes discriminatoires du SNV en utilisant 14 sondes et 3 amplicons couvrant 5 sites couramment mutés dans le gène de pointe du SRAS-CoV-2 pour permettre la différenciation entre les variantes préoccupantes (417, 452, 484, 501 et 614 ; fig. S2)9,10,11.
Pour évaluer les performances des sondes robustes de notre test RT-caPCR pour la détection de cibles sensibles, nous avons effectué 65 tests RT-caPCR en utilisant des échantillons de milieux de transport viral (VTM) négatifs pour le coronavirus couvrant une gamme de données démographiques de patients (Tableaux S1-S4) enrichi d'ARN synthétique provenant de chacun des 7 coronavirus humains, soit achetés auprès de Twist Bioscience, soit synthétisés en interne par transcription in vitro (IVT) à partir de fragments de gènes (Tableau S5). L'ARN a été isolé et purifié à partir de ces échantillons cliniques artificiels à l'aide d'une méthode d'extraction à base de billes13 en raison de la priorité accordée à l'utilisation d'une telle méthodologie pour l'extraction de l'ARN dans les flux de travail de diagnostic du SRAS-CoV-214 et a été utilisé comme entrée dans notre système. Ce test comprenait également à la fois un contrôle d'extraction (l'ARN du bactériophage MS2 ajouté à l'échantillon artificiel avant la purification) et un contrôle d'amplification (l'ARN du contrôle positif interne (IPC) ajouté au mélange réactionnel RT-caPCR). Sauf indication contraire, les expériences ici ont reçu environ 106 copies d'ARN MS2 et 107 copies d'ARN viral. La réaction RT-caPCR a été réalisée en 40 minutes en utilisant 5,000 11 molécules d’ARN de contrôle IPC, l’échantillon d’ARN extrait et XNUMX paires d’amorces couvrant toutes les cibles et tous les contrôles.
Les données RT-caPCR ont été analysées pour identifier les cibles virales présentes dans chaque échantillon et, dans le cas du SRAS-CoV-2, quelle variante spécifique était présente (Fig. 4). Figure 4a montre une analyse représentative avec la variante SARS-CoV-2 Omicron comme cible ARN. Toutes les sondes robustes pour la discrimination virale sont affichées en rouge tandis que les sondes discriminantes SNV sont affichées en bleu. Les contrôles d’extraction et d’amplification sont représentés en vert. Les spots de sonde positifs et négatifs correspondent à des sondes totalement non désactivées ou entièrement désactivées utilisant des séquences pour lesquelles aucune amorce ni matrice n'ont été incluses et sont affichées en gris. Ce type de données peut être résumé dans un graphique compressé comme le montre la Fig. 4b: toutes les courbes positives attendues sont représentées sur le tracé de gauche, et toutes les courbes négatives attendues sont représentées sur le tracé de droite. Bien qu'il existe un petit niveau d'activité hors cible sur certaines sondes spécifiques au SNV, la comparaison des courbes des sondes spécifiques au SNV sur la cible (484 A, 452 L, 501Y-Om, 417 N et 614 G) avec d'autres SNV- des sondes spécifiques confirment que les sondes ciblées répondent le plus fortement dans tous les cas, comme évalué à l'œil nu ainsi que par notre algorithme de discrimination SNV basé sur un dérivé de fluorescence (voir Matériels et méthodes et Méthodes supplémentaires). Nous notons qu'en raison de la charge mutationnelle élevée du variant Omicron par rapport aux autres variants11, nous avons conçu la séquence de la sonde 484 A pour inclure également les mutations S477N et T478K trouvées dans cette variante afin d'améliorer la détection10. De même, une sonde 501Y-Om unique comprend les mutations Q493R, G496S et Q498R qui se trouvent spécifiquement dans la variante Omicron. Une sonde spécifique de la sous-variante BA.2 de la lignée Omicron (501Y-Om2) a été introduite ultérieurement. Cette sonde est identique à la séquence 501Y-Om mais ne possède pas la mutation G496S10.
a L’exemple résulte d’une analyse RT-caPCR détectant la variante préoccupante du SARS-CoV-2 Omicron. Les sondes de détection virale robustes sont en rouge, les sondes de discrimination SNV sont en bleu, les contrôles d'extraction et d'amplification sont en vert et les contrôles positifs et négatifs sont en gris. Chaque courbe représente l'intensité du point correspondant du réseau de sondes à chaque instant sur les 60 images acquises, normalisée au point le plus brillant du réseau. b Résultats récapitulatifs correspondant au rodage a. Tous les positifs attendus (contrôles d'extraction et d'amplification, identification virale sur cible et toutes les sondes discriminantes du SNV sur la cible) sont présentés dans le graphique de gauche, tandis que toutes les sondes négatives attendues (toutes les autres sondes virales et discriminantes du SNV, comme ainsi que les contrôles négatifs) sont présentés dans le graphique de droite. c Résultats de détection virale pour le coronavirus humain 229E, le coronavirus du syndrome respiratoire du Moyen-Orient (MERS-CoV) et le SRAS-CoV-1 dans le format décrit dans (b). d. La discrimination SNV résulte d’un modèle de variante SARS-CoV-2 Delta dans le format spécifié ci-dessus.
Figure 4c montre les résultats représentatifs compressés de 3 analyses pour 3 modèles d'agents pathogènes viraux différents : coronavirus humain 229E, MERS-CoV et SARS-CoV-1. Les 3 échantillons montrent une détection claire de la cible d’ARN viral avec des sondes d’identification d’agents pathogènes robustes, aucune activité de sonde d’identification virale hors cible et la présence de tous les contrôles.
lampe de table 1 résume les résultats d’identification virale de tous les essais valides. La compétition pour les ressources de réaction PCR (par exemple, polymérase, dNTP) peut supprimer l'amplification des cibles à nombre de copies inférieur, de sorte qu'un test était considéré comme valide si un agent pathogène viral était détecté et qu'au moins 1 des 2 contrôles (MS2 et IPC) était détecté , ou aucun agent pathogène viral n'a été détecté mais les deux contrôles ont été détectés. 11 des échantillons étaient des contrôles négatifs, qui comprenaient uniquement des ARN de contrôle MS2 et IPC. Dans chacun de ces 11 échantillons, IPC et MS2 ont été détectés (100 %). 36 des 36 échantillons de SARS-CoV-2 ont été identifiés (100 %), comme évalué par détection de la sonde nucléocapside du SARS-CoV-2. Les autres coronavirus humains (229E, NL63, OC43 et HKU1) ainsi que les cibles du SRAS-CoV-1 et du MERS-CoV présentent également une sensibilité élevée, avec une détection de 100 % pour toutes les cibles. Le processus de liaison des amorces et le déplacement des sondes par l'extincteur médié par l'amplicon contribuent à la spécificité du panel, qui était de 100 % pour toutes les cibles. Ici, la spécificité est calculée comme la proportion de vrais négatifs correctement détectés. En bref, aucun modèle n’induit de détection au niveau d’une sonde robuste autre que celle pour laquelle il a été spécifiquement conçu.
Figure 4d montre une analyse représentative mettant en évidence la capacité de notre test RT-caPCR à identifier la cible de l'ARN et à fournir une résolution mononucléotidique en montrant la détection des variantes virales en même temps que l'identification des agents pathogènes pour la variante Delta du SRAS-CoV-2. Bien que la sonde 452 L montre une activité hors cible significative, sa détection est nettement moins efficace que la sonde 452 R sur cible (la courbe bleue avec la plus forte intensité dans le tracé de gauche). Ainsi, les sondes SNV-discriminantes permettent une distinction claire entre les variantes du SRAS-CoV-2 qui ne diffèrent que par quelques mutations uniques.
lampe de table 2 résume les performances des sondes spécifiques au SNV pour les variantes du SRAS-CoV-2 dans 36 essais. Le nombre de détections attendues est égal au nombre d'analyses pour lesquelles un modèle affichant cette mutation a été inclus, tandis que le nombre de négatifs attendus est le nombre d'analyses pour lesquelles le modèle correspondant n'a pas été inclus. Les vrais positifs sont des analyses pour lesquelles la sonde d'intérêt était la sonde dominante spécifique du SNV pour ce site de mutation (telle qu'évaluée algorithmiquement), et les vrais négatifs sont les analyses pour lesquelles ce n'était pas le cas. Lors d'une analyse, nous remarquons que la sonde 614 G a été obscurcie par une particule étrangère (poussière), le total pour ces cibles est donc de 35, au lieu de 36. Hormis la 484E, toutes les sondes affichent une sensibilité d'au moins 90 %. Les mauvaises performances de la sonde 484E peuvent s’expliquer en partie par la présence d’un SNV (mutation T478K) dans l’amplicon des variants Delta. Toutes les autres sondes présentent une spécificité de 100 %, car un amplicon déplacera toujours préférentiellement son extincteur correspondant par rapport à celui présentant une mutation, en supposant que les énergies sur la cible soient égales.
Ensemble, ces données mettent en évidence la capacité des sondes robustes et des sondes discriminatoires du SNV à travailler en tandem pour identifier rapidement la cible d'ARN présente (avec la sonde robuste) et fournir une résolution mononucléotidique (avec la sonde discriminatoire du SNV).
Évaluation de la limite de détection de la RT-caPCR
Nous avons ensuite réalisé une série d'expériences pour déterminer la limite de détection de notre test RT-caPCR. Dans tous les tests précédents de la Fig. 4, une entrée virale de 107 les molécules ont été introduites dans le VTM avant l'extraction, similaire aux estimations de la charge virale maximale pour les patients Omicron et Delta15,16. Ici, nous avons quantifié la LOD de notre test en faisant varier le nombre de copies d’entrée d’ARN viral et en gardant les mêmes copies de contrôle. En utilisant de l’ARN purifié obtenu auprès de Twist Bioscience ou synthétisé en interne à partir de fragments de gènes via IVT, nous avons atteint une LOD de 1,000 7 molécules pour les XNUMX coronavirus. Les résultats représentatifs sont présentés dans la Fig. S3. Dans les deux Fig. S3a ainsi que S3b, nous constatons que nous pouvons détecter avec succès jusqu'à 1,000 229 molécules par réaction pour une seule cible avec les deux contrôles (HCoV XNUMXE ; Fig. S3a) et pour une variante du SRAS-CoV-2 avec les deux contrôles (Omicron ; Fig. S3b), une quantité 3 ordres de grandeur inférieure à celle utilisée pour les tests VTM lors de l'utilisation d'ARN purifié. Lorsque seule la présence d’une seule cible varie, comme sur la Fig. S3a, nous trouvons un retard clair dans le temps de seuil pour chaque ordre de grandeur pour cette cible uniquement, alors que les 2 contrôles sont cohérents d'une exécution à l'autre. Cette tendance est moins nette pour le SARS-CoV-2, ce qui était prévisible étant donné que cette cible nécessite l’amplification simultanée de 4 amplicons et de 2 contrôles. La compétition pour les ressources enzymatiques et les dNTP semble perturber la tendance claire observée pour les cibles à amplicon unique. En figue. S3c, une série de dilutions a été réalisée en faisant varier la quantité d'ARN entrant dans le pipeline d'extraction pour la variante Omicron. Ici aussi, nous constatons une détection jusqu'à la valeur testée la plus basse (entrée de 10,000 1,000 molécules dans l'étape d'extraction de l'ARN avec environ 25 XNUMX molécules entrant dans l'étape de RT en raison de pertes lors de l'extraction et n'utilisant pas la totalité du volume d'élution de XNUMX µL dans la réaction) et un retard attendu dans l'amplification en fonction de la concentration d'entrée.
Détection de plusieurs cibles dans un seul échantillon
Après avoir démontré le succès de notre test RT-caPCR à sonde accordable pour identifier des cibles d'ARN avec des sondes robustes (chaque virus) avec une spécificité mononucléotidique avec des sondes discriminantes du SNV (variantes du SRAS-CoV-2), nous avons ensuite exploré comment notre test fonctionnait lorsque évaluer des entrées plus complexes en simulant des échantillons de co-infection contenant plusieurs cibles d’ARN. Ici, un montant d'entrée de 105 les molécules de chaque matrice ont été ajoutées directement dans la réaction RT-caPCR sans étapes d'extraction VTM. Nous avons également étendu notre test RT-caPCR pour tester davantage de cibles d'ARN en incluant 3 jeux d'amorces supplémentaires et 4 sondes pour tester les virus de la grippe A et B (dont 2 ciblant le gène de la protéine matricielle M1 de la grippe A).17 et 2 ciblant la protéine non structurale-1 (nsp1) dans les lignées Yamagata et Victoria de la grippe B18). Ce test 14-plex a été testé pour les 4 nouvelles cibles afin de confirmer son efficacité (Fig. S4).
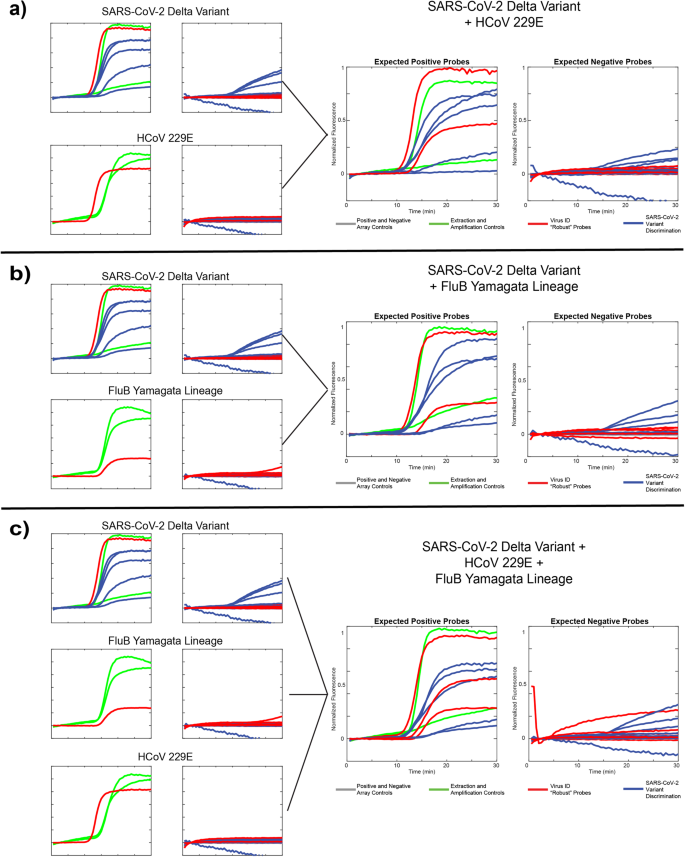
Nous avons évalué chacun des 3 modèles viraux différents seuls ainsi que dans 3 combinaisons différentes : variant Delta du SARS-CoV-2 + HCoV229E (Fig. 5a), variante Delta du SRAS-CoV-2 + lignée FluB Yamagata (Fig. 5b), et lignée SARS-CoV-2 + HCoV229E + FluB Yamagata (Fig. 5c). Les données sont présentées sous forme d'images de traces fluorescentes compressées comme décrit ci-dessus avec les sondes positives attendues dans le panneau de gauche et les sondes négatives attendues dans le panneau de droite. Dans toutes les analyses, nous avons observé le comportement attendu : des sondes robustes (identification du virus) signalent la présence de tous les modèles d'agents pathogènes inclus, et les sondes discriminantes SNV (identification des variantes) identifient correctement la variante Delta avec de faibles niveaux d'activité de sonde hors cible dans tous les cas. 5 sites SNV. La sonde 484E est la plus performante, probablement en raison de la disparité susmentionnée dans la région du domaine de cette sonde. De plus, dans certaines expériences, le contrôle d'extraction MS2 a donné de mauvais résultats en raison de la concurrence pour les ressources entre 8 amplicons différents. Nous notons également que ces analyses n'incluent qu'une seule répétition de chacun des contrôles MS1 et IPC, au lieu de 2 comme dans la Fig. 4, car il s'agissait d'un panneau mis à jour comprenant la sonde 501Y-Om2. Ces données démontrent la capacité de notre technologie à évaluer avec précision des échantillons plus complexes contenant plusieurs cibles d’ARN d’intérêt, démontrant ainsi la polyvalence du test.
a Résultats à modèle unique de la variante Delta du SRAS-CoV-2 et du coronavirus humain 229E, ainsi qu'un échantillon de « co-infection » comprenant les deux modèles. b Résultats à modèle unique de la variante Delta du SRAS-CoV-2 et de la lignée Yamagata de la grippe B, ainsi qu'un échantillon de co-infection comprenant les deux modèles. c. Résultats à cible unique provenant des mêmes cibles que dans a ainsi que b, ainsi qu'un échantillon de co-infection comprenant les trois modèles.
- Contenu propulsé par le référencement et distribution de relations publiques. Soyez amplifié aujourd'hui.
- PlatoData.Network Ai générative verticale. Autonomisez-vous. Accéder ici.
- PlatoAiStream. Intelligence Web3. Connaissance Amplifiée. Accéder ici.
- PlatonESG. Carbone, Technologie propre, Énergie, Environnement, Solaire, La gestion des déchets. Accéder ici.
- PlatoHealth. Veille biotechnologique et essais cliniques. Accéder ici.
- La source: https://www.nature.com/articles/s42003-023-05346-4