Dans cet article, nous démontrons comment affiner efficacement un modèle de langage protéique (pLM) de pointe pour prédire la localisation subcellulaire des protéines en utilisant Amazon Sage Maker.
Les protéines sont les machines moléculaires du corps, responsables de tout, du mouvement de vos muscles à la réponse aux infections. Malgré cette variété, toutes les protéines sont constituées de chaînes répétitives de molécules appelées acides aminés. Le génome humain code pour 20 acides aminés standards, chacun ayant une structure chimique légèrement différente. Celles-ci peuvent être représentées par des lettres de l’alphabet, ce qui nous permet ensuite d’analyser et d’explorer les protéines sous forme de chaîne de texte. L’énorme nombre possible de séquences et de structures protéiques est ce qui confère aux protéines leur grande variété d’utilisations.
Les protéines jouent également un rôle clé dans le développement de médicaments, en tant que cibles potentielles mais également thérapeutiques. Comme le montre le tableau suivant, bon nombre des médicaments les plus vendus en 2022 étaient soit des protéines (en particulier des anticorps), soit d’autres molécules comme l’ARNm traduites en protéines dans l’organisme. Pour cette raison, de nombreux chercheurs en sciences de la vie doivent répondre aux questions sur les protéines plus rapidement, à moindre coût et avec plus de précision.
| Nom | Fabricants | Ventes mondiales 2022 (en milliards de dollars) | Les indications |
| Commirnaty | Pfizer / BioNTech | $40.8 | COVID-19 |
| Spikevax | Moderne | $21.8 | COVID-19 |
| Humira | AbbVie | $21.6 | Arthrite, maladie de Crohn et autres |
| keytruda | Merck | $21.0 | Cancers divers |
Source des données : Urquhart, L. Principales entreprises et médicaments par ventes en 2022. Nature Reviews Drug Discovery 22, 260-260 (2023).
Parce que nous pouvons représenter les protéines sous forme de séquences de caractères, nous pouvons les analyser à l’aide de techniques développées à l’origine pour le langage écrit. Cela inclut des modèles de langage étendus (LLM) pré-entraînés sur d'énormes ensembles de données, qui peuvent ensuite être adaptés à des tâches spécifiques, comme la synthèse de texte ou les chatbots. De même, les pLM sont pré-entraînés sur de grandes bases de données de séquences protéiques à l’aide d’un apprentissage non étiqueté et auto-supervisé. Nous pouvons les adapter pour prédire des éléments tels que la structure 3D d’une protéine ou la manière dont elle peut interagir avec d’autres molécules. Les chercheurs ont même utilisé les pLM pour concevoir de nouvelles protéines à partir de zéro. Ces outils ne remplacent pas l’expertise scientifique humaine, mais ils ont le potentiel d’accélérer le développement préclinique et la conception des essais.
L’un des défis de ces modèles est leur taille. Les LLM et les PLM ont connu une croissance fulgurante au cours des dernières années, comme l'illustre la figure suivante. Cela signifie que cela peut prendre beaucoup de temps pour les entraîner avec une précision suffisante. Cela signifie également que vous devez utiliser du matériel, notamment des GPU, dotés de grandes quantités de mémoire pour stocker les paramètres du modèle.
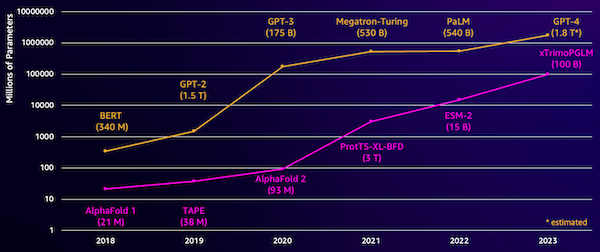
De longues durées de formation et des instances volumineuses équivaut à un coût élevé, ce qui peut rendre ce travail hors de portée pour de nombreux chercheurs. Par exemple, en 2023, un équipe de recherche a décrit la formation d'un pLM de 100 milliards de paramètres sur 768 GPU A100 pendant 164 jours ! Heureusement, dans de nombreux cas, nous pouvons économiser du temps et des ressources en adaptant un pLM existant à notre tâche spécifique. Cette technique est appelée réglage fin, et nous permet également d'emprunter des outils avancés à d'autres types de modélisation linguistique.
Vue d'ensemble de la solution
Le problème spécifique que nous abordons dans cet article est localisation subcellulaire: Étant donné une séquence protéique, peut-on construire un modèle capable de prédire si elle vit à l'extérieur (membrane cellulaire) ou à l'intérieur d'une cellule ? Il s’agit d’une information importante qui peut nous aider à comprendre la fonction et à savoir si elle constituerait une bonne cible médicamenteuse.
Nous commençons par télécharger un ensemble de données public en utilisant Amazon SageMakerStudio. Ensuite, nous utilisons SageMaker pour affiner le modèle de langage protéique ESM-2 à l'aide d'une méthode de formation efficace. Enfin, nous déployons le modèle comme point final d'inférence en temps réel et l'utilisons pour tester certaines protéines connues. Le diagramme suivant illustre ce flux de travail.

Dans les sections suivantes, nous passons en revue les étapes permettant de préparer vos données de formation, de créer un script de formation et d'exécuter une tâche de formation SageMaker. Tout le code présenté dans cet article est disponible sur GitHub.
Préparer les données d'entraînement
Nous utilisons une partie du Ensemble de données DeepLoc-2, qui contient plusieurs milliers de protéines SwissProt avec des emplacements déterminés expérimentalement. Nous filtrons des séquences de haute qualité comprises entre 100 et 512 acides aminés :
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(["Unnamed: 0", "Partition"], axis=1)
df["Membrane"] = df["Membrane"].astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df[df["Sequence"].apply(lambda x: len(x)).between(100, 512)]
# Remove unnecessary features
df = df[["Sequence", "Kingdom", "Membrane"]]
Ensuite, nous symbolisons les séquences et les divisons en ensembles de formation et d'évaluation :
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples["Sequence"]
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding["labels"] = examples["Membrane"]
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset["train"].column_names,
)
encoded_dataset.set_format("torch")
Enfin, nous téléchargeons les données de formation et d'évaluation traitées sur Service de stockage simple Amazon (Amazon S3) :
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset["train"].save_to_disk(train_s3_uri)
encoded_dataset["test"].save_to_disk(test_s3_uri)Créer un script de formation
Mode script SageMaker vous permet d'exécuter votre code de formation personnalisé dans des conteneurs de cadre d'apprentissage automatique (ML) optimisés gérés par AWS. Pour cet exemple, nous adaptons un script existant pour la classification de texte de Hugging Face. Cela nous permet d'essayer plusieurs méthodes pour améliorer l'efficacité de notre travail de formation.
Méthode 1 : Cours d’entraînement pondéré
Comme de nombreux ensembles de données biologiques, les données DeepLoc sont inégalement réparties, ce qui signifie qu’il n’y a pas un nombre égal de protéines membranaires et non membranaires. Nous pourrions rééchantillonner nos données et supprimer les enregistrements de la classe majoritaire. Cependant, cela réduirait le total des données d'entraînement et pourrait nuire à notre précision. Au lieu de cela, nous calculons les poids des classes pendant le travail de formation et les utilisons pour ajuster la perte.
Dans notre script de formation, nous sous-classons le Trainer classe de transformers avec une WeightedTrainer classe qui prend en compte les pondérations de classe lors du calcul de la perte d’entropie croisée. Cela permet d’éviter les biais dans notre modèle :
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMéthode 2 : accumulation de dégradés
L'accumulation de gradient est une technique de formation qui permet aux modèles de simuler la formation sur des lots de plus grande taille. En règle générale, la taille du lot (le nombre d'échantillons utilisés pour calculer le gradient en une étape d'entraînement) est limitée par la capacité de la mémoire du GPU. Avec l'accumulation de gradients, le modèle calcule d'abord les gradients sur des lots plus petits. Ensuite, au lieu de mettre à jour immédiatement les poids du modèle, les dégradés sont accumulés sur plusieurs petits lots. Lorsque les gradients accumulés sont égaux à la taille de lot cible plus grande, l'étape d'optimisation est effectuée pour mettre à jour le modèle. Cela permet aux modèles de s'entraîner avec des lots effectivement plus importants sans dépasser la limite de mémoire GPU.
Cependant, des calculs supplémentaires sont nécessaires pour les passes avant et arrière de lots plus petits. L'augmentation de la taille des lots via l'accumulation de gradient peut ralentir l'entraînement, surtout si trop d'étapes d'accumulation sont utilisées. L'objectif est de maximiser l'utilisation du GPU tout en évitant les ralentissements excessifs dus à un trop grand nombre d'étapes de calcul de gradient supplémentaires.
Méthode 3 : points de contrôle du dégradé
Le point de contrôle de gradient est une technique qui réduit la mémoire nécessaire pendant l'entraînement tout en gardant un temps de calcul raisonnable. Les grands réseaux de neurones occupent beaucoup de mémoire car ils doivent stocker toutes les valeurs intermédiaires du passage aller afin de calculer les gradients lors du passage arrière. Cela peut entraîner des problèmes de mémoire. Une solution est de ne pas stocker ces valeurs intermédiaires, mais il faut alors les recalculer lors du retour en arrière, ce qui prend beaucoup de temps.
Les points de contrôle de dégradé offrent une approche équilibrée. Il enregistre seulement certaines des valeurs intermédiaires, appelées les points de contrôle, et recalcule les autres si nécessaire. Par conséquent, cela utilise moins de mémoire que de tout stocker, mais aussi moins de calculs que de tout recalculer. En sélectionnant stratégiquement les activations à contrôler, le contrôle par gradient permet de former de grands réseaux de neurones avec une utilisation de la mémoire et un temps de calcul gérables. Cette technique importante permet de former des modèles très volumineux qui autrement se heurteraient à des limitations de mémoire.
Dans notre script de formation, nous activons l'activation du gradient et les points de contrôle en ajoutant les paramètres nécessaires au TrainingArguments objet:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Méthode 4 : Adaptation de bas rang des LLM
Les grands modèles de langage comme ESM-2 peuvent contenir des milliards de paramètres coûteux à former et à exécuter. Les chercheurs a développé une méthode de formation appelée Low-Rank Adaptation (LoRA) pour rendre plus efficace le réglage précis de ces énormes modèles.
L'idée clé derrière LoRA est que lors du réglage fin d'un modèle pour une tâche spécifique, vous n'avez pas besoin de mettre à jour tous les paramètres d'origine. Au lieu de cela, LoRA ajoute de nouvelles matrices plus petites au modèle qui transforment les entrées et les sorties. Seules ces matrices plus petites sont mises à jour lors du réglage fin, ce qui est beaucoup plus rapide et utilise moins de mémoire. Les paramètres du modèle d'origine restent figés.
Après un réglage fin avec LoRA, vous pouvez fusionner les petites matrices adaptées dans le modèle d'origine. Ou vous pouvez les garder séparés si vous souhaitez affiner rapidement le modèle pour d'autres tâches sans oublier les précédentes. Dans l’ensemble, LoRA permet aux LLM d’être adaptés efficacement à de nouvelles tâches pour une fraction du coût habituel.
Dans notre script de formation, nous configurons LoRA en utilisant le PEFT bibliothèque de Hugging Face :
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=[
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
]
)
model = get_peft_model(model, peft_config)Soumettre une tâche de formation SageMaker
Après avoir défini votre script de formation, vous pouvez configurer et soumettre une tâche de formation SageMaker. Tout d’abord, spécifiez les hyperparamètres :
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Ensuite, définissez les métriques à capturer à partir des journaux de formation :
metric_definitions = [
{"Name": "epoch", "Regex": "'epoch': ([0-9.]*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ([0-9.e-]*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ([0-9.e-]*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ([0-9.e-]*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ([0-9.e-]*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ([0-9.e-]*)"},
]Enfin, définissez un estimateur Hugging Face et soumettez-le pour formation sur un type d'instance ml.g5.2xlarge. Il s'agit d'un type d'instance rentable qui est largement disponible dans de nombreuses régions AWS :
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=[{"Key": "project", "Value": "esm-fine-tuning"}],
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)Le tableau suivant compare les différentes méthodes de formation dont nous avons discuté et leur effet sur le temps d'exécution, la précision et les besoins en mémoire GPU de notre travail.
| configuration | Temps facturable (min) | Précision de l'évaluation | Utilisation maximale de la mémoire GPU (Go) |
| Modèle de base | 28 | 0.91 | 22.6 |
| Base + AG | 21 | 0.90 | 17.8 |
| Base + CPG | 29 | 0.91 | 10.2 |
| Base + LoRA | 23 | 0.90 | 18.6 |
Toutes les méthodes ont produit des modèles avec une grande précision d’évaluation. L'utilisation de LoRA et de l'activation par gradient a réduit le temps d'exécution (et le coût) de 18 % et 25 %, respectivement. L'utilisation des points de contrôle de gradient a réduit l'utilisation maximale de la mémoire GPU de 55 %. En fonction de vos contraintes (coût, délai, matériel), l'une de ces approches peut être plus judicieuse qu'une autre.
Chacune de ces méthodes fonctionne bien seule, mais que se passe-t-il lorsque nous les utilisons en combinaison ? Le tableau suivant résume les résultats.
| configuration | Temps facturable (min) | Précision de l'évaluation | Utilisation maximale de la mémoire GPU (Go) |
| Toutes les méthodes | 12 | 0.80 | 3.3 |
Dans ce cas, nous constatons une réduction de précision de 12 %. Cependant, nous avons réduit le temps d'exécution de 57 % et l'utilisation de la mémoire GPU de 85 % ! Il s'agit d'une diminution massive qui nous permet de nous entraîner sur un large éventail de types d'instances rentables.
Nettoyer
Si vous suivez votre propre compte AWS, supprimez tous les points de terminaison et données d'inférence en temps réel que vous avez créés pour éviter des frais supplémentaires.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Conclusion
Dans cet article, nous avons démontré comment affiner efficacement les modèles de langage protéique comme ESM-2 pour une tâche scientifiquement pertinente. Pour plus d'informations sur l'utilisation des bibliothèques Transformers et PEFT pour former pLMS, consultez les articles Apprentissage profond avec des protéines ainsi que ESMBind (ESMB) : adaptation de bas rang d'ESM-2 pour la prédiction du site de liaison aux protéines sur le blog Hugging Face. Vous pouvez également trouver d’autres exemples d’utilisation de l’apprentissage automatique pour prédire les propriétés des protéines dans le Analyse de protéines impressionnante sur AWS Référentiel GitHub.
À propos de l’auteur
 Brian Loyal est architecte principal de solutions IA/ML au sein de l'équipe mondiale des soins de santé et des sciences de la vie chez Amazon Web Services. Il a plus de 17 ans d'expérience en biotechnologie et en apprentissage automatique, et se passionne pour aider les clients à résoudre les défis génomiques et protéomiques. Dans ses temps libres, il aime cuisiner et manger avec ses amis et sa famille.
Brian Loyal est architecte principal de solutions IA/ML au sein de l'équipe mondiale des soins de santé et des sciences de la vie chez Amazon Web Services. Il a plus de 17 ans d'expérience en biotechnologie et en apprentissage automatique, et se passionne pour aider les clients à résoudre les défis génomiques et protéomiques. Dans ses temps libres, il aime cuisiner et manger avec ses amis et sa famille.
- Contenu propulsé par le référencement et distribution de relations publiques. Soyez amplifié aujourd'hui.
- PlatoData.Network Ai générative verticale. Autonomisez-vous. Accéder ici.
- PlatoAiStream. Intelligence Web3. Connaissance Amplifiée. Accéder ici.
- PlatonESG. Carbone, Technologie propre, Énergie, Environnement, Solaire, La gestion des déchets. Accéder ici.
- PlatoHealth. Veille biotechnologique et essais cliniques. Accéder ici.
- La source: https://aws.amazon.com/blogs/machine-learning/efficiently-fine-tune-the-esm-2-protein-language-model-with-amazon-sagemaker/



